-
石油是由烷烃、多环芳烃、以及少量沥青和树脂组成的复杂混合物。在石油开采、炼制等生产过程中产生的废水中含有一定数量的原油,这些石油类污染物质以浮油、分散油、乳化油和溶解油等形式存在于水体中[1],如果不经处理直接排放,会对地表水及土壤等环境介质造成污染。目前对于含油废水的处理主要包括浮选、生物降解、膜分离、高级氧化等技术[2]。其中,在微生物降解基础上发展起来的生物处理法具有操作简单,成本低,不易产生二次污染等优点,是一种环境友好型的处理技术[3-4]。
生物处理法是利用微生物的新陈代谢分解废水中有机物和某些无机毒物(如氰化物、硫化物),并将其转化为稳定无害物质的一种污染处理方法[5]。在利用生物法处理含油废水时,能否筛选出对毒性物质耐受性强且具有降解作用的功能菌是方法成功与否的关键。一些研究工作对于接种降解菌强化处理含油废水进行了报道[6-8]。由于石油烃的复杂性和降解异质性,多数情况下,靠单一的微生物菌种很难实现其完全降解。将不同种属的降解菌进行组合,构建结构稳定、功能广泛的高效微生物菌群,可以弥补接种单株菌强化方法上的不足,有望成为生物法净化含油废水的有效途径。然而,在降解机制探究方面,很难阐明复杂菌群中各种菌株的详细功能作用和细微结构[9-12]。
石油烃降解功能基因是指微生物产生的对于石油烃具有特定降解作用的酶编码基因,通过对功能基因进行测定,可用于分析微生物对石油烃的生物降解潜势[13-15]。在好氧条件下,烷烃单加氧酶基因alkB2和多环芳香烃末端双加氧酶基因Ahd是微生物代谢石油不同组分的主要酶系控制基因[16]。检测石油烃降解功能关键基因是评价石油烃生物降解潜势的重要依据。
本文以采自陕北地区的陈旧性石油污染土壤作为石油烃降解菌源,通过富集培养获得石油烃降解菌群LW-10,通过向高浓度含油废水中接种降解菌群LW-10进行强化处理,探究了石油烃不同组分的生物去除特性、降解菌在含油废水中的生长情况以及石油烃降解关键功能基因的变化规律。研究可为探明水体中石油烃的生物去除特性提供理论基础,同时可为接种降解菌群强化去除水体中原油提供技术支撑。
-
石油烃降解菌菌源为采自陕北志丹县(北纬36°47'44.97",东经108°28'24.65")油井场区受石油污染的黄绵土,土壤中的含油量为26633 mg·kg−1。土壤的理化性质测定方法参考《土壤农化分析》[17]。测定结果见表1[18]。
-
以上述土壤作为石油烃降解菌菌源,以灭菌的L9作为富集培养基,采用富集培养法筛选石油烃降解菌群。将获得的降解菌群溶于PBS缓冲液中,并调节菌悬液的光密度值为OD600=1.0备用,石油烃降解菌群悬液记为LW-10(Accession number: SRR15082184)。详细的富集培养方法见文献[18]。
将获得的LW-10菌群送往上海生工生物工程股份有限公司(www.sangon.com),采用Illumina MiSeq进行高通量测序 。测序所得结果利用 QIIME、Mothur进行序列过滤和嵌合体去除。利用Usearch软件依据97%的相似度进行OTU(Operational taxonomic unit)分类,在python中利用ete3 package绘制LW-10菌群组成的树状图。
-
向6个50 mL锥形瓶中分别加入20 mL L9培养基和0.1 g原油(取自长庆油田采油六厂),向其中3瓶中分别接种1 mL OD600 =1.0(菌浓度为 1×108 cfu·mL−1)的LW-10菌悬液,另外3瓶不进行接种作为控制实验(KB)。放入恒温水浴振荡器中在30 ℃、150 r·min−1条件下进行17 d的降解实验。
-
总石油烃的测定 降解17 d时,将1.3节中的降解体系转移至50 mL离心管中,以石油醚作为萃取剂,利用超声波破碎仪(JY96-IIN,宁波新芝科技股份有限公司)进行提取,利用紫外光度法测定总石油烃含量[19],测定波长为225 nm。在进行标准曲线的绘制时,标准油为取自长庆油田采油六厂的原油,属于石蜡基轻质原油,其基本理化性质为:密度(0.83±0.02)g·cm−1;蜡(15.46±1.23)%;胶质(6.03±0.85)%;沥青质(2.2±0.2)%。
烷烃(C7—C40)和16 PAHs的测定 利用氧化铝硅胶层析柱[20]从总石油烃中分离提取烷烃(Alks)和多环芳烃(PAHs)。根据美国国家环保局EPA8270C[21,22]方法利用气相色谱-质谱联用仪(安捷伦7890B 5977C气-质联用仪)测定上述分离中的烷烃和16种PAHs的含量。16种PAHs 标准品(Accustandard,美国);烷烃标准品(Sigma,美国)。
-
在降解进行至17 d时,在8000 r·min−1条件下离心收集降解体系中的菌体,用L9培养基配制成OD600=1.0的菌悬液后稀释10000 倍,取450 μL于1.5 mL离心管中,加入10 μL SYBR Green I/PI染液。样品经过涡轮混匀振荡15 s,于35 ℃恒温金属浴中避光孵育20 min,利用流式细胞仪Accuri C6测定降解菌的数量和活性。
流式细胞检测参数 利用CyFlow-SL装置进行检测。绿色荧光信号采集于FL1=500 nm通道。设定阈值为600收集荧光强度,计数率保持在500 cell·s−1以下,以30 mL·min−1的中速进行细胞计数[23]。
-
DNA的提取与测定 利用细菌基因组DNA提取试剂盒(天根生化科技(北京)有限公司,DP302)提取降解体系中的DNA,利用微量分光光度计(Nanodrop 2000,美国Thermo Fisher Scientific公司)检测DNA浓度与纯度。
荧光定量PCR测定 根据文献报道的Ahd、PA4513、alkB2、基因定性检测引物(表2),以提取的DNA为模板,在Mastercycler ep gradient PCR仪(德国,Eppendorf)上对降解基因进行PCR扩增。以此为靶序列设计高效特异性扩增qPCR引物,经由上海生工生物工程股份有限公司进行引物合成。
提取鉴定后重组质粒的DNA,进行10倍梯度稀释获得梯度浓度标准品,以此为模板利用荧光定量PCR仪(Bio-Rad iQ5,美国伯乐)构建测定降解基因的标准曲线(表3所示)。以提取的降解体系中的DNA为模板,利用相同的扩增程序进行起始拷贝数的定量测定。扩增体系为:总体积为20 μL,包括10 μL的FastStart Essential DNA Green Master(美国,Roche公司),上、下游引物各0.4 μL(10 μmol·L−1),3.2 μL ddH2O,6.0 μL DNA模板,另设置无DNA模板的阴性对照。扩增程序:95℃预变性10 min,95℃变性10 s,退火温度保持15 s,72 ℃延伸10 s,共设40个循环。熔解曲线程序:以0.5 ℃·s−1由55 ℃升至95 ℃,每个温度下保持5 s。
-
以原油为唯一碳源和能源,以L9为富集筛选培养基,利用富集培养法从石油污染土壤中筛选出的石油烃降解菌群LW-10(Accession number: SRR15082184)组成如图1所示。
降解菌群在门水平上的主要组成为:变形菌门( Proteobacteria,54. 65% )、拟杆菌门(Bacteroidetes,34.42%)和放线菌门(Actinobacteria,10.83%);在属水平上组成为:不动菌属(Acinetobacter,25.06%)、假单胞菌属(Pseudomonas,2.25%)、伯克氏菌属(Pandoraea,14.3%)、无色杆菌属(Achromobacter,8.57%)、橄榄菌属(Olivibacter ,33.42%)、红球菌属(Rhodococcus,6.02%)、白杆菌属(Leucobacter,2.86%)。
-
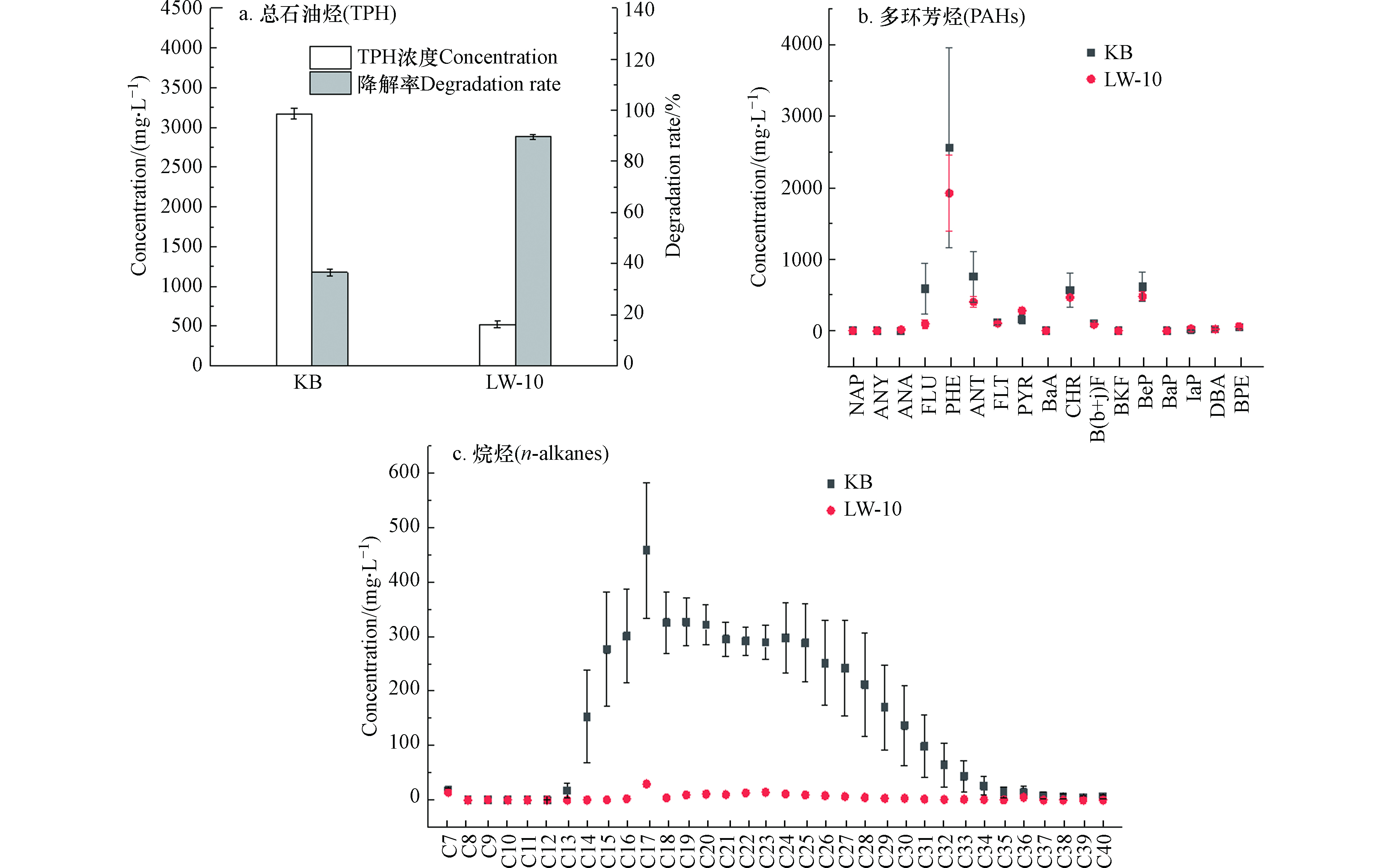
向高浓度含油废水(5000 mg·L−1)中接种LW-10菌群进行17 d的降解实验,对水相中原油的去除率达到89.6%。不进行接种的控制实验中石油烃降解率为36.6%(图2a)。说明筛选获得的石油烃降解菌群对水相中原油具有高效降解能力。未接种LW-10的控制体系中有36.6%的石油被去除,可能是由于轻质油的挥发和光降解作用所致。
利用柱层析-GC/MS测定体系中的C7-C40正烷烃和16种多环芳烃[26]。经过17 d的降解,接种降解菌群LW-10的体系中,C7-C40正烷烃和16 PAHs的总量分别为164.0 mg·L−1和3969.0 µg·L−1,而控制实验(KB)中C7-C40正烷烃和16PAHs的总量分别为4958.6 mg·L−1和5543.2 µg·L−1。接种降解菌的体系对正烷烃和16 PAHs的去除率分别达到96.7 %和28.4 %(图2b;图2c),说明降解菌群LW-10对原油中的正烷烃组分具有高效降解作用,对16种PAHs去除效果较差。
石油烃组分中,由于多环芳烃的稳定环状结构[27],使得其生物可降解性低于烷烃组分。本文中筛选获得的降解菌群LW-10对原油中的正烷烃组分降解效果明显高于多环芳烃。在后续构建多功能混合菌群时,应分别以烷烃和多环芳烃为碳源筛选获得对不同组分烃具有特异降解能力的菌株,并将不同类型菌株按一定比例混合后降解原油,通过测定石油不同组分的去除率以获得混合菌群的最佳配比。
-
本文利用流式细胞术测定体系中细菌总数和活细菌数量[],结果如图3a和3b所示。体系中降解菌群LW-10的初始接种量为1.0×108 cfu·mL−1,经过17 d的培养,降解菌总量增加至2.1×109 cfu·mL−1,其中高活性菌数量为8.3×108 cfu·mL−1。细菌总量约是初始接种量的21倍,高活性菌数量是初始接种量的8.3倍,均明显高于初始接种菌的数量,说明接种的降解菌在降解石油烃的过程中发生同化增殖作用。体系中的总菌数多于活性菌数,有可能是由于LW-10在降解石油烃的过程中发生了细胞凋亡[28]。
-
分别采用 Ahd、PA4513、alkB2基因的特异引物对降解体系中石油烃降解功能基因进行实时定量 PCR扩增,根据相应标准曲线计算得到Ahd、PA4513、alkB2基因拷贝数,得到降解体系中3种基因定量检测结果(表4)。降解过程中,体系中控制烷烃降解的单加氧酶基因(alkB2)和氧化还原酶基因(PA4513)拷贝数分别由1.06×108、4.09×106 copy·mL−1增加至2.84×108 、9.26×108 copy·mL−1,然而,控制多环芳烃组分降解的芳烃双加氧酶基因(Ahd)由1.06×108 copy·mL−1降低至1.65×107 copy·mL−1。体系中烷烃单加氧酶基因拷贝数多于芳烃双加氧酶的基因拷贝数。在降解过程中,烷烃单加氧酶基因(alkB2)拷贝数增加,芳烃双加氧酶基因(Ahd)基因拷贝数显著减少。结合烷烃和多环芳烃降解效果(图2b;图2c),说明石油烃降解菌群的基因表达情况可以指示其对不同组分烃的降解能力,降解过程中基因拷贝数的变化与菌群对不同组分烃的降解能力有关。
-
从石油污染土壤中筛选出的由多种门、属组成的复合型石油烃降解菌群LW-10可以降解水相中高浓度原油,对烷烃组分具有高效降解能力,对多环芳烃的降解能力较差。LW-10菌群代谢石油烃的过程中发生细菌增殖。降解过程中基因拷贝数的变化与不同组分烃的降解效果存在一致性,石油烃降解功能基因的表达情况可以指示其对不同组分烃的降解能力。本研究结果表明不同组分烃降解功能基因的定量检测可为生物强化去除不同石油烃的菌群构建和修复效果评价提供科学依据。
高浓度含油废水中不同组分烃的生物强化去除特性
Removal efficiencies of different components of crude oil by bioaugmentation
-
摘要: 为探究石油烃降解菌群对高浓度含油废水中不同组分烃的生物降解特性,向含油水相中接种石油烃降解菌群LW-10(Accession number: SRR15082184)进行降解实验。利用GC-MS研究了LW-10对原油中不同组分烃的降解性能,采用流式细胞术检测降解体系中的菌量变化。利用qPCR技术对控制不同组分烃降解的关键基因进行检测。结果表明,原油浓度为5000 mg·L−1的含油废水中接种LW-10降解17 d,对原油中烷烃和多环芳烃组分的降解率分别为96.7%和28.4%。体系中的降解菌总浓度与高活性菌浓度由接种时的1.0×108 cfu·mL−1增加至2.1×109 cfu·mL−1和8.3×108 cfu·mL−1。检测的3种石油烃降解功能基因中,烷烃单加氧酶基因alkB2拷贝数由1.06×108 copy·mL−1变为2.84×108 copy·mL−1;芳烃双加氧酶基因Ahd由1.06×108 copy·mL−1降低至1.65×107 copy·mL−1;氧化还原酶基因PA4513由4.09×106 copy·mL−1增加增加至9.26×108 copy·mL−1。结果表明降解菌群LW-10对含油废水中的烷烃组分具有高效降解能力,对多环芳烃组分的降解能力较差。LW-10在降解石油烃过程中通过同化作用发生增殖,不同组分烃的降解效果与石油烃降解功能基因之间存在正相关关系。Abstract: The aim of this study is to investigate the biodegradation efficiencies of different components of crude oil and functional gene shift by inoculating petroleum degrading flora LW-10 (Accession number: SRR15082184) in the oily aqueous phase. The bacterial numbers in the degradation systems were detected by flow cytometry. The copy numbers of functional genes were determined by using real-time polymerase chain reaction (qPCR). Results showed that petroleum-degrading flora LW-10 isolated from the petroleum-contaminated soil was composed by Acinetobacter, Pseudomonas, Pandoraea, Achromobacter, Olivibacter, Rhodococcus, and Leucobacter. After 17 d of inoculation, the numbers of the total bacteria and highly active bacteria in the systems increased from 1.0×108 cfu·mL−1 to 2.1×109 cfu·mL−1 and 8.3×108 cfu·mL−1, respectively. The degradation rates of alkanes and polycyclic aromatic hydrocarbons (PAHs) were 96.7% and 28.4%, respectively. For hydrocarbon degradation functional genes detected, the copy numbers of alkane monooxygenase genes AlkB2 changed from 1.06×108 copy·mL−1 to 2.84×108 copy·mL−1; aromatics dioxygenase genes Ahd decreased from 1.06×108 copy·mL−1 to 1.65×107 copy·mL−1; the oxidoreductase gene PA4513 increased from 4.09×106 copy·mL−1 to 9.26×108 copy·mL−1. Results suggested that flora LW-10 had high degradation capacity towards alkane components in the aqueous phase, but its degradation ability towards PAHs is poor. Also, the flora LW-10 proliferated in the process of degrading petroleum hydrocarbons. There is a positive correlation between the degradation effects of different components and the function genes that encode the enzymes controlling hydrocarbon biodegradation.
-
-
图 3 降解体系中细菌数量变化
Figure 3. The numbers of petroleum-degrading microorganisms in the degradation systems (3-a: Flow cytometry scatter diagram, P5 is the number of total cells in the systems, and P1 is the number of highly active cells in the systems; 3-b: the total number of bacteria and the highly active bacteria in the systems which were calculated according to the flow cytometers diagram)
表 1 土壤基本理化性质[18]
Table 1. Basic physical and chemical properties of the soil
指标
Index含量
Content测定方法
Assay method含水率/% 4.5±0.3 烘干称重法 pH 7.69±0.05 pH电极法 氧化还原电位/mV 137±5 电位法 氨氮/(mg·kg−1) 13.02±0.41 靛酚蓝 硝氮/(mg·kg−1) 0.45±0.03 紫外分光光度法 全氮/(g·kg−1) 0.61±0.10 元素分析仪 石油烃/(mg·kg−1) 26633±325 重量法 表 2 定性PCR扩增引物
Table 2. Qualitative PCR primers
目的基因
Target genes引物序列(5’- 3’)
Primer sequence
(5’-3’)扩增尺寸/bp
Amplicon size退火温度/℃
Annealing temperature参考文献
Reference芳烃双加氧酶基因Ahd F: AACATTGGCACTGATTACGAT 340 54 [24] R: GTAAACTACCGTCAAGTGCAT 氧化还原酶基因PA4513 F: GTGAAGAAAGTCTGGTTCCAGT 58 502 [25] R: GCCAGTCGAAAACCTTGC 烷烃单加氧酶基因alkB2 F: GCGATCTTCTTCCTCGGCCAGT 60 334 [25] R: CGCGCACCTTCGGGTCCATC 表 3 各目的基因标准曲线公式
Table 3. Standard curve formula of h target genes
目的基因
Target genes标准曲线公式
Standard curve formulaR2 E/% 芳烃双加氧酶基因Ahd Ct=−3.462 lgX0+39.395 0.997 94.5 氧化还原酶基因PA4513 Ct=−3.838 lgX0+43.436 0.999 82.2 烷烃单加氧酶基因alkB2 Ct=−3.247 lgX0+35.771 0.996 103.2 表 4 石油烃降解基因变化
Table 4. The changes of petroleum hydrocarbon degradation genes
基因名称
Gene name0 d拷贝数/(copy·mL−1)
0 d Copy number17 d拷贝数/(copy·mL−1)
17 d Copy numberAhd 1.06×108 1.65×107 PA4513 4.09×106 9.26×108 alkB2 1.06×108 2.84×108 -
[1] 胡天佑, 唐瑾, 陈志莉, 等. 石油工业含油废水处理进展 [J]. 水处理技术, 2021, 47(6): 12-17. doi: 10.16796/j.cnki.1000-3770.2021.06.003 HU T Y, TANG J, CHEN Z L, et al. Progress in oily wastewater treatment in the petroleum industry [J]. Water Treatment Technology, 2021, 47(6): 12-17(in Chinese). doi: 10.16796/j.cnki.1000-3770.2021.06.003
[2] JAMALY S, GIWA A, HASAN S W. Recent improvements in oily wastewater treatment: Progress, challenges, and future opportunities [J]. Journal of Environmental Sciences (China), 2015, 37: 15-30. doi: 10.1016/j.jes.2015.04.011 [3] 吴亮, 梁生康, 王修林, 等. 两种缓释肥料在海水中的养分释放特性及强化石油烃生物降解 [J]. 环境化学, 2010, 29(3): 455-461. WU L, LIANG S K, WANG X L, et al. Study on nutrient release characteristics of two slow-release fertilizers in seawater and their abilities to enhance biodegradation of petroleum hydrocarbons [J]. Environmental Chemistry, 2010, 29(3): 455-461(in Chinese).
[4] 李沅蔚, 王传远, 邹艳梅, 等. 渤海表面沉积物中石油-重金属复合污染的生物修复 [J]. 环境化学, 2019, 38(1): 186-194. doi: 10.7524/j.issn.0254-6108.2018012902 LI Y W, WANG C Y, ZOU Y M, et al. Bioremediation of combined pollution of petroleum and heavy metal in the surface sediments of Bohai Sea [J]. Environmental Chemistry, 2019, 38(1): 186-194(in Chinese). doi: 10.7524/j.issn.0254-6108.2018012902
[5] PENDASHTECH A R, ABDULLAH L C, FAKHRU L A, et al. Evaluation of membrane bioreactor for hyper saline oily wastewater treatment [J]. Process Safety & Environmental Protection, 2012, 90(1): 45-55. [6] MARIA S. K, IRENA B. I, MARINA K, et al. Oilfield wastewater biotreatment in a fluidized-bed bioreactor using co-immobilized Rhodococcus cultures [J]. Journal of Environmental Chemical Engineering, 2017, 5(1): 1252-1260. doi: 10.1016/j.jece.2017.01.043 [7] KOUTINAS M, KYRIAKOU M, ANDREOU K, et al. Enhanced biodegradation and valorization of drilling wastewater via simultaneous production of biosurfactants and polyhydroxyalkanoates by Pseudomonas citronellolis SJTE-3 [J]. Bioresource Technology, 2021, 340: 125679. doi: 10.1016/j.biortech.2021.125679 [8] NIE M Q, NIE H Y, HE M L, et al. Immobilization of biofilms of Pseudomonas aeruginosa NY3 and their application in the removal of hydrocarbons from highly concentrated oil-containing wastewater on the laboratory scale [J]. Journal of Environmental Management, 2016, 173: 34-40. [9] ZAINAB Z. I, HANEEN A. K. Biotreatment of real petroleum wastewater using non-acclimated immobilized mixed cells in spouted bed bioreactor [J]. Biochemical Engineering Journal, 2018, 131: 17-23. doi: 10.1016/j.bej.2017.12.005 [10] BANDAR A A, ARULAZHAGA P, MAMDOH T. J, et al. Application of integrated extremophilic (halo-alkalo-thermophilic) bacterial consortium in the degradation of petroleum hydrocarbons and treatment of petroleum refinery wastewater under extreme condition [J]. Journal of Hazardous Materials, 2021, 413: 125351. doi: 10.1016/j.jhazmat.2021.125351 [11] KE X, SUN J C, LIU C, et al. Fed-in-situ biological reduction treatment of food waste via high-temperature-resistant oil degrading microbial consortium [J]. Bioresource Technology, 2021, 340: 125635. doi: 10.1016/j.biortech.2021.125635 [12] KE X, HUA X, SUN J C, et al. Synergetic degradation of waste oil by constructed bacterial consortium for rapid in situ reduction of kitchen waste [J]. Journal of Bioscience and Bioengineering, 2021, 131(4): 412-419. doi: 10.1016/j.jbiosc.2020.12.005 [13] 于寒颖, 杨慧. 石油烃降解酶及其基因的结构、功能和表达调控 [J]. 应用与环境生物学报, 2012, 18(6): 1066-1074. YU H Y, YANG H. Regulation of the structure, function, and expression of petroleum hydrocarbon degrading enzymes and their genes [J]. Application and Environmental Biology, 2012, 18(6): 1066-1074(in Chinese).
[14] 吴慧君, 宋权威, 郑瑾, 等. 微生物降解石油烃的功能基因研究进展 [J]. 微生物学通报, 2020, 47(10): 3355-3368. doi: 10.13344/j.microbiol.china.200402 WU H J, SONG Q W, ZHENG J, et al. Function genes in microorganisms capable of degrading petroleum hydrocarbon [J]. Microbiology China, 2020, 47(10): 3355-3368(in Chinese). doi: 10.13344/j.microbiol.china.200402
[15] 吴彬彬, 卢滇楠, 刘铮. 石油污染土壤生物修复过程中氮循环功能基因的动态检测 [J]. 环境科学, 2012, 33(6): 2068-2074. doi: 10.13227/j.hjkx.2012.06.045 WU B B, LU D N, LIU Z. Dynamic detection of nitrogen cycle functional genes during bioremediation in petroleum-contaminated soil [J]. Environmental Science, 2012, 33(6): 2068-2074(in Chinese). doi: 10.13227/j.hjkx.2012.06.045
[16] 刘庆龙, 唐景春, 万晓彤. 污染土壤中主要石油降解基因AlkB和Nah定量检测方法的建立和应用 [J]. 分析化学, 2014, 42(9): 1348-1353. doi: 10.11895/j.issn.0253-3820.140103 LIU Q L, TANG J C, WAN X T. Establishment of analysis method for detection of petroleum degrading genes AlkB and nah in contaminated soil and its application [J]. Analytical Chemistry, 2014, 42(9): 1348-1353(in Chinese). doi: 10.11895/j.issn.0253-3820.140103
[17] 鲍士旦. 土壤农化分析[M]. 北京: 中国农业出版社, 2000. BAO S D. Soil agrochemical analysis [M]. Beijing: China Agricultural Publishing House, 2000 (in Chinese).
[18] 许殷瑞, 吴蔓莉, 王丽, 等. 陕北油田区石油污染土壤微生物种群变化及影响因素 [J]. 中国环境科学, 2021, 41(9): 4349-4359. doi: 10.3969/j.issn.1000-6923.2021.09.042 XU Y R, WU M L, WANG L, et al. Population changes and influencing factors of petroleum polluted soil microorganisms in northern Shaanxi oilfield area [J]. Chinese Environmental Sciences, 2021, 41(9): 4349-4359(in Chinese). doi: 10.3969/j.issn.1000-6923.2021.09.042
[19] 中国人民共和国生态环境部. 水质石油类的测定-紫外分光光度法(试行). HJ 970-2018[S]., Ministry of Ecology and Environment of the People’s Republic of China. Water quality—Determination of petroleum—Ultraviolet spectrophotometric method. HJ 970-2018[S].(inChinese)
[20] WU M L, LI W, DICK W A, et al. Bioremediation of hydrocarbon degradation in a petroleum-contaminated soil and microbial population and activity determination [J]. Chemosphere, 2017, 169: 124-130. doi: 10.1016/j.chemosphere.2016.11.059 [21] 8270C USEPA. Semivolatile Organic Compounds by Gas Chromatography/Mass Spectrometry (GC/qVIS) [S]. Revision 3ed. United States Environmental Protection Agency, 1996. [22] 8275A USEPA. Semivolatile Organic Compounds (PAHs and PCBs) in Soils/Sludges and Solid Wastes Using Thermal Extraction/Gas Chromatography/Mass Spectrometry fTE/GC/MS) [S]. Revision 1ed. United States Environmental Protection Agency, 1996. [23] 李可欣, 吴蔓莉, 高欢, 等. 基于流式细胞术的活性多环芳烃降解菌检测技术 [J]. 分析化学, 2021, 49(8): 1357-1365. doi: 10.19756/j.issn.0253-3820.201598 LI K X, WU M L, GAO H, et al. Determination of active polycyclic aromatic hydrocarbons degrading bacteria using flow cytometry [J]. Analytical Chemistry, 2021, 49(8): 1357-1365(in Chinese). doi: 10.19756/j.issn.0253-3820.201598
[24] 周婷, 陈吉祥, 杨智, 等. 一株嗜油不动杆菌(Acinetobacter oleivorans)的分离鉴定及石油降解特性 [J]. 环境工程学报, 2015, 9(11): 5626-5632. doi: 10.12030/j.cjee.20151178 ZHOU T, CHEN J X, YANG Z, et al. Isolation and characterization of an oil-degrading Acinetobacter oleivorans [J]. Environmental Engineering, 2015, 9(11): 5626-5632(in Chinese). doi: 10.12030/j.cjee.20151178
[25] 杨智, 陈吉祥, 李文新, 等. 1株铜绿假单胞菌(Pseudomonas aeruginosa)1217石油降解特性及相关基因分析 [J]. 微生物学杂志, 2017, 37(6): 70-80. doi: 10.3969/j.issn.1005-7021.2017.06.012 YANG Z, CHEN J X, LI W X, et al. Characterization and catabolic genes analyses of oil-degrading Pseudomonas aeruginosa 1217 [J]. Journal of Microbiology, 2017, 37(6): 70-80(in Chinese). doi: 10.3969/j.issn.1005-7021.2017.06.012
[26] 张晗, 吕占禄, 张金良, 等. 农村地区浅层地下水、沟塘水及沉积物中PAHs的污染特性及风险评估 [J]. 环境化学, 2021, 40(10): 3100-3111. doi: 10.7524/j.issn.0254-6108.2020062806 ZHANG H, LV Z L, ZHANG J L, et al. Pollution characteristics and risk assessment of PAHs in shallow groundwater, ditches and sediments in rural areas [J]. Environmental Chemistry, 2021, 40(10): 3100-3111(in Chinese). doi: 10.7524/j.issn.0254-6108.2020062806
[27] 王建龙, 夏旭, 冯伟. 不同类型生物滞留介质对雨水径流中多环芳烃的去除效果 [J]. 环境化学, 2020, 39(3): 670-676. doi: 10.7524/j.issn.0254-6108.2019102501 WANG J L, XIA X, FENG W, et al. Effect of different types of biological retention media on PAHs removal in rainwater runoff [J]. Environmental Chemistry, 2020, 39(3): 670-676(in Chinese). doi: 10.7524/j.issn.0254-6108.2019102501
[28] 文刚, 王静怡, 黄廷林, 等. 流式细胞仪在水处理中的应用现状与展望 [J]. 中国给水排水, 2014, 30(18): 58-62. doi: 10.19853/j.zgjsps.1000-4602.2014.18.013 WEN G, WANG J Y, HUANG T L, et al. Application of flow cytometer to water treatment: Status and perspectives [J]. China Water & Wastewater, 2014, 30(18): 58-62(in Chinese). doi: 10.19853/j.zgjsps.1000-4602.2014.18.013
-



 下载:
下载: