截至2020年12月21日,美国化学文摘社(www.cas.org)已经登记了1.55亿余种化合物,包括合金、配位化合物、矿物质、混合物、聚合物和盐等,并且还以每天1.5万余种的速度增加[1]。目前,以动物实验为主的化学品环境风险评估效率,还远不能满足大量进入市场的新化学品环境风险测评的需求。据统计,全世界平均每年动物实验花费近140亿美元,其中30亿用于毒理学实验[2]。而且,进入环境的日常使用化学品数目已经超过10万种,测试一个化合物毒性的平均周期为3.5年[2],如果逐一检测这些化学品的毒性,需要消耗大量的时间和金钱。定量结构-活性关系(quantitative structure-activity relationship, QSAR)模型可为化学品的暴露和效应模拟提供大量的基础参数[3]。目前,基于QSAR模型开发了许多毒性预测软件,例如EPI suite[4]、QSAR Toolbox[5]和ECOSAR[6]等,并且随着数据集的不断扩大,预测精度和应用域也在不断提升。
2008年,美国国家环境卫生科学研究所(NIEHS)、美国国家卫生院(NIH)和美国环境保护局(US EPA)签订了《高通量筛选、毒性途径分析和生物学解释》[7],正式开展Tox21计划。Tox21计划的毒性测试策略,由传统的以整体动物(in vivo)为基础的毒性测试体系转向基于人源细胞系或细胞组分的体外测试体系(in vitro),通过分析化合物暴露后细胞或细胞组分的变化,探究化合物对生物学基本过程的影响,大大提高了毒性测试的速度[8]。目前,Tox21已经完成了第二阶段工作,完成了1万多种化学品的筛选,并在PubChem等公共平台上实现了数据共享[2]。
Tox21计划的顺利实施使得毒性数据迅速增长,数据挖掘和机器学习的快速发展为计算毒理学提供了机遇和挑战。计算毒理学通过运用计算化学、生物信息学和系统生物学的跨学科知识,构建in silico模型,以实现化学品环境暴露、危害与风险的高效模拟,从而确定化学物质的潜在危害,减轻高通量化合物评估的压力[9]。计算毒理学在通量、成本和对化合物的可扩展性方面具有无可比拟的优势。目前的研究主要包含3个方面[10]:(1) 通过对高通量体外测试体系的数据挖掘,构建污染物影响复杂生命体和环境的相关知识库;(2) 通过蛋白质组学、转录组学、基因组学和代谢组学等相互作用网络的构建,结合计算机模拟等手段阐明污染物的致毒机制;(3) 建立具有预测功能的综合毒理学模型,以定量和全面地评估化合物的安全性。
2010年,Ankley等[11]提出了有害结局路径(adverse outcome pathway, AOP)框架,进一步阐述了毒性作用机制、毒性通路和生物学网络的含义。它假设外源物质与生物系统的初始相互作用作为分子起始事件(molecular initiating event, MIE),通过细胞信号传导等一系列关键事件(key events, KE),导致最终的有害结局(adverse outcome, AO)。AOP开发的模型涵盖了一系列毒性终点和化学品,包括纳米颗粒[12]和微塑料[13]等新型污染物。此外,数据挖掘和机器学习技术的不断发展为AOP框架提供了新的思路[14]。在大数据的时代背景下,AOP可以帮助识别风险评估中的关键信息,从而确定监管关注的重点,提高决策的效率(图1)。
确定MIE对发展AOP框架尤为重要。分子模拟能够从机理上解释实验现象,辅助化学品的风险评价工作,建立in silico预筛的优先化学品清单,有潜力阐明关键的MIE。化学品在触发MIE之后,会进一步激活细胞信号传导等一系列关键事件。随着高通量测序技术和多组学技术(基因组学、转录组学、蛋白质组学和代谢组学等)的不断进步,海量数据应运而生。面向多维度、多粒度和海量庞杂的各种组学数据,生物信息学作为一种分析和研究的手段发挥了不可替代的作用。生物信息学可通过网络模型来映射细胞生化组分的关系,例如DNA序列与转录获得的RNA或翻译得到的蛋白质之间的关系。由此产生的相互作用网络,在下游相关KE和AO的确定中发挥了重要作用。
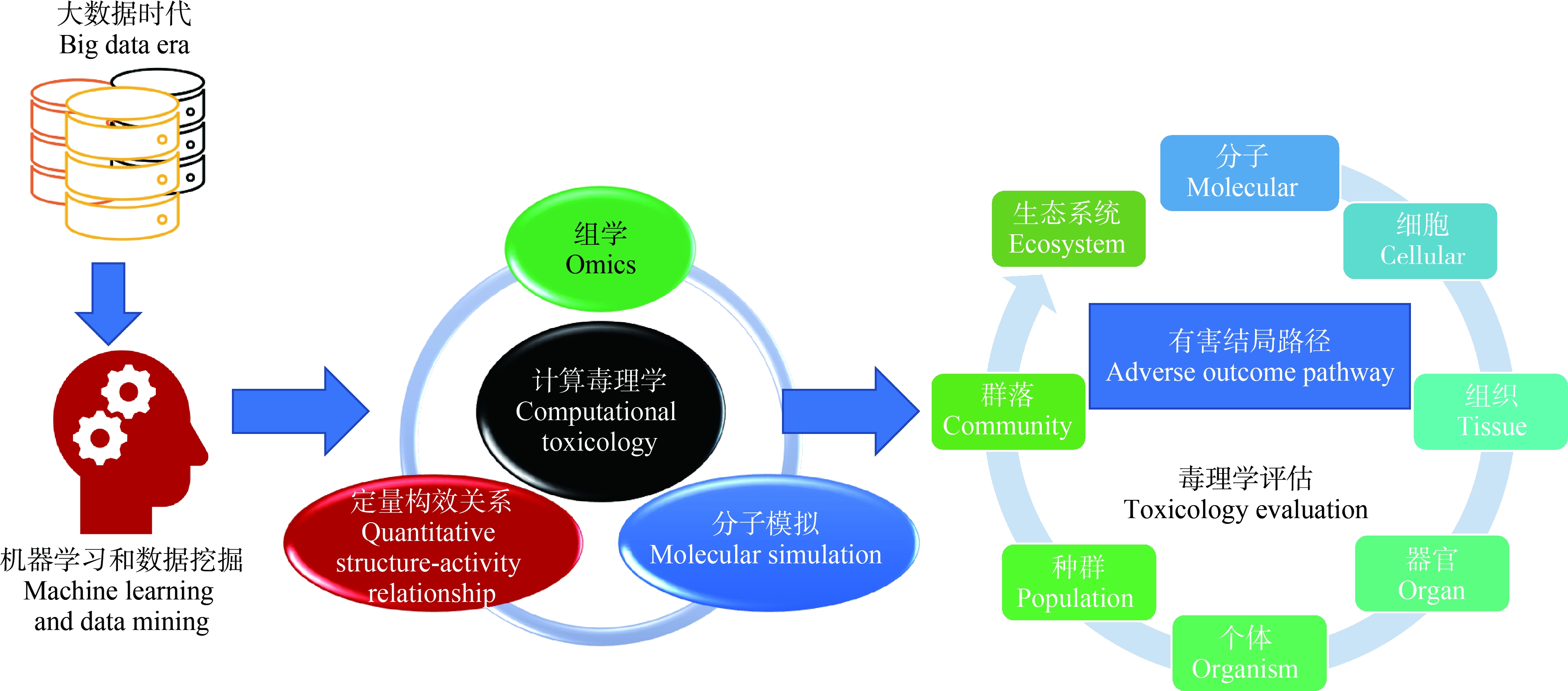
图1 毒理学评估的整体框架
Fig. 1 The overview of comprehensive toxicological assessment
目前,数据挖掘和机器学习在毒理学中的研究日益增多,AOP也迅速发展。在Web of Science数据库中输入关键词“machine learning toxicology or data mining toxicology”获取近10年来数据挖掘和机器学习在毒理学领域的文章发表数量;输入关键词“adverse outcome pathway”获取AOP文章的发表数量(图2)。因此本文在阐明数据挖掘和机器学习的基础上,结合AOP框架对计算毒理学发展现状进行了进一步总结。第1节对机器学习和数据挖掘进行了简要的概述;第2~4节分别介绍了QSAR模型、分子模拟和组学在毒理学中的应用;最后针对当前研究的不足提出了几条针对性建议,以更好地适应当前大数据时代的研究背景。
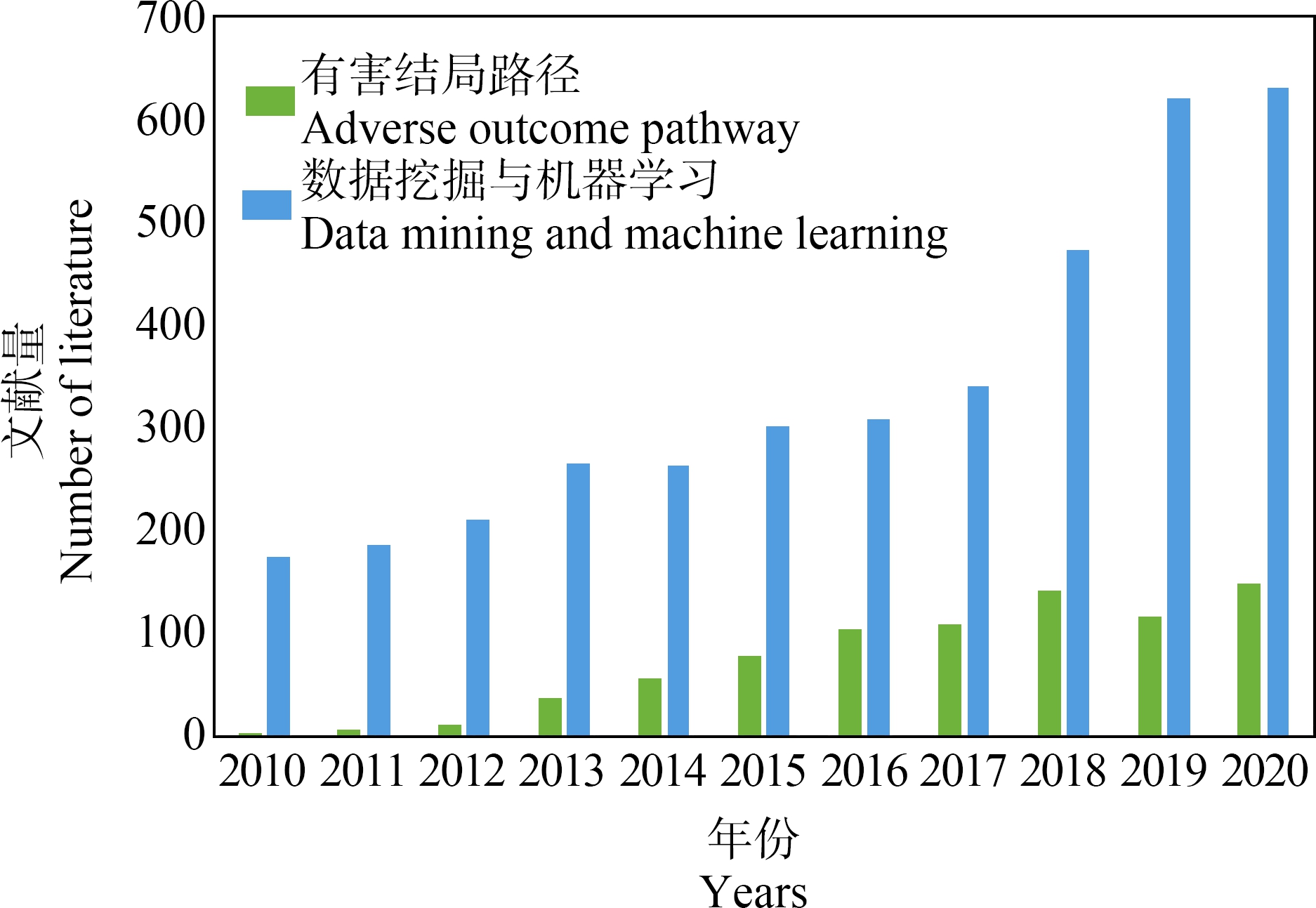
图2 近10年毒理学领域有害结局路径(AOP)
与数据挖掘和机器学习的文献发表情况
Fig. 2 Publication of data mining, machine learning
and adverse outcome pathway (AOP) in the
field of toxicology in the past ten years
1 机器学习和数据挖掘的概述(Overview of machine learning and data mining)
机器学习能从大数据集中提取复杂的模式和关系,以预测数据的特定属性[15]。经典统计建模方法一直是生物数据分析及其解释的标准选择。近年来,随着人工智能技术的日益普及和高通量数据的不断生成,机器学习在毒理学和健康风险领域也逐渐得到广泛关注[16]。传统模型通常是基于统计假设开发,相比之下机器学习可以挖掘隐藏在实验数据中的信息[17]。机器学习主要分为监督学习、无监督学习[18]和强化学习[19]。监督学习通过学习已有自变量和因变量的对应关系,推断出其他数据的功能,常用的算法包括支持向量机、决策树和线性回归等;无监督学习仅获取数据中的一组输入,并在数据中寻找共性,例如数据的聚类,常用的算法有K-means和DBSCAN;强化学习主要用于描述和解决智能体在与环境的交互过程中通过学习策略以达成回报最大化或实现特定目标的问题。在毒理学领域,研究人员使用各种计算方法开发了许多毒性预测模型(如支持向量机[20]、随机森林[21]、贝叶斯网络[22]和神经网络[23]等),在致癌性、致突变性和肝毒性等毒性终点方面也取得了重大进展。
数据挖掘与机器学习相辅相成,是从大型数据库中提取有价值信息并且发现新科学规律的过程[24]。数据挖掘任务要求研究人员对数据足够熟悉,以更好地对数据进行处理,达成应有的效果。毒性预测的准确性与数据质量和数量息息相关。在过去的几年中,广泛的数据收集已经建立了一些常用的毒性数据库。表1提供了常用数据源及其描述。
2 QSAR模型在毒理学中的应用(Application of QSAR models in toxicology)
QSAR主要包含分类模型和回归模型。分类模型根据正负类毒性的信息空间,构建数学模型对未知化合物进行毒性分类;回归模型可以预测单一的毒性值[33]。传统QSAR模型往往针对单一毒性终点进行构建,难以解决复杂的毒性机制。AOP的提出把污染物毒性按照机制进行描述和分类,克服了传统QSAR的关键难题,整合了孤立的单一毒性终点。在分子层面,可选择不同受体的结合活性开发QSAR模型,例如过氧化物酶体增殖物激活受体γ(PPARγ)、雌激素受体(ER)和雄激素受体(AR)等;在细胞层面,可针对细胞活性和细胞增殖等毒性终点构建相应的QSAR模型;在个体层面,可针对发育毒性、致死浓度等毒性终点开发相应的QSAR模型。
自Tox21计划实施以来,高通量数据集已成为数据挖掘任务的主要来源,它提高了模型的应用范围,有利于更大范围的化学品评估工作。例如,Tang等[34]利用Tox21高通量数据集和5种机器学习算法构建了QSAR分类模型。在进行数据预处理之后,作者筛选了4 811个数据点进行建模,将可预测化学品的领域扩展到杀虫剂、市售药品和商业化合物等,获得的最佳模型在十倍交叉验证和外部验证中的正确分类率达到了81.8%和88.3%,充分展现出模型的预测性能,提高了模型的应用域。此外,Capuzzi等[35]使用随机森林、深度神经网络和各种描述符建立了高通量筛选的12种核受体信号通路的QSAR模型。结果表明,在平衡数据集后,模型平衡准确度在58%和82%之间。总之,随着对高通量数据集研究的深入,未来毒性预测的质量也将不断提高。
尽管高通量筛选技术极大地提高了毒性数据质量和效率,但目前的Tox21检测获得的数据仍然有限。为完善AOP的构建,前人针对其他数据库展开了深入的挖掘。ECOTOX数据库提供了有关化学品对水生和陆生物种的毒性信息,合计超过400多万条测试记录。Cao等[36]基于ECOTOX数据库选取了824种化合物,结合6种机器学习方法与7种类型的分子指纹,开发了一系列QSAR模型。最佳模型外部验证集精度达到了0.87。Vegosen和Martin等[37]从新西兰环境保护机构、ACTOR数据库、ECHA数据库和TEST数据库中获取了85 000种化学品的信息,涉及急性口服毒性、急性吸入毒性和发育毒性等毒性终点,通过QSAR模型将所得化学品分为5级,完善了在个体层面的毒性风险评估。
表1 常用的毒性数据库及其描述
Table 1 Toxicity database and its description

数据库Database描述Description参考文献ReferencesToxCast高通量筛选的数千种化合物A collection of thousands of compounds by high-throughput screening[25]ACToR美国环境保护局(US EPA)进行高通量筛选、化学暴露和虚拟组织数据The U.S. Environmental Protection Agency (US EPA) CompTox warehouse containing high-throughput screening, chemical exposure and virtual tissues data[26]ChEMBL包含大量类似药物的生物活性化合物集合A public repository for a large number of drug-like bioactive compounds[27]OFF-X包含药物靶点和警示子结构A database contains targets and structural alerts[28]PubChem主要包含化合物和生物测定的大规模化学资料库数据库A publicly accessible platform for compounds and bioassays[29]Drugbank详细的药物数据和相应的药物靶标信息A source for combined drug and target data[30]ECOTOX提供有关水生生物、陆生植物和野生生物的单一环境毒性数据A comprehensive publicly available knowledgebase providing single chemical environmental toxicity data on aquatic life, terrestrial plants and wildlife[31]CTD整合了化学品、基因、表型、疾病和暴露的毒理学信息及其相互关系A publicly available database provides manually curated information about chemical-gene/protein interactions, chemical-disease and gene-disease relationships[32]
此外,在增强模型的可解释性上,前人亦做了深入探索。在大多数机器学习研究中,对可解释性的探索通常停留在揭示特征的重要性上,而忽略了多个特征联合作用下的关系。并且传统基于决策树的特征重要性评估往往取决于单一指标,所获得的结果可能与经验值大相径庭。为了解决此问题,Yu等[38]提出了一种随机森林特征重要性和特征交互网络分析框架,该框架通过多因素分析克服了小数据集引发的特征重要性偏差,并根据新的特征重要性排名建立了特征交互网络,提高了模型的可解释性,揭示了隐藏的交互因素对纳米材料毒性的影响。
3 分子模拟在毒理学中的应用(Application of molecular simulation in toxicology)
发掘污染物与生物分子间的相互作用的关系网络,探索分子作用的靶点和解析关键毒性通路,有助于评估污染物的生态风险性。锚定毒性作用的MIE是链接污染物AOP的关键环节,是协助构建AOP和完善毒性风险评估的基础。目前的实验仪器不完全具备观测微观分子原子运动过程的时空分辨率,而这些过程却蕴含了关键的机理。分子模拟作为计算毒理学中的一项新兴技术,可在分子尺度上构建化合物-生物大分子靶标的分子模拟模型,有助于探索分子水平的微观机理[39]。分子模拟的计算方法主要包括分子对接[40]和分子动力学[41]等。分子对接通过靶标和化学品之间能量匹配和空间匹配等实现相互识别,有利于分子间相互作用的快速评价;分子动力学是在牛顿力学的基础上描述分子运动时间演化的方法,属于经典力学的范畴,主要应用于蛋白质变换、通路机制分析以及小分子识别。
核受体是需要与配体结合后激活的一类转录因子,可调节与生物体发育和代谢控制相关的基因表达,经常作为AOP中的MIE,因此探究核受体的分子机制尤其关键。Li等[42]利用分子对接研究了不同条件下全氟烷基物质(PFASs)与雌激素受体(ER)蛋白的结合方式。结果表明,所有测试的PFASs均能结合在ER的表面域中,影响了ER与其他活化因子的结合。Xue等[43]通过数据挖掘与分子动力学相结合的方法,将ER与60个警示结构进行了模拟。结果显示,大部分警示结构都通过氢键或范德华力结合在ER的口袋两端并与氨基酸相互作用影响其表面构象,从而影响其活性。Cao等[44]利用分子对接和分子动力学模拟阐述全氟辛酸(PFOA)和全氟辛烷磺酰基化合物(PFOS)与ER结合特性,结果表明,PFOA和PFOS受到关键残基His524的质子化状态的调节。在亚酸性条件下,PFOA和PFOS更喜欢与质子化的His524形成氢键相互作用,从而导致共激活因子募集和转录激活。de Araujo等[45]将高亲和力配体与甲状腺激素受体(TR)的配体结合域对接,随后进行分子动力学模拟,获得了几种配体-复合物的结构模型。结果表明,配体极性头部的化学性质极大影响了受体-配体的亲和力,从而影响其结合效率。
虽然高通量筛选的快速发展使得化学品的筛选速度和体量得到了巨大提升,但仍旧存在许多问题[46],一是标准化学品的来源稀缺;二是高通量筛选的方法不够完善,检测的指标不足;三是高通量筛选的试剂和仪器都非常昂贵,许多实验室无法配备。因此在生物测试之前,可以采用虚拟筛选的方法确定优先级较高的化学品和靶点。虚拟筛选方法包含2类:一是基于化学品相似性,二是基于靶标结构。前者需要搭建一个药效团结构,从待筛化学品中选择出特定毒性的化学品清单;后者需要搭建一个靶蛋白模型,从待筛生物靶点中选择出体内作用靶点与代谢通路的清单。构建高通量虚拟筛选模型有利于MIE的确定。例如,Troger等[47]通过诱导契合对接的方法,找出了2种基于结构的药效团模型,并在DrugBank和Chemspace数据库中进行了虚拟筛选。筛选完成后,作者利用3种不同的机器学习模型进一步细化了命中列表,并选择了排名最靠前的化合物进行实验测试;通过层叠式方法,化合物在3种体外实验中得到了测试,最终成功识别出特殊的线粒体呼吸复合体抑制物。作者强调了虚拟筛选和机器学习在化学品筛选中的潜力,为基于结构的复杂毒理学终点预测提供依据。
4 组学在毒理学中的应用(Application of omics in toxicology)
高通量组学技术结合生物信息学分析为进一步研究关键靶点和毒性通路提供了新的思路,有望加速AOP的发展和应用。生物信息学在整合基因组学、转录组学、蛋白质组学和代谢组学的毒理学研究中发挥了重要作用,从序列比对到基因发现与功能研究,从基因表达到蛋白质结构与功能预测,乃至于更为复杂的调控网络、代谢网络及蛋白质相互作用网络等[48]。生物信息学作为以数据为驱动的组学研究领域常用技术,可以将化学品与其引发的DNA、RNA和蛋白质网络变化关联到一起,并进一步链接至in vitro或in vivo毒性终点,有利于进一步阐述KE和AO。利用生物信息学构建数据驱动的计算毒理学模型有利于综合评估污染物引起的毒理效应。
多组学数据的整合为AOP网络的构建提供了扎实的数据基础。例如,Wang等[49]利用基于生物信息学数据挖掘的多组学技术揭示了磷酸三苯酯毒性机制,发现磷酸三苯酯可以激活癌基因,损伤DNA,导致氧化还原失衡和影响代谢水平,从而诱导肝毒性。Kang等[50]通过对代谢组学和转录组学进行差异表达分析和富集分析阐明了小球藻应对氧化石墨烯的应激和恢复模式,发现氧化石墨烯通过减少小球藻氮元素的水平影响其三羧酸循环,从而进一步影响脂肪酸和氨基酸代谢等。Xia等[51]利用浓度依赖型简化转录组学揭示了短链氯化石蜡跨物种机制差异,发现短链氯化石蜡可能介导与神经毒性相关的KE。Song等[52]通过差异表达分析和富集分析方法研究转录组学数据发现,内分泌干扰物质(EDCs)可以通过影响ER活性而影响上皮基因的表达,导致上皮细胞的扩增,且减少了细胞凋亡的诱发,最终导致癌症的发生。
近年来相关毒理学数据库也在不断增加。例如,比较毒理学数据库(CTD)包含了230万种化学物质和4 700多万个毒性基因组关系[32]。通过生物信息学的方法对大量调控方式和作用网络的综合分析,有利于确定生物标志物和相关信号通路。![]() 等[53]通过CTD数据库筛选出邻苯二甲酸酯和双酚A(BPA)联合暴露导致肥胖的31个相关基因和蛋白质,共计471条相互作用。Yu等[54]在CTD中鉴定出145个氟化物暴露引起的骨损伤环境响应基因,并通过KEGG(kyoto encyclopedia of genes and genomes)富集证明骨损伤与Hippo信号通路显著相关。多组学数据反映了不同水平下的生物效应,机器学习可以有效地结合多组学数据进行预测,以便用于污染物的特征选择和分类。Peng等[55]整合多种机器学习模型和代谢组学数据,准确地预测了33种工程纳米颗粒(ENP)诱导的代谢途径。作者根据9种ENP的特性和随机森林算法构建了机器学习模型,最终模型的预测精度达到了75%,特征重要性评估表明工程ENP类型和大小是影响代谢途径的首要特征。Yamane等[56]采用多种污染物处理人类胚胎干细胞并分析其转录组变化,将化学物质分为神经毒素、基因毒性致癌物和非基因毒性致癌物等不同类型,并通过支持向量机构建了分类模型。
等[53]通过CTD数据库筛选出邻苯二甲酸酯和双酚A(BPA)联合暴露导致肥胖的31个相关基因和蛋白质,共计471条相互作用。Yu等[54]在CTD中鉴定出145个氟化物暴露引起的骨损伤环境响应基因,并通过KEGG(kyoto encyclopedia of genes and genomes)富集证明骨损伤与Hippo信号通路显著相关。多组学数据反映了不同水平下的生物效应,机器学习可以有效地结合多组学数据进行预测,以便用于污染物的特征选择和分类。Peng等[55]整合多种机器学习模型和代谢组学数据,准确地预测了33种工程纳米颗粒(ENP)诱导的代谢途径。作者根据9种ENP的特性和随机森林算法构建了机器学习模型,最终模型的预测精度达到了75%,特征重要性评估表明工程ENP类型和大小是影响代谢途径的首要特征。Yamane等[56]采用多种污染物处理人类胚胎干细胞并分析其转录组变化,将化学物质分为神经毒素、基因毒性致癌物和非基因毒性致癌物等不同类型,并通过支持向量机构建了分类模型。
5 总结与展望(Summary and prospect)
数据挖掘和机器学习在预测化学品毒性方面的关键作用已得到充分证明。毒理学数据的增长、计算能力的提高和计算方法的优化加速了计算毒理学在毒性预测和风险评估中的应用。但目前仍然存在一些挑战,例如:(1) 高度异构的数据源。不同数据库所记录的数据不一,需要开发能够整合多种数据库的工具。Neves等[57]已经利用KNIME平台开发了PubChem和CHEMBL数据处理的工作流,可为今后数据的整合提供借鉴。(2) 在许多已报道的研究工作中,在生成特征之前,清洗和标准化化合物的过程仍不清楚且无法重现。因此,设计更加透明、自动化的数据清洗流程以满足整合不同数据库数据的预处理显得十分必要。(3) 大多数机器学习模型通常被称为“黑箱模型”,难以对污染物的毒性机制进行合理解释。即使是高精确度的模型也不能轻易揭示这种预测背后的生物学机制[58]。因此开发具有透明机制和明确科学原理的“白箱”模型十分关键。(4) 分子描述符在建立QSAR模型中起着不可或缺的作用,有必要发展更多包含分子信息和可解释性的描述符。例如Seal等[59]提出可用细胞形态特征作为QSAR建模中新的描述符。新描述符可从不同角度对模型进行机理解释,从而提高模型的说服力和机理可解释性。(5) Tox21项目只专注于生物学的几个特定通路,目前的Tox21检测还远远不全面。同时在进行毒性评估的过程中,许多工作仅仅关注于毒性评估的某个方面,缺少对污染物的综合评估。(6) 生态风险评估的问题形成阶段是确定AOP的关键。试图阐明所有机制而不关心它们与特定结果的相关性将导致资源使用效率低下。因此可以从监管关注的重点出发,确定哪些毒性机制最有可能导致这些结果。总体而言,随着高通量筛选获得数据的不断累积以及计算机技术的不断更新,机器学习将在理解化学品毒性机制和风险防控领域起到关键作用。
通讯作者简介:李斐(1982—),女,博士,副研究员,主要研究方向为生态毒理学和计算毒理学。
共同通讯作者简介:于进福(1980—),男,高级工程师,主要研究方向为大数据挖掘和机器学习。
[1] Stokes W. The interagency coordinating committee on the validation of alternative methods (ICCVAM): Recent progress in the evaluation of alternative toxicity testing methods [R]. Bethesda: NTP Interagency Center for the Evaluation of Alternative Toxicological Methods, 2017
[2] 郭家彬, 彭双清. 动物实验替代方法与21世纪毒性测试发展策略[J]. 中国比较医学杂志, 2011, 21(S1): 157-161, 156
Guo J B, Peng S Q. Animal alternative methods and the development of strategy for toxicity testing in the Twenty-First Century [J]. Chinese Journal of Comparative Medicine, 2011, 21(S1): 157-161, 156 (in Chinese)
[3] 王中钰, 陈景文, 乔显亮, 等. 面向化学品风险评价的计算(预测)毒理学[J]. 中国科学: 化学, 2016, 46(2): 222-240
Wang Z Y, Chen J W, Qiao X L, et al. Computational toxicology: Oriented for chemicals risk assessment [J]. Scientia Sinica Chimica, 2016, 46(2): 222-240 (in Chinese)
[4] Card M L, Gomez-Alvarez V, Lee W H, et al. History of EPI SuiteTM and future perspectives on chemical property estimation in US Toxic Substances Control Act new chemical risk assessments [J]. Environmental Science Processes & Impacts, 2017, 19(3): 203-212
[5] Dimitrov S D, Diderich R, Sobanski T, et al. QSAR toolbox: Workflow and major functionalities [J]. SAR and QSAR in Environmental Research, 2016, 27(3): 203-219
[6] Fatoyinbo T, Rincon R F, Sun G Q, et al. Ecosar: A P- band digital beamforming polarimetric interferometric SAR instrument to measure ecosystem structure and biomass [C]// Vancouver, BC, Canada: IEEE International Geoscience and Remote Sensing Symposium, 2011: 1524-1527
[7] Tice R R, Austin C P, Kavlock R J, et al. Improving the human hazard characterization of chemicals: A Tox21 update [J]. Environmental Health Perspectives, 2013, 121(7): 756-765
[8] Shukla S J, Huang R L, Austin C P, et al. The future of toxicity testing: A focus on in vitro methods using a quantitative high-throughput screening platform [J]. Drug Discovery Today, 2010, 15(23-24): 997-1007
[9] Sturla S J, Boobis A R, FitzGerald R E, et al. Systems toxicology: From basic research to risk assessment [J]. Chemical Research in Toxicology, 2014, 27(3): 314-329
[10] 李杰, 李柯佳, 张臣, 等. 计算系统毒理学: 形成、发展及应用[J]. 科学通报, 2015, 60(19): 1751-1761
Li J, Li K J, Zhang C, et al. Computational systems toxicology: Emergence, development and application [J]. Chinese Science Bulletin, 2015, 60(19): 1751-1761 (in Chinese)
[11] Ankley G T, Bennett R S, Erickson R J, et al. Adverse outcome pathways: A conceptual framework to support ecotoxicology research and risk assessment [J]. Environmental Toxicology and Chemistry, 2010, 29(3): 730-741
[12] Jagiello K, Halappanavar S, Rybińska-Fryca A, et al. Transcriptomics-based and AOP-informed structure-activity relationships to predict pulmonary pathology induced by multiwalled carbon nanotubes [J]. Small, 2021, 17(15): e2003465
[13] Hu M Y, ![]() D A. Micro- and nano-plastics activation of oxidative and inflammatory adverse outcome pathways [J]. Redox Biology, 2020, 37: 101620
D A. Micro- and nano-plastics activation of oxidative and inflammatory adverse outcome pathways [J]. Redox Biology, 2020, 37: 101620
[14] Rugard M, Coumoul X, Carvaillo J C, et al. Deciphering adverse outcome pathway network linked to bisphenol F using text mining and systems toxicology approaches [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2020, 173(1): 32-40
[15] Jordan M I, Mitchell T M. Machine learning: Trends, perspectives, and prospects [J]. Science, 2015, 349(6245): 255-260
[16] Lu S H, Zhou Q H, Ouyang Y X, et al. Accelerated discovery of stable lead-free hybrid organic-inorganic perovskites via machine learning [J]. Nature Communications, 2018, 9(1): 3405
[17] Caruana R, Niculescu-Mizil A. An empirical comparison of supervised learning algorithms [C]//Proceedings of the 23rd International Conference on Machine Learning. Pittsburgh: ACM Press, 2006
[18] Sathya R, Abraham A. Comparison of supervised and unsupervised learning algorithms for pattern classification [J]. International Journal of Advanced Research in Artificial Intelligence, 2013, 2(2): 34-38
[19] Hu J Y, Niu H L, Carrasco J, et al. Voronoi-based multi-robot autonomous exploration in unknown environments via deep reinforcement learning [J]. IEEE Transactions on Vehicular Technology, 2020, 69(12): 14413-14423
[20] Qin R, Wang H, Yan A. Classification and QSAR models of leukotriene A4 hydrolase (LTA4H) inhibitors by machine learning methods [J]. SAR and QSAR in Environmental Research, 2021, 32(5): 411-431
[21] Ha M K, Trinh T X, Choi J S, et al. Toxicity classification of oxide nanomaterials: Effects of data gap filling and PChem score-based screening approaches [J]. Scientific Reports, 2018, 8(1): 3141
[22] Furxhi I, Murphy F, Poland C A, et al. Application of Bayesian networks in determining nanoparticle-induced cellular outcomes using transcriptomics [J]. Nanotoxicology, 2019, 13(6): 827-848
[23] Drgan V, Bajželj B. Application of supervised SOM algorithms in predicting the hepatotoxic potential of drugs [J]. International Journal of Molecular Sciences, 2021, 22(9): 4443
[24] Ge Z Q, Song Z H, Ding S X, et al. Data mining and analytics in the process industry: The role of machine learning [J]. IEEE Access, 2017, 5: 20590-20616
[25] United States Environmental Protection Agency (US EPA). Exploring ToxCast Data [EB/OL]. [2021-09-15]. https://www.epa.gov/chemical-research/exploring-toxcast-data-downloadable-data
[26] United States Environmental Protection Agency (US EPA). ACToR Safer Chemicals Research [EB/OL]. [2021-09-15]. https://19january2017snapshot.epa.gov/chemical-research/actor.html
[27] European Bioinformatics Institute. ChEMBL: A large-scale bioactivity database for drug discovery [EB/OL]. [2021-09-15]. https://www.ebi.ac.uk/chembl/
[28] Bioinfogate. OFF-X Website [EB/OL]. [2021-09-15]. https://www.targetsafety.info/
[29] National Library of Medicine. PubChem:A public information system for analyzing bioactivities of small molecules. [EB/OL]. [2021-09-15]. https://pubchem.ncbi.nlm.nih.gov/
[30] Wishart D S. DrugBank Online. Database for Drug and Drug Target Info. [EB/OL]. [2021-09-15]. https://go.drugbank.com/
[31] United States Environmental Protection Agency (US EPA). ECOTOX Knowledgebase [EB/OL]. [2021-09-15]. https://cfpub.epa.gov/ecotox/
[32] National Institute of Environmental Health Sciences. The Comparative Toxicogenomics Database [EB/OL]. [2021-09-15]. https://ctdbase.org/
[33] Cherkasov A, Muratov E N, Fourches D, et al. QSAR modeling: Where have You been? Where are you going to? [J]. Journal of Medicinal Chemistry, 2014, 57(12): 4977-5010
[34] Tang W H, Chen J W, Hong H X. Discriminant models on mitochondrial toxicity improved by consensus modeling and resolving imbalance in training [J]. Chemosphere, 2020, 253: 126768
[35] Capuzzi S J, Politi R, Isayev O, et al. QSAR modeling of Tox21 challenge stress response and nuclear receptor signaling toxicity assays [J]. Frontiers in Environmental Science, 2016, 4: 3
[36] Cao Q Q, Liu L, Yang H B, et al. in silico estimation of chemical aquatic toxicity on crustaceans using chemical category methods [J]. Environmental Science Processes & Impacts, 2018, 20(9): 1234-1243
[37] Vegosen L, Martin T M. An automated framework for compiling and integrating chemical hazard data [J]. Clean Technologies and Environmental Policy, 2020, 22(2): 441-458
[38] Yu F B, Wei C H, Deng P, et al. Deep exploration of random forest model boosts the interpretability of machine learning studies of complicated immune responses and lung burden of nanoparticles [J]. Science Advances, 2021, 7(22): eabf4130
[39] Rabinowitz J R, Goldsmith M R, Little S B, et al. Computational molecular modeling for evaluating the toxicity of environmental chemicals: Prioritizing bioassay requirements [J]. Environmental Health Perspectives, 2008, 116(5): 573-577
[40] Walden D M, Bundey Y, Jagarapu A, et al. Molecular simulation and statistical learning methods toward predicting drug-polymer amorphous solid dispersion miscibility, stability, and formulation design [J]. Molecules, 2021, 26(1): E182
[41] Mazurek A H, Szeleszczuk  , Pisklak D M. A review on combination of ab initio molecular dynamics and NMR parameters calculations [J]. International Journal of Molecular Sciences, 2021, 22(9): 4378
, Pisklak D M. A review on combination of ab initio molecular dynamics and NMR parameters calculations [J]. International Journal of Molecular Sciences, 2021, 22(9): 4378
[42] Li J, Cao H M, Feng H R, et al. Evaluation of the estrogenic/antiestrogenic activities of perfluoroalkyl substances and their interactions with the human estrogen receptor by combining in vitro assays and in silico modeling [J]. Environmental Science & Technology, 2020, 54(22): 14514-14524
[43] Xue Q, Liu X, Liu X C, et al. The effect of structural diversity on ligand specificity and resulting signaling differences of estrogen receptor Α [J]. Chemical Research in Toxicology, 2019, 32(6): 1002-1013
[44] Cao H M, Wang L, Liang Y, et al. Protonation state effects of estrogen receptor α on the recognition mechanisms by perfluorooctanoic acid and perfluorooctane sulfonate: A computational study [J]. Ecotoxicology and Environmental Safety, 2019, 171: 647-656
[45] de Araujo A S, Martínez L, de Paula Nicoluci R, et al. Structural modeling of high-affinity thyroid receptor-ligand complexes [J]. European Biophysics Journal, 2010, 39(11): 1523-1536
[46] Subramaniam S, Mehrotra M, Gupta D. Virtual high throughput screening (vHTS): A perspective [J]. Bioinformation, 2008, 3(1): 14-17
[47] Troger F, Delp J, Funke M, et al. Identification of mitochondrial toxicants by combined in silico and in vitro studies: A structure-based view on the adverse outcome pathway [J]. Computational Toxicology, 2020, 14: 100123
[48] Kanehisa M, Bork P. Bioinformatics in the post-sequence era [J]. Nature Genetics, 2003, 33(Suppl.): 305-310
[49] Wang X Q, Li F, Liu J L, et al. Transcriptomic, proteomic and metabolomic profiling unravel the mechanisms of hepatotoxicity pathway induced by triphenyl phosphate (TPP) [J]. Ecotoxicology and Environmental Safety, 2020, 205: 111126
[50] Kang W L, Li X K, Sun A Q, et al. Study of the persistence of the phytotoxicity induced by graphene oxide quantum dots and of the specific molecular mechanisms by integrating omics and regular analyses [J]. Environmental Science & Technology, 2019, 53(7): 3791-3801
[51] Xia P, Peng Y, Fang W D, et al. Cross-model comparison of transcriptomic dose-response of short-chain chlorinated paraffins [J]. Environmental Science & Technology, 2021, 55(12): 8149-8158
[52] Song Y, Villeneuve D L, Toyota K, et al. Ecdysone receptor agonism leading to lethal molting disruption in arthropods: Review and adverse outcome pathway development [J]. Environmental Science & Technology, 2017, 51(8): 4142-4157
![]() D, et al. Potential genomic biomarkers of obesity and its comorbidities for phthalates and bisphenol A mixture: In silico toxicogenomic approach [J]. BIOCELL, 2022, 46(2): 519-533
D, et al. Potential genomic biomarkers of obesity and its comorbidities for phthalates and bisphenol A mixture: In silico toxicogenomic approach [J]. BIOCELL, 2022, 46(2): 519-533
[54] Yu F F, Zuo J, Fu X L, et al. Role of the hippo signaling pathway in the extracellular matrix degradation of chondrocytes induced by fluoride exposure [J]. Ecotoxicology and Environmental Safety, 2021, 225: 112796
[55] Peng T, Wei C H, Yu F B, et al. Predicting nanotoxicity by an integrated machine learning and metabolomics approach [J]. Environmental Pollution, 2020, 267: 115434
[56] Yamane J, Aburatani S, Imanishi S, et al. Prediction of developmental chemical toxicity based on gene networks of human embryonic stem cells [J]. Nucleic Acids Research, 2019, 47(3): 1600
[57] Neves B, Moreira-Filho J, Silva A, et al. Automated framework for developing predictive machine learning models for data-driven drug discovery [J]. Journal of the Brazilian Chemical Society, 2021: 110-122
[58] Chen H M, Engkvist O, Wang Y H, et al. The rise of deep learning in drug discovery [J]. Drug Discovery Today, 2018, 23(6): 1241-1250
[59] Seal S, Yang H B, Vollmers L, et al. Comparison of cellular morphological descriptors and molecular fingerprints for the prediction of cytotoxicity- and proliferation-related assays [J]. Chemical Research in Toxicology, 2021, 34(2): 422-437