了解化学品的毒性机制是开展化学品人类健康风险评估的基础。化学品污染已经成为严重威胁人类健康的重要因素[1]。一些环境化学品的暴露可导致诸多人类慢性疾病,例如肺癌、神经退行性疾病等[2-4]。了解和掌握化学品的毒性机制是准确识别介导化学品有害效应的关键分子事件的基础,而这对于指导检测化学品的最大无效应剂量(no observed adverse effect level, NOAEL)是必要的[5-8]。毒理基因组学已经被广泛应用于环境化学品的毒性机制研究中。而传统的毒理基因组学方法,例如:转录组学,通过对差异表达基因的识别与相关的生物学通路的分析,可以提供化学品潜在的毒性机制信息[9-11]。但这些分子信息缺乏直接的生物学表型的关联,因此不能直接将这些分子信息与化学品的有害效应和毒性机制关联起来[11-13]。
掌握化学品有害效应的易感性机制是开展精准的人类健康风险评估的重要基础[14-17]。由于人类个体易感性差异的存在,使得当前的化学品人类健康风险评估往往存在不确定性[5-6, 14, 18]。遗传变异由于可以在亲本和子代间进行传递,且可以直接在分子水平影响化学品暴露导致的有害效应,因此由遗传变异介导的易感性差异受到普遍关注[14-15]。传统的分子流行病学方法,例如:全基因组关联分析研究(genome-wide association studies, GWAS)和基因组-环境关联分析研究(gene-environment-wide association studies, GEWAS)基于关联性统计分析,来研究表型与遗传变异之间的关联(图1),难以提供相关的分子机制的解析[17, 19]。另外,传统的毒理基因组学也不能提供与遗传易感性关联的分子响应信息。通过测试细胞的表型,功能基因组学可以识别基因与化学品有害效应的直接关联,进而揭示与化学品有害效应直接关联的分子机制[20-22],同时提供与遗传易感性相关的分子响应信息[23]。功能基因组学已经被广泛应用于环境化学品的毒性机制研究中。目前,依赖于基因编辑技术的发展,功能基因组学已经从传统的单细胞生物(酵母)发展到人类细胞,同时实现了全基因组水平的基因敲除[13, 20]。这些功能基因组学方法拥有各自的优点与局限性,适合不同类型的化学品的生物学测试。
本论文综述了功能基因组学的原理及其主要发展,介绍了不同功能基因组学方法的优点和局限性。重点详述了CRISPR-Cas9功能基因组学的原理与特点,以及其在环境化学品毒性机制研究中的应用。最后,本论文对联合运用CRISPR-Cas9功能基因组学与分子流行病学分析,开展化学品有害效应的易感性机制研究进行了展望。
1 传统的功能基因组学(Conventional functional genomics)
1.1 功能基因组学的原理与特点1.1.1 功能基因组学的原理
功能基因组学通过检测化学品暴露后的表型,筛选出与生物表型直接关联的基因。其中所检测的表型既可以是细胞活性,也可以是特定的细胞毒性终点,例如氧化应激、内质网应激等。通过分析筛选出的基因,可以获取与特定生物表型或毒性终点直接关联的生物学通路,进而获取介导化学品有害效应的关键基因和分子通路[20]。目前,功能基因组学的筛选介质已经从传统的酵母细胞[24]发展至活体动物[25],但活体动物的筛选目前仅用于癌症药物研发,尚未应用于环境化学品的毒性测试。而功能基因组学所仰仗的基因编辑技术,已经从传统的RNA干扰(RNA interference, RNAi)[26]发展至当前主流的CRISPR技术[27]。
功能基因组学的原理:依靠基因编辑技术,在细胞中敲除(knockout)或者敲降(knockdown)全基因组或者特定的基因集,并且实现一个细胞中只敲除或敲降一个基因。与环境化学品有害效应关联的基因,它们的功能缺失会使细胞对化学品的有害效应更加敏感或者更具抗性,通过检测化学品暴露后细胞的表型,可以将这些敏感性变化的细胞筛选出来。因为每个细胞内均有一个特定的标签序列(barcode),可以通过高通量测序的方法,识别筛选出的细胞中被敲除或者敲降的基因,进而将与环境化学品有害效应关联的基因筛选出来(图2)[12, 20, 28]。
1.1.2 功能基因组学的特点
功能基因组学的特点主要包括:
(1)功能基因组学识别的基因具有“表型锚定”。功能基因组学通过检测化学品暴露后的细胞表型(这些细胞表型可以是细胞活性,或者是基于特定毒性终点的荧光信号),识别敏感性变化的突变细胞,进而筛选出与化学品有害效应或者细胞毒性终点直接关联的基因。这些基因的功能缺失使细胞对相应的化学品的有害效应变得更敏感或者更加抗性,体现在毒性机制上,这些基因的功能就是介导化学品细胞毒性或者抑制化学品的细胞毒性。因此,功能基因组学可以凭借构建基因与化学品的有害效应或者毒性终点的直接关联,发掘环境化学品的新颖的毒性机制[12, 20]。
(2)功能基因组学可以提供基于遗传变异的分子视角。功能基因组学通过基因编辑技术,尤其以CRISPR-Cas9为代表的当前主流的基因编辑技术,对基因的DNA序列进行编辑,使其产生突变,形成基因功能缺失。或者通过RNA干扰技术降低基因的表达。这些基因的表达被抑制,进而导致细胞对化学品的暴露产生敏感性变化。人体的有效应的遗传变异,主要分为两大类:一是影响基因的表达,二是影响蛋白的活性,而这2种效应突变均体现在了基因功能的变化上[29]。基因功能的变化进而导致人体细胞对化学品暴露的敏感性发生变化[14-15]。因此,功能基因组学可以提供具有遗传变异视角的化学品毒性机制[30]。

图1 全基因组关联分析研究(GWAS)和基因组-环境关联分析研究(GEWAS)通过相关性分析
预测遗传变异与疾病表型的关联
Fig. 1 The associations between genetic variants and disease phenotypes were predicted by genome-wide association
studies (GWAS) and gene-environment-wide association studies (GEWAS) through correlation analysis

图2 功能基因组学的原理
Fig. 2 The mechanism of functional genomics screen
(3)功能基因组学同样可以用于化学品高通量测试。利用流式细胞仪筛选和高通量测序,可以实现多种化学品多剂量暴露下的表型检测和基因功能缺失的识别,进而可以开展更多的化学品的机制毒理学研究[20]。
1.2 经典的功能基因组学
早期的功能基因组学根据生物载体和基因突变的方式可以分为:单细胞生物的功能基因组学(大肠杆菌和酵母)、鸡的DT40细胞的筛选、单倍体哺乳动物细胞的筛选和RNA干扰。这些传统的功能基因组学有各自的优点,在对于某些特定作用机制的化学品,例如:遗传毒性化学品的检测,仍然具有较多的应用价值。但这些方法也存在不少局限性,主要集中于和人类之间的物种差异以及能否实现全基因组水平的基因敲除(表1)。因此,当前的功能基因组学筛选需要一种基于人类细胞的且可以实现全基因组水平的基因的特异性敲除的技术。由于大肠杆菌属于原核生物,与人类的物种差异较大,在此,本文主要综述真核生物的功能基因组学方法。
1.2.1 酵母的功能基因组学
以酵母作为生物载体的功能基因组学筛选,已经被广泛用于环境化学品的毒性测试,尤其以遗传毒性化学品为代表。酵母作为功能基因组学的生物载体,有以下优势:(1) 酵母是一种常见的真核单细胞生物,无论是单倍体或者双倍体,酵母均是一种易扩繁、易培养的非致病性微生物;(2) 酵母的基因组已经完全解析,比较容易实现全基因组的编辑和敲除[33, 45]。(3) 酵母的基因组在进化上与其他高等真核生物保持了较高的同源性,与人类遗传疾病相关的一半的基因都具有酵母的同源基因。对于环境化学品的暴露,酵母细胞的基本响应的分子机制与人类细胞和其他高等真核生物是一致的[31, 46]。
在解析响应环境化学品暴露的基因功能的时候,酵母突变株的表型分析是一种有效的方法。构建酵母细胞的突变株,目前主要包括:遗传印迹法、随机突变法和基于聚合酶链式反应(polymerase chain reaction, PCR)的基因突变法[20]。目前已有较多种类的商业化的酵母突变株细胞库,包括删除了5 916个基因(含有1 159个必需基因)的纯合子和杂合子细胞库,以及删除了4 757个非必需基因的单倍体细胞库。这些酵母突变株均含有特异性的标签序列,可以开展细胞库的筛选研究[32]。目前,已有大量研究使用酵母的突变细胞库进行环境化学品的毒性测试,主要包括遗传毒性化学品和抑制氧化磷酸化的化学品[20],例如苯并芘、甲醛和砷[21, 47-48]。使用酵母突变细胞库开展这些化学品的功能基因组筛选的优势在于:这些化学品的作用机制是通过造成DNA损伤或是线粒体呼吸链传递的抑制,而这些生物学过程的分子机制在真核生物物种进化过程中比较保守,便于进行同源基因的物种外推[49-50]。
虽然酵母的功能基因组学筛选在识别环境化学品的比较保守的分子机制中可以发挥重要作用,但酵母突变株细胞库在环境化学品的毒性测试中依然有局限性:(1) 酵母是一种单细胞生物,对于涉及细胞间相互作用的分子机制,无法依靠酵母的功能基因组学开展研究;(2) 酵母细胞可以忍受更高剂量化学品的暴露,它的一些效应浓度往往远高于人体细胞相应化学品的效应浓度;(3) 虽然酵母基因组中有很多人类的同源基因,但会出现一个酵母基因同时对应多个人类基因的情况,这给分子机制的外推带来了困难。因此,需要使用与人类物种差异更小的物种开展功能基因组学研究[20-21]。
1.2.2 鸡DT40细胞的功能基因组学
在鸡DT40细胞中可以通过靶向整合(targeted integration)技术较为容易地实现基因敲除,因为该细胞具有非常高的靶向整合效率[51]。目前,DT40细胞主要应用于遗传毒性化学品的功能基因组学的筛选[52]。DT40细胞具有以下特点使其适合应用于遗传毒性化学品的毒性测试:(1) DT40细胞分裂具有较长的S期,大概占细胞循环的70%,这意味着在遗传毒性化学品的暴露期间,大多数的DT40细胞处于DNA合成阶段,这使细胞的DNA更容易遭受损伤。(2) 因为DT40细胞P53基因功能缺失,DT40细胞的细胞循环的G1/S检测点是没有激活的,因此就会导致在G1期没有被完全修复的DNA损伤会随着细胞循环不断积累,进而影响DNA复制。以上2个特征使DT40细胞对化学品的遗传毒性相较于其他类型细胞更敏感[52]。
目前的DT40细胞的突变株主要遍及2类与DNA损伤修复相关的生物学通路。(1)DNA复制:跨损伤修复合成(translesion synthesis, TLS)、同源修复(homologous recombination, HR)、范科尼贫血修复通路(Fanconi anemia repair)、非同源末端连接(non-homologous end joining)、链间交联修复(interstrand cross-link repair)、核苷酸剪切修复(nucleotide excision repair)和碱基剪切修复(base excision repair);
表1 不同功能基因组学方法比较
Table 1 Comparison between different functional genomics
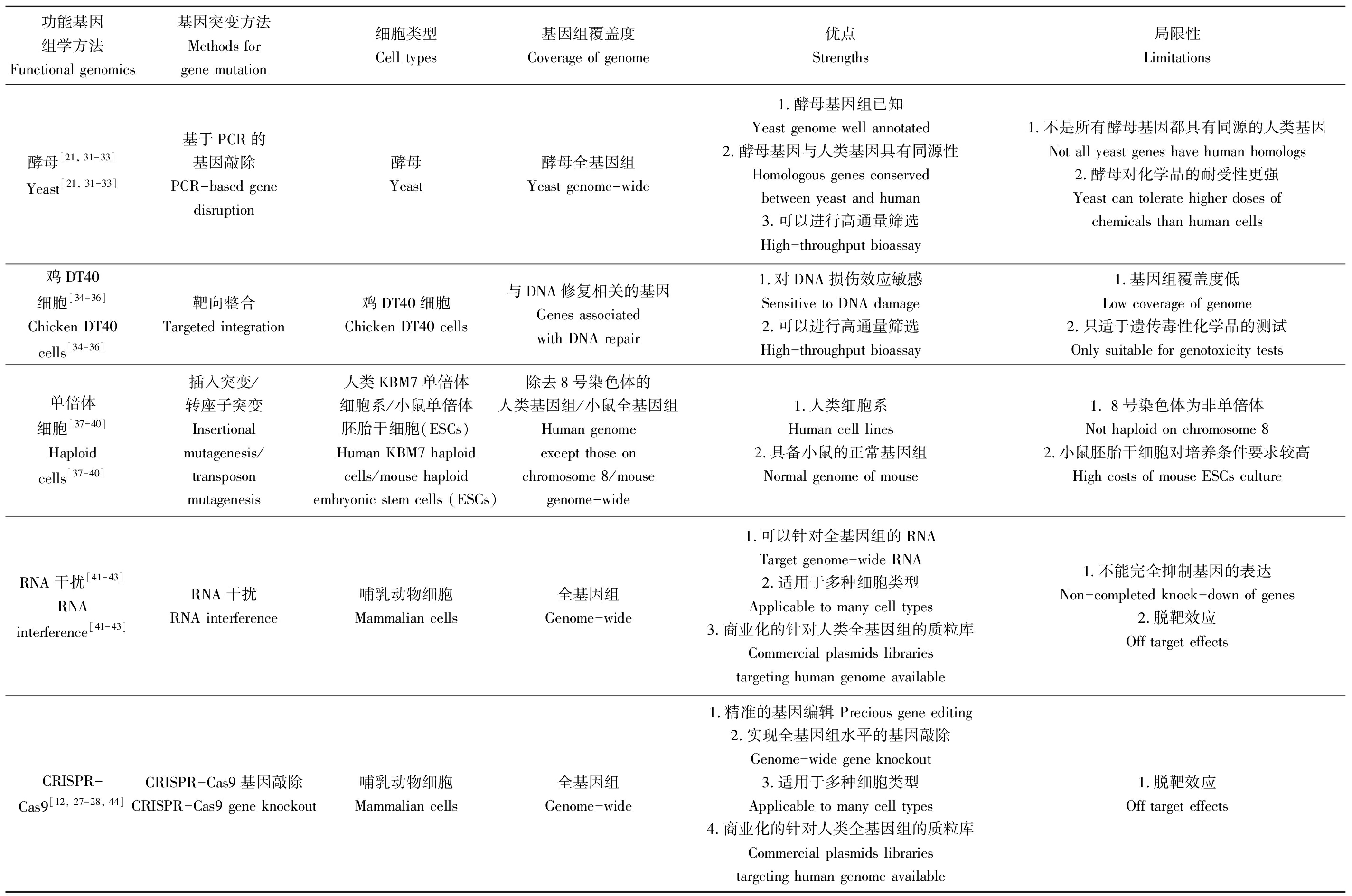
功能基因组学方法Functional genomics基因突变方法Methods for gene mutation细胞类型Cell types基因组覆盖度Coverage of genome优点Strengths局限性Limitations酵母[21, 31-33]Yeast[21, 31-33]基于PCR的基因敲除PCR-based gene disruption酵母 Yeast酵母全基因组Yeast genome-wide1.酵母基因组已知Yeast genome well annotated2.酵母基因与人类基因具有同源性Homologous genes conserved between yeast and human3.可以进行高通量筛选High-throughput bioassay1.不是所有酵母基因都具有同源的人类基因Not all yeast genes have human homologs2.酵母对化学品的耐受性更强Yeast can tolerate higher doses of chemicals than human cells鸡DT40细胞[34-36]Chicken DT40 cells[34-36]靶向整合Targeted integration鸡DT40细胞Chicken DT40 cells与DNA修复相关的基因Genes associated with DNA repair1.对DNA损伤效应敏感Sensitive to DNA damage2.可以进行高通量筛选High-throughput bioassay1.基因组覆盖度低Low coverage of genome2.只适于遗传毒性化学品的测试Only suitable for genotoxicity tests单倍体细胞[37-40]Haploid cells[37-40]插入突变/转座子突变Insertionalmutagenesis/transposonmutagenesis人类KBM7单倍体细胞系/小鼠单倍体胚胎干细胞(ESCs)Human KBM7 haploid cells/mouse haploid embryonic stem cells (ESCs)除去8号染色体的人类基因组/小鼠全基因组Human genomeexcept those onchromosome 8/mouse genome-wide1.人类细胞系Human cell lines2.具备小鼠的正常基因组Normal genome of mouse1. 8号染色体为非单倍体Not haploid on chromosome 82.小鼠胚胎干细胞对培养条件要求较高High costs of mouse ESCs cultureRNA干扰[41-43]RNA interference[41-43]RNA干扰RNA interference哺乳动物细胞Mammalian cells全基因组Genome-wide1.可以针对全基因组的RNATarget genome-wide RNA2.适用于多种细胞类型Applicable to many cell types3.商业化的针对人类全基因组的质粒库Commercial plasmids libraries targeting human genome available1.不能完全抑制基因的表达Non-completed knock-down of genes2.脱靶效应Off target effectsCRISPR-Cas9[12, 27-28, 44]CRISPR-Cas9基因敲除CRISPR-Cas9 gene knockout哺乳动物细胞Mammalian cells全基因组Genome-wide1.精准的基因编辑 Precious gene editing2.实现全基因组水平的基因敲除Genome-wide gene knockout3.适用于多种细胞类型Applicable to many cell types4.商业化的针对人类全基因组的质粒库Commercial plasmids libraries targeting human genome available1.脱靶效应Off target effects
(2)细胞循环:DNA损伤检查点(DNA damage checkpoint) [52]。共有超过100种DT40细胞突变株遍及以上DNA损伤修复通路[35, 52]。DT40细胞突变株由于DNA损伤修复的基因功能缺失,导致无法修复遗传毒性化学品的毒性效应导致的DNA损伤,进而抑制了细胞增殖,导致细胞死亡[53]。基于此原理,目前已经开展了较多利用DT40突变细胞库进行遗传毒性化学品毒性测试的研究。Ooka等[36]利用DT40细胞功能基因组学筛选发现,对于苯并芘(BaP)和N-亚硝基二甲胺(NDMA),需要使用S9代谢激活才能显示出遗传毒性。跨损伤修复合成缺陷的REV3-/- DT40细胞对BaP和NDMA均表现出最高的敏感性,此外双链断裂修复缺陷的RAD54-/-/KU70-/- DT40细胞也对BaP表现出较高的敏感性。Ooka等[36]使用酵母的功能基因组筛选发现跨损伤修复合成通路在三氯乙烯代谢产物的遗传毒性分子机制中扮演重要作用,此外,通过DT40的筛选发现,跨损伤修复合成通路缺陷的DT40细胞对三氯乙烯代谢产物更敏感,相比之下,同源重组修复缺陷的DT40细胞对三氯乙烯代谢产物抗性比野生型DT40细胞更强。Ji等[54]使用DT40筛选发现碱基剪切修复缺陷Polβ-/-和跨损伤修复合成缺陷REV3-/- DT40细胞对tetra-BDEs和OH-tetra-BDEs的敏感性比野生型更高。使用半胱氨酸预处理可以降低Polβ-/-和REV3-/- DT40细胞的敏感性,表明多溴联苯醚(PBDEs)和羟基-多溴联苯醚(OH-BDEs)通过引发氧化应激导致遗传毒性效应。
DT40细胞的功能基因组筛选的局限性表现在:(1) DT40细胞来源于鸡的淋巴B细胞,与人类存在物种差异,不利于进行跨物种外推;(2) DT40细胞的功能基因组学筛选不能覆盖全基因组水平,只是集中于DNA损伤修复通路的基因的研究。
1.2.3 单倍体哺乳动物细胞的功能基因组学
人类近单倍体细胞的功能基因组学筛选已经被广泛应用于识别与化学品细胞毒性相关的基因。人类KBM7细胞来源于白血病病人的骨髓,该病人患有近单倍体慢性髓系白血病[55]。P1-55亚克隆细胞系的构建,使得细胞的单倍体构型可以保持至少12周,此外,改良后的KBM7 (HAP1)[56]是贴壁生长细胞,其8号染色体为单倍体。这使得使用KBM7细胞系进行功能基因组学筛选成为可能[37]。Birsoy等[57]将其应用于溴丙酮酸的抗性机制的识别,溴丙酮酸是一种糖酵解抑制剂,所识别的关键基因MCT1所编码的蛋白是溴丙酮酸的转运蛋白,且MCT1的mRNA水平可以被用来预测癌细胞对溴丙酮酸的敏感性,因此也被看作为肿瘤的潜在生物标志物。Shen等[58]利用KBM7细胞的功能基因组学识别了与甲醛和格列卫的遗传毒性相关的基因,实现了利用单倍体细胞的功能基因组学揭示遗传毒性化学品的分子机制的先例。目前,人类近单倍体细胞的功能基因组学筛选往往以细胞死亡作为检测表型,以非细胞死亡作为毒性终点的功能基因组学研究也已经发表。Duncan等[39]利用人类单倍体细胞筛选,基于细胞荧光检测,通过筛选MHC1的细胞表面表达缺陷的突变细胞,识别了与MHC一类抗原呈递相关的基因。Lee等[59]利用转录因子报告基因,筛选了KBM7细胞中参与NF-κB的激活抑制的基因,并发现了新的分子机制。
目前可以多代传代的小鼠的单倍体胚胎干细胞已经被构建,这些单倍体细胞依然具有诱导分化的能力,且分化后依然保持单倍体核型。这些细胞的构建使得研究环境化学品对正常干细胞的毒性效应以及模拟早期生命暴露提供了可能[60-61]。Elling等[60]利用转座子介导的近于全基因组的基因突变,识别了与介导蓖麻毒素相关的基因。此外,小鼠的单倍体胚胎干细胞的功能基因组学被应用识别与6-巯基嘌呤的毒性关联的DNA错配修复基因[40]和介导奥拉帕尼毒性的基因[38]。以上这些研究发掘了小鼠的单倍体胚胎干细胞的功能基因组学的应用价值和潜力,扩展了在多种细胞和筛选多种发育相关通路中的应用。但是小鼠胚胎干细胞对培养条件的要求较高,此为该方法的局限性。
使用KBM7细胞的功能基因组学具有如下局限性:(1)这些细胞不是完全的单倍体;(2)使用基因诱捕逆转录病毒介导的随机突变的方法产生单倍体细胞突变株,无法完全覆盖全基因组[62]。因此,需要靶向且针对特定基因的方法,例如RNA干扰和由核酸介导的基因编辑,对基因组进行系统性的敲除或者敲降。
1.2.4 使用RNA干扰(RNA interference, RNAi)的功能基因组学
RNA干扰是一项在众多真核生物中比较保守的生物过程。通过与一种短的20 bp左右的序列特异地干扰RNA结合,进而导致了对转录产物的切割和降解[63]。通过向细胞转染体外合成的siRNA、或者siRNA的前体(short-hairpin RNA, shRNA)以及双链RNA (double stranded RNA),来抑制基因的表达。转染方式包括脂质体介导的转染、电转和病毒介导的转染(慢病毒和逆转录病毒)[41]。RNAi已经被广泛应用于功能基因组学筛选后的单基因功能验证。通过抑制特定基因的表达,检测表型的变化[49]。
RNAi技术也可以应用于功能基因组学筛选,其筛选方式包括混合筛选和阵列筛选[64]。混合筛选是指在细胞内转染siRNA或者shRNA库,实现基因组水平的基因表达抑制,然后进行化学品暴露,使用高通量测序或者流式细胞仪分选的方式,对细胞的表型进行检测,细胞的表型既可以是细胞活性,也可以是关联特定毒性终点的荧光。阵列筛选是指将细胞接种在不同的孔中,在不同的孔里面进行单独的转染、化学品暴露和表型的检测,包括细胞活性检测和荧光检测。例如:可以使用高内涵细胞筛选的方法对已经标记过多个荧光标签的细胞进行表型筛选[65]。目前,较为成熟的RNA干扰质粒库,例如RNAi Consortium Lentiviral Library,可以靶向人类基因组17 200个基因,且每个质粒均含有一段标签序列,已经被广泛用于RNAi的功能基因组筛选中[42]。通过采用混合筛选的方法,采用慢病毒转染,将质粒转染到细胞中,构建全基因组水平表达抑制的细胞库。在环境化学品的暴露下,特定基因的表达抑制会赋予细胞抗性或者敏感性,导致相应的细胞在细胞库中的丰度发生变化。通过流式细胞仪筛选以及高通量测序的方法,获取不同标签序列的丰度信息,通过比较处理组与对照组的丰度,识别介导对化学品暴露抗性和敏感性的基因。RNAi功能基因组学筛选使用细胞系或者经过与特定表型关联的改造后的细胞,已经被广泛地应用于化学品细胞毒性的抗性(例如环境毒害重金属[66-67])、特定的生物学过程以及病原物的响应相关的研究[68-71]。
和其他功能基因组学筛选方法一样,RNAi筛选的局限性主要体现在:(1) RNAi存在显著的脱靶效应[72],部分siRNA会结合到与其只有部分序列匹配的转录产物上,导致基因表达抑制效应的特异性下降[43]。(2) RNAi存在对基因表达的抑制效率参差不齐的情况。首先,RNAi不能完全抑制基因的表达,有些基因的绝大多数转录产物即使被抑制,但依然会有显著的蛋白表达。其次,有些siRNA对转录产物的抑制效率较低,这就需要对一个基因设计更多的siRNA来保证抑制效率[73]。
2 CRISPR功能基因组学(CRISPR functional genomics)
2.1 CRISPR功能基因组学的原理和特点2.1.1 CRISPR-Cas9基因编辑技术介绍
CRISPR-Cas9技术为在各种细胞类型中进行基因组编辑提供了一种有效的方法[28, 74]。与RNAi相比,这种RNA引导的基因编辑技术可以在基因组中产生永久性突变,导致基因功能丧失或获得[74]。CRISPR-Cas9是一种经济、快速、高效且特异性强的基因编辑方法。Cas9蛋白由一个单一的引导RNA(single guide RNA, sgRNA)引导并在特定的基因组位点诱导双链DNA断裂。切割位点的识别和靶向特异性由一个超过20 bp的与目标DNA序列匹配的sgRNA和一个与之相邻的短核苷酸序列(一个序列为NGG的三核苷酸序列,其中N是任意核苷酸)决定[75]。DNA双链断裂(double-strand DNA breaks, DSB)通过非同源末端连接(non-homologous end joining, NHEJ) DNA修复途径或同源定向修复途径(homology directed repair, HDR)修复[20]。NHEJ修复在DSB位点产生插入/缺失(indels),导致转译框位移或提前终止密码子,导致基因敲除。HDR途径将修复模板(供体DNA)并入DSB,将特定核苷酸变化引入靶基因(图3)。在线性同源修复供体存在的情况下,通过将2个sgRNAs与Cas9酶结合可有效引发高达10 kb的DNA的缺失[44]。
通过对寡核苷酸的引导序列sgRNA的大规模合成,可实现在全基因组水平上对基因功能的探究[76]。与shRNA库仅介导基因敲降不同,sgRNA库可与Cas9核酸酶结合用于产生基因敲除突变的细胞库。Sander和Joung[77]运用电穿孔法、核裂解和脂质体转染,在哺乳动物细胞中实现瞬时表达质粒DNA中的Cas9和sgRNAs,而慢病毒载体可用于人类和小鼠细胞中持续性表达Cas9和sgRNA。研究人员可以根据细胞的数量和细胞系的类型,从而选择单一或双载体用于转导Cas9与sgRNA。在双载体系统中,首先进行的是细胞Cas9的初转导,然后筛选出阳性克隆体进行扩增,随后进行sgRNA的转导。例如,Sabatini和Lander的团队开发了一种双载体文库,该文库由73 151个sgRNA质粒组成,共靶向7 114个人类基因和100个非靶向对照基因[78]。使用单载体系统,Cas9和sgRNA在一个载体中被转导入细胞。这种系统是由Zhang的团队开发的,使用一个单独的慢病毒载体将Cas9、sgRNA和嘌呤霉素选择性标记物转染进靶细胞[28]。Zhang的团队最初开发了一个人类全基因组CRISPR-Cas9敲除文库(GeCKOv1),包含64 751个特异导向序列,共靶向18 080个人类基因。该团队对GeCKOv1文库进行了改良,改良后的文库含有123 411个sgRNAs,共靶向19 050个基因(GeCKOv2)[79] (图4)。GeCKOv2文库比GeCKOv1文库多靶向约1 000个基因,每个基因均拥有6个靶向的sgRNAs,且均保证了最小的脱靶效应。此外,v2文库里sgRNA通过产生miRNA前体的发夹结构突变来使相应的miRNA产生功能缺失。在全基因组小鼠慢病毒sgRNA文库开发方面,Koike-Yusa等[80]开发了包含87 897个特异sgRNA、共靶向19 150个编码蛋白的基因文库,而Sanjana等[79]开发了130 209个sgRNA、靶向20 611个基因的全基因组小鼠慢病毒sgRNA文库。

图3 非同源末端连接(NHEJ)和同源定向修复途径(HDR)的原理[20]
注:DSB表示DNA双链断裂,sgRNA表示单一的引导RNA。
Fig. 3 The mechanisms of non-homologous end joining (NHEJ) and homology directed repair (HDR)[20]
Note: DSB stands for double-strand DNA breaks; sgRNA stands for single guide RNA.
2.1.2 CRISPR-Cas9功能基因组筛选工作原理和流程
目前广泛使用的针对环境化学品的CRISPR-Cas9功能基因组学筛选均是混合筛选法。以下内容均以混合筛选法展开。
由慢病毒转染介导sgRNA和Cas9进入哺乳动物细胞后,编码sgRNA和Cas9蛋白的核酸会整合到细胞的基因组中,产生sgRNA和Cas9蛋白的稳定表达(图4)。整合到细胞基因组当中的sgRNA序列可以发挥标签序列的作用,进而对被转染进sgRNA的细胞进行特异性的标记。由于基因的功能缺失,使得细胞在暴露于环境化学品的时候,参与介导化学品毒性效应的或者维持细胞稳态的生物学过程会发生改变,进而导致细胞对环境化学品的有害效应的敏感性发生变化,表现为细胞活性或者其他特定表型的不同。基于对这些变化的生物表型的检测(细胞活性或者荧光标记),可以将与特定表型关联的基因功能缺失筛选出来。通过深度测序,可以通过识别每个细胞的基因组中的sgRNA序列来构建基因功能缺失与特定表型的关联[12, 28]。

图4 GeCKOv2的工作原理
Fig. 4 The working mechanisms of the GeCKOv2 library
CRISPR-Cas9功能基因组学筛选工作流程主要包括以下6个环节(图5)。
(1)合成靶向全基因组的基因的sgRNA质粒文库。通过转化大肠杆菌感受态细胞,对质粒文库进行扩增,获取每个sgRNA克隆数超过500的质粒文库[81]。

图5 CRISPR-Cas9功能基因组学筛选工作流程[11]
Fig. 5 The workflow of CRISPR-Cas9 functional
genomics screen [11]
(2)用以转染人类细胞的慢病毒的制备。制备慢病毒需要将sgRNA载体质粒与慢病毒包装质粒共转染进HEK293T细胞(图4),通过超高速离心的方法收集慢病毒颗粒。同时需要测定纯化后的慢病毒的滴度,一般需要超过1×108 IFU·mL-1的滴度[81]。
(3)利用慢病毒将sgRNA和Cas9转染进人类细胞系。因为同时需要考虑覆盖所有的sgRNA,一般对于一个包含100 000个sgRNA的库,在进行慢病毒转染的时候,最少需要1.7×108个细胞。慢病毒转染结束后,需要维持一定剂量的抗生素选择压力,去除掉那些没有转染进质粒的细胞。
(4)通过深度测序,进行细胞库的质控。
(5)进行环境化学品的CRISPR功能基因组筛选。这种筛选分为2种:正向筛选(positive screen)和反向筛选(negative screen)[30]。
(6)通过深度测序,识别sgRNA标签序列,构建基因功能缺失与特定生物表型的关联。
2.2 CRISPR-Cas9功能基因组学在化学品毒性机制研究中的应用
基于CRISPR-Cas9技术开发的功能基因组学筛选可以实现在全基因组水平系统性基因敲除的基础上,以人类细胞和高等哺乳动物细胞为生物载体,开展环境化学品的毒性机制研究。以此方法获取的全基因组水平的分子响应信息可以提供基因与化学品有害效应的直接关联,实现基因功能的表型锚定,进而发掘环境化学品的新的毒性机制,为在有害结局路径(adverse outcome pathway, AOP)框架下的环境化学品毒性预测与风险评估提供分子机制基础。另外,由于CRISPR-Cas9功能基因组学的一个重要的表型检测是细胞活性,即以细胞死亡为毒性终点,这恰恰是某些环境化学品产生有害效应的重要的中间事件。从AOP的角度看,细胞死亡往往是通往有害结局(adverse outcome, AO)的重要关键事件(key events, KE)。目前,以细胞死亡为关键事件的疾病,往往是一些死亡率较高且难以治愈的疾病,例如神经退行性疾病,包括帕金森、老年痴呆等,其重要病因是神经细胞的大量坏死,导致神经系统的功能退化[82]。近年来,这些疾病的发生和环境污染之间的联系受到普遍关注[4, 83]。应用功能基因组学可以发掘环境化学品暴露导致的细胞死亡与基因功能之间的直接关联,因此,CRISPR-Cas9功能基因组学筛选在研究环境化学品暴露导致细胞死亡的分子机制与相应疾病之间的联系上,具有重大优势[30]。
2.2.1 建立基因与化学品有害效应的直接关联
目前,CRISPR-Cas9功能基因组学筛选已经比较广泛用于药物和有毒物质的生物学测试,包括治疗癌症的药物、微生物毒素、有毒重金属、农药与空气污染物的研究。对于药物和微生物毒素的报道,Shen等[20]已做了较为完整的综述。本文只综述CRISPR-Cas9功能基因组筛选在环境化学品的毒性机制的研究。
GeCKOv1质粒库首次被应用于识别化学品细胞毒性关联基因。本课题组的Xia等[12]首次将CRISPR-Cas9功能基因组学筛选应用于环境化学品的毒性机制研究。他们利用GeCKOv1质粒库构建了全基因组水平敲除的人类肝癌的HepG2细胞,通过暴露于3种不同剂量(细胞毒性的IC50、IC20、IC10)的三氯生(triclosan, TCS),通过检测以细胞死亡为毒性终点的表型,识别了一些已知的三氯生毒性机制,同时也发掘了三氯生的潜在毒性机制。排名靠前的2个重要基因FTO和MAP2K3的功能缺失均介导了HepG2细胞对三氯生引发的细胞死亡的抗性。三氯生低剂量反向筛选识别出的基因显著富集了与免疫相关的生物学通路,这与低剂量三氯生暴露下的转录组提供的分子响应信息是一致的。通过联合分析疾病数据库(DisGeNET和CTD),发现了FTO和MAP2K3与乳腺癌和肥胖的关联。而流行病学研究表明乳腺癌和肥胖均与三氯生暴露相关[84-85]。该研究通过CRISPR功能基因组筛选发掘了三氯生的潜在毒性机制,并且提示了与三氯生暴露相关的疾病的遗传风险因素。
基于GeCKOv2质粒库的二次筛选增加CRISPR筛选结果的准确性。Sobh等[86]利用GeCKOv2质粒库构建了全基因组敲除的人类白血病细胞K562,揭示了乙醛的潜在毒性机制。该团队通过将sgRNA质粒文库分成2个亚库,并进行了2次反向筛选,通过第二次的筛选,对第一次的筛选结果进行了验证,该方法是一种利用二次筛选对多个备选基因进行验证的尝试与创新。此外,OVCA2被识别出与细胞对乙醛的细胞毒性相关,OVCA2的基因功能缺失导致细胞对乙醛导致的细胞死亡敏感性增加,同时,OVCA2的缺失导致细胞在乙醛暴露下的DNA加合物积累的增加,提示OVCA2与乙醛介导的遗传毒性有关。此外,该团队还利用K562全基因组敲除的细胞库识别了与三价砷细胞毒性相关的基因[87],其中KEAP1和TXNDC17敲除会显著增强细胞对三价砷细胞毒性的抗性。AQP3、ZNT1和MTF1的功能缺失也可以增强对三价砷的抗性,ABCC1的功能缺失可以增强细胞对三价砷的敏感性。硒代半胱氨酸代谢通路上的基因的功能缺失会显著增强细胞对三价砷的抗性,提示细胞内的硒代谢与三价砷的相互关系会影响砷的细胞毒性。
通过检测细胞荧光识别基因与低剂量化学品有害效应之间的关联。Panganiban等[88]使用CHOP报告基因法为检测终点,通过CRISPR全基因组筛选识别了功能缺失会增加内质网应激介导的细胞凋亡的基因。CRISPR全基因组筛选识别出L3MBTL2、MGA和microRNA-124-3是打分最高的基因。这3个基因的功能缺失会导致细胞对多种通过诱导细胞内质网应激导致细胞凋亡的化学品的敏感性增强,而这些基因的过表达,可以导致敏感性降低。L3MBTL2在未产生内质网应激的细胞中,与CHOP的启动子结合,抑制CHOP的表达,但在发生内质网应激的细胞中,与CHOP启动子分离。此外,miR-124-3直接靶向内质网应激信号通路的关键基因。
目前,CRISPR功能基因组筛选已经通过检测细胞活性或者与特定毒性终点关联的荧光,构建了基因与环境化学品有害效应的直接关联。检测细胞活性的CRISPR筛选可以识别与介导细胞死亡关联的基因,而检测与特定毒性终点关联的荧光的CRISPR筛选可以识别与介导相应细胞毒性关联的基因。前者基于检测细胞活性,因此其识别的基因不受已有毒性机制的约束,可以在最大程度上获取与介导细胞死亡关联的基因。相比之下,检测与特定毒性终点关联的荧光的CRISPR筛选,其检测指标是基于已知毒性机制设置的报告基因,可能导致识别的基因范围受限。但这种筛选方法因为检测细胞荧光,进行筛选所需要的化学品暴露剂量往往比较低,且暴露时间更短,便于进行对低剂量化学品或者基于环境暴露剂量的化学品的毒性机制研究。
2.2.2 识别导致细胞死亡的化学品毒性机制
由于以细胞死亡为毒性终点的表型检测是CRISPR功能基因组学筛选最主要的检测指标,因此,目前大多数CRISPR筛选研究均是识别与细胞死亡关联的分子机制。这类研究主要分为对癌症药物和环境化学品暴露导致细胞死亡的功能基因组学筛选。而对于环境化学品的毒性机制研究,细胞死亡是相应化学品所导致有害效应的重要中间事件,这类化学品往往通过诱导氧化应激、线粒体损伤等而导致细胞死亡[89-90]。而与这类化学品暴露关联的疾病的重要病因就包括靶细胞的死亡,因此,这类化学品导致细胞死亡的分子机制往往是导致疾病的重要机制或者是可以开发成治疗靶标的生物标志物[91]。
CRISPR功能基因组学筛选可以识别导致细胞死亡的分子启动事件。Reczek等[30]使用CRISPR功能基因组学筛选识别了与百草枯暴露导致细胞死亡关联的分子机制。百草枯可以通过引发细胞氧化应激导致细胞死亡,而百草枯诱导细胞氧化应激的根源是不清楚的。该团队使用覆盖所有与代谢有关的基因的CRISPR-Cas9功能基因组学筛选,通过进行正向筛选(百草枯暴露浓度模拟百草枯急性暴露的人体内暴露剂量),识别了POR、ATP7A和SLC45A4是百草枯暴露导致细胞死亡所必需的3个基因。此外,POR是百草枯诱导细胞产生氧化应激的来源。由于百草枯是诱导帕金森疾病的重要的环境污染物之一[92],帕金森病是一种神经退行性疾病,其主要病因为多巴胺能神经元的损伤与坏死。这次报道中发现的POR基因所编码的蛋白位于线粒体呼吸链,而线粒体的功能紊乱与帕金森疾病的病因有重要联系[93],此研究的成果对于开发治疗帕金森疾病的药物具有重要参考价值。
多时间点的CRISPR筛选完善了导致细胞死亡的分子机制。Shortt等[23]使用全基因组敲除的人类肝细胞系HUH7细胞开展了对乙酰氨基酚(acetaminophen, APAP)导致肝损伤机制的研究。APAP是一种常见的感冒药,用于镇痛消炎,但其也是一种可以导致肝损伤的化学品。该团队通过开展多时间点的CRISPR功能基因组学筛选,发现了众多与介导肝细胞死亡和维持肝细胞活性的基因,例如BMPR1A和FCGR3A,这些基因富集了钙离子信号通路、TNF信号通路和脂肪酸代谢通路。这些基因和生物学通路的识别为解析APAP导致的肝损伤分子机制提供了依据,同时也为肝损伤的治疗提供了潜在治疗靶标。
3 展望(Perspectives)
人类对化学品暴露的有害效应存在易感性差异,而不同个体间的遗传变异是导致这种易感性差异的重要原因。目前,以GWAS为代表的分子流行病学研究依赖关联性统计分析来获取遗传变异与疾病表型的关联,但难以为这种关联提供机制证据。同时,基于统计分析筛选出的备选基因往往数量较多,GWAS研究无法提供优先性评估。而CRISPR-Cas9功能基因组学可以提供基于遗传变异视角的化学品的毒性机制[30],通过联合CRISPR筛选与分子流行病学分析,可以开展针对化学品有害效应的易感性机制研究,进而为有效识别易感人群提供机制证据,为开展环境化学品的精准风险评估奠定基础(图6)。
当前的CRISPR-Cas9功能基因组学依赖于CRISPR-Cas9基因编辑技术进行对基因的编辑或敲除。目前,针对于其在化学品毒性机制研究的应用,CRISPR-Cas9技术主要的局限性表现在脱靶效应[94]和物种的限制。脱靶效应可以通过生物信息学的计算,对sgRNA序列的设计进行优化,进而降低脱靶概率[94-95]。同时,可以通过技术改进,例如:可以事先将细胞系稳定表达优化设计的Cas9蛋白,然后将只含有sgRNA的质粒转染进细胞中,这样既增加了转染效率,也可以降低脱靶概率[96]。另一方面,当前的CRISPR筛选主要应用于哺乳动物细胞,在其他物种中的应用较少。而这种限制不是来源于CRISPR技术本身,而是因为对其他物种的基因组的解析程度不足。目前,已有少量在非哺乳动物细胞中进行CRISPR功能基因组筛选的报道,例如家蚕[97]。随着其他物种的基因组的不断解析,可以实现CRISPR功能基因组筛选在更多生态物种中的应用,进而为该技术在生态毒理学中的广泛应用奠定基础。
分子流行病学利用基因组学测序,通过关联性统计分析,建立遗传变异与个体疾病表型之间的关联。传统的分子流行病学方法主要是全基因组关联性研究(GWAS)。依赖于组学技术和高通量测序技术的进步,基于人群的环境化学品暴露的转录组学、代谢组学和表观遗传组学已经逐渐被应用于环境化学品的毒性机制研究。这些人群组学方法的最大优势在于检测人体样本,可以直接基于个体疾病表型进行分组和分析[98-99]。虽然这些方法获取的分子信息具有一定程度的表型锚定,但由于疾病的复杂性,以上组学方法获取的分子扰动信息与疾病的关系不能准确确定,例如:获取的生物学通路扰动的信息,不能准确确定其是导致疾病的原因还是疾病本身所产生的症状效应,因此,这种类型的表型锚定具有不确定性。此外,由于往往不能及时获取化学品暴露后的人体样品,所以基于外暴露浓度的剂量-关系效应往往具有不确定性,此时,必须使用对应的内暴露剂量,这又提高了研究的成本与难度。

图6 联合CRISPR筛选和分子流行病学分析,开展化学品有害效应的易感性机制研究
Fig. 6 To uncover the biological mechanisms underlying the susceptibility to adverse effects of
chemicals exposure by combining using of CRISPR screen and molecular epidemiology analysis
因此,对于化学品有害效应的易感性机制研究,需要有一种低成本且高效的替代测试方法为分子流行病学研究所识别的遗传风险因素(genetic risk factor)提供机制证据和优先性评估。这就要求这种替代测试方法具备以下功能[20]:(1) 可以获取全基因组水平的分子响应信息;(2) 可以建立化学品有害效应与基因功能的直接关联;(3) 可以提供基于遗传变异的分子事件视角;(4) 可以实现高通量的化学品测试。而CRISPR-Cas9功能基因组学筛选就是一个可以同时满足以上要求的替代测试方法。由CRISPR-Cas9介导的对基因的编辑,可以实现对基因特定片段的特异性敲除,进而产生相应的基因功能缺失,这种基因功能缺失可以导致细胞的某些代谢和功能的变化,进而导致细胞对化学品有害效应的易感性变化。这个过程与由遗传变异介导的化学品有害效应的易感性变化类似。遗传变异与基因编辑所产生的效应均体现为基因功能的变化,而化学品有害效应的易感性最终是归因于基因功能的变化,因此CRISPR-Cas9功能基因组学可以提供基于遗传变异的分子事件视角。
未来可将CRISPR-Cas9功能基因组学与分子流行病学分析联合运用,来探究化学品有害效应易感性机制[13, 20]。Shortt等[23]使用全基因组敲除的人类肝细胞系HUH7细胞开展了对乙酰氨基酚(APAP)导致肝损伤机制的研究。通过将CRISPR筛选获取的基因信息与GEO数据库中人群在APAP暴露下的转录组数据和肝损伤个体的转录组数据进行比较分析,发现了一些相同的基因。此外,通过对比疾病数据库,也发现了一些CRISPR筛选与人群肝损伤相关基因的相同基因。这些相同的基因可以作为APAP导致肝损伤的易感性机制的潜在研究对象。但该研究对GEO数据的分析过于简单,只是简单地将差异表达基因与CRISPR筛选识别的基因进行维恩图分析,寻找其中的相同基因,并未对GEO数据进行深入的再分析。此外,针对与APAP暴露导致的肝损伤相关的遗传变异,该研究尚无对这些单核苷酸多态性(single nucleotide polymorphisms, SNPs)在人群中的基因频率和药物导致的肝损伤的患病率或者其他相关表型数据进行关联性分析。因此,该研究后续仍需要进行更多深入的分子机制的验证和相关分子流行病学数据的深入分析,以在更深层次揭示对APAP导致的肝损伤的易感性机制。
总之,不同的功能基因组学具有相应的优势和局限性,可以根据具体的研究目的和化学品特性来选择合适的测试方法开展环境化学品的毒性机制研究。同时,在化学品有害效应的易感性机制研究方面,则需要联合其他的组学方法以及分子流行病学分析来开展相关研究。
通讯作者简介:张效伟(1978—),男,博士,教授,博士生导师,主要研究方向为生态毒理学和健康风险评估。
[1] United Nations Environment Programme (UNEP). Global chemicals outlook: Towards sound management of chemicals [R]. Nairobi, Kenya: UNEP, 2013
[2] Rappaport S M, Smith M T. Environment and disease risks [J]. Science, 2010, 330(6003): 460-461
[3] Gruber K. Cleaning up pollutants to protect future health [J]. Nature, 2018, 555(7695): S20-S22
[4] Cicero C E, Mostile G, Vasta R, et al. Metals and neurodegenerative diseases. A systematic review [J]. Environmental Research, 2017, 159: 82-94
[5] Backhaus T, Faust M. Predictive environmental risk assessment of chemical mixtures: A conceptual framework [J]. Environmental Science & Technology, 2012, 46(5): 2564-2573
[6] Syberg K, Hansen S F. Environmental risk assessment of chemicals and nanomaterials: The best foundation for regulatory decision-making? [J]. Science of the Total Environment, 2016, 541: 784-794
[7] Farmahin R, Williams A, Kuo B, et al. Recommended approaches in the application of toxicogenomics to derive points of departure for chemical risk assessment [J]. Archives of Toxicology, 2017, 91(5): 2045-2065
[8] Moffat I, Chepelev N, Labib S, et al. Comparison of toxicogenomics and traditional approaches to inform mode of action and points of departure in human health risk assessment of benzo[a]pyrene in drinking water [J]. Critical Reviews in Toxicology, 2015, 45(1): 1-43
[9] Waters M D, Fostel J M. Toxicogenomics and systems toxicology: Aims and prospects [J]. Nature Reviews Genetics, 2004, 5(12): 936-948
[10] Aardema M J, MacGregor J T. Toxicology and genetic toxicology in the new era of “toxicogenomics”: Impact of “-omics” technologies [J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2002, 499(1): 13-25
[11] Zhang X W, Xia P, Wang P P, et al. Omics advances in ecotoxicology [J]. Environmental Science & Technology, 2018, 52(7): 3842-3851
[12] Xia P, Zhang X W, Xie Y W, et al. Functional toxicogenomic assessment of triclosan in human HepG2 cells using genome-wide CRISPR-Cas9 screening [J]. Environmental Science & Technology, 2016, 50(19): 10682-10692
[13] Lujan H, Romer E, Salisbury R, et al. Determining the biological mechanisms of action for environmental exposures: Applying CRISPR/Cas9 to toxicological assessments [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2020, 175(1): 5-18
[14] de Jong K, Vonk J M, Imboden M, et al. Genes and pathways underlying susceptibility to impaired lung function in the context of environmental tobacco smoke exposure [J]. Respiratory Research, 2017, 18(1): 142
[15] Smith B M, Traboulsi H, Austin J H M, et al. Human airway branch variation and chronic obstructive pulmonary disease [J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(5): E974-E981
[16] Khreis H, Kelly C, Tate J, et al. Exposure to traffic-related air pollution and risk of development of childhood asthma: A systematic review and meta-analysis [J]. Environment International, 2017, 100: 1-31
[17] Thomas D. Gene: Environment-wide association studies: Emerging approaches [J]. Nature Reviews Genetics, 2010, 11(4): 259-272
[18] Sepand M R, Aliomrani M, Hasani-Nourian Y, et al. Mechanisms and pathogenesis underlying environmental chemical-induced necroptosis [J]. Environmental Science and Pollution Research International, 2020, 27(30): 37488-37501
[19] Rodgers K M, Udesky J O, Rudel R A, et al. Environmental chemicals and breast cancer: An updated review of epidemiological literature informed by biological mechanisms [J]. Environmental Research, 2018, 160: 152-182
[20] Shen H, McHale C M, Smith M T, et al. Functional genomic screening approaches in mechanistic toxicology and potential future applications of CRISPR-Cas9 [J]. Mutation Research Reviews in Mutation Research, 2015, 764: 31-42
[21] Jo W J, Loguinov A, Wintz H, et al. Comparative functional genomic analysis identifies distinct and overlapping sets of genes required for resistance to monomethylarsonous acid (MMAⅢ) and arsenite (AsⅢ) in yeast [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2009, 111(2): 424-436
[22] Zhou Y X, Zhu S Y, Cai C Z, et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells [J]. Nature, 2014, 509(7501): 487-491
[23] Shortt K, Heruth D P, Zhang N N, et al. Identification of novel regulatory genes in APAP induced hepatocyte toxicity by a genome-wide CRISPR-Cas9 screen [J]. Scientific Reports, 2019, 9(1): 1396
[24] Guan M, Xia P, Tian M M, et al. Molecular fingerprints of conazoles via functional genomic profiling of Saccharomyces cerevisiae [J]. Toxicology in Vitro: An International Journal Published in Association with BIBRA, 2020, 69: 104998
[25] Chow R D, Chen S D. Cancer CRISPR screens in vivo [J]. Trends in Cancer, 2018, 4(5): 349-358
[26] Shan G. RNA interference as a gene knockdown technique [J]. The International Journal of Biochemistry & Cell Biology, 2010, 42(8): 1243-1251
[27] Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: Progress, implications and challenges [J]. Human Molecular Genetics, 2014, 23(R1): R40-R46
[28] Shalem O, Sanjana N E, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells [J]. Science, 2014, 343(6166): 84-87
[29] Cenik C, Cenik E S, Byeon G W, et al. Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans [J]. Genome Research, 2015, 25(11): 1610-1621
[30] Reczek C R, Birsoy K, Kong H, et al. A CRISPR screen identifies a pathway required for paraquat-induced cell death [J]. Nature Chemical Biology, 2017, 13(12): 1274-1279
[31] Bassett D E, Boguski M S, Hieter P. Yeast genes and human disease [J]. Nature, 1996, 379(6566): 589-590
[32] Scherens B, Goffeau A. The uses of genome-wide yeast mutant collections [J]. Genome Biology, 2004, 5(7): 229
[33] Steinmetz L M, Scharfe C, Deutschbauer A M, et al. Systematic screen for human disease genes in yeast [J]. Nature Genetics, 2002, 31(4): 400-404
[34] Yamazoe M, Sonoda E, Hochegger H, et al. Reverse genetic studies of the DNA damage response in the chicken B lymphocyte line DT40 [J]. DNA Repair, 2004, 3(8-9): 1175-1185
[35] Ji K, Kogame T, Choi K, et al. A novel approach using DNA-repair-deficient chicken DT40 cell lines for screening and characterizing the genotoxicity of environmental contaminants [J]. Environmental Health Perspectives, 2009, 117(11): 1737-1744
[36] Ooka M, Takazawa H, Takeda S, et al. Cytotoxic and genotoxic profiles of benzo[a]pyrene and N-nitrosodimethylamine demonstrated using DNA repair deficient DT40 cells with metabolic activation [J]. Chemosphere, 2016, 144: 1901-1907
[37] Kotecki M, Reddy P S, Cochran B H. Isolation and characterization of a near-haploid human cell line [J]. Experimental Cell Research, 1999, 252(2): 273-280
[38] Pettitt S J, Rehman F L, Bajrami I, et al. A genetic screen using the PiggyBac transposon in haploid cells identifies Parp1 as a mediator of olaparib toxicity [J]. PLoS One, 2013, 8(4): e61520
[39] Duncan L M, Timms R T, Zavodszky E, et al. Fluorescence-based phenotypic selection allows forward genetic screens in haploid human cells [J]. PLoS One, 2012, 7(6): e39651
[40] Leeb M, Wutz A. Derivation of haploid embryonic stem cells from mouse embryos [J]. Nature, 2011, 479(7371): 131-134
[41] Wolters N M, MacKeigan J P. From sequence to function: Using RNAi to elucidate mechanisms of human disease [J]. Cell Death & Differentiation, 2008, 15(5): 809-819
[42] Root D E, Hacohen N, Hahn W C, et al. Genome-scale loss-of-function screening with a lentiviral RNAi library [J]. Nature Methods, 2006, 3(9): 715-719
[43] Mohr S E, Smith J A, Shamu C E, et al. RNAi screening comes of age: Improved techniques and complementary approaches [J]. Nature Reviews Molecular Cell Biology, 2014, 15(9): 591-600
[44] Zheng Q P, Cai X H, Tan M H, et al. Precise gene deletion and replacement using the CRISPR/Cas9 system in human cells [J]. BioTechniques, 2014, 57(3): 115-124
[45] Costanzo M C, Hogan J D, Cusick M E, et al. The yeast proteome database (YPD) and Caenorhabditis elegans proteome database (WormPD): Comprehensive resources for the organization and comparison of model organism protein information [J]. Nucleic Acids Research, 2000, 28(1): 73-76
[46] Parsons A B, Geyer R, Hughes T R, et al. Yeast genomics and proteomics in drug discovery and target validation [J]. Progress in Cell Cycle Research, 2003, 5: 159-166
[47] O’Connor S T, Lan J Q, North M, et al. Genome-wide functional and stress response profiling reveals toxic mechanism and genes required for tolerance to benzo[a]pyrene in S. cerevisiae [J]. Frontiers in Genetics, 2012, 3: 316
[48] de Graaf B, Clore A, McCullough A K. Cellular pathways for DNA repair and damage tolerance of formaldehyde-induced DNA-protein crosslinks [J]. DNA Repair, 2009, 8(10): 1207-1214
[49] Ren X F, Lim S, Ji Z Y, et al. Comparison of proliferation and genomic instability responses to WRN silencing in hematopoietic HL60 and TK6 cells [J]. PLoS One, 2011, 6(1): e14546
[50] Ren X F, Aleshin M, Jo W J, et al. Involvement of N-6 adenine-specific DNA methyltransferase 1 (N6AMT1) in arsenic biomethylation and its role in arsenic-induced toxicity [J]. Environmental Health Perspectives, 2011, 119(6): 771-777
[51] Buerstedde J M, Takeda S. Increased ratio of targeted to random integration after transfection of chicken B cell lines [J]. Cell, 1991, 67(1): 179-188
[52] Evans T J, Yamamoto K N, Hirota K, et al. Mutant cells defective in DNA repair pathways provide a sensitive high-throughput assay for genotoxicity [J]. DNA Repair, 2010, 9(12): 1292-1298
[53] Mizutani A, Okada T, Shibutani S, et al. Extensive chromosomal breaks are induced by tamoxifen and estrogen in DNA repair-deficient cells [J]. Cancer Research, 2004, 64(9): 3144-3147
[54] Ji K, Choi K, Giesy J P, et al. Genotoxicity of several polybrominated diphenyl ethers (PBDEs) and hydroxylated PBDEs, and their mechanisms of toxicity [J]. Environmental Science & Technology, 2011, 45(11): 5003-5008
[55] Andersson B S, Beran M, Pathak S, et al. pH-positive chronic myeloid leukemia with near-haploid conversion in vivo and establishment of a continuously growing cell line with similar cytogenetic pattern [J]. Cancer Genetics and Cytogenetics, 1987, 24(2): 335-343
[56] Carette J E, Raaben M, Wong A C, et al. Ebola virus entry requires the cholesterol transporter Niemann-Pick C1 [J]. Nature, 2011, 477(7364): 340-343
[57] Birsoy K, Wang T, Possemato R, et al. MCT1-mediated transport of a toxic molecule is an effective strategy for targeting glycolytic tumors [J]. Nature Genetics, 2013, 45(1): 104-108
[58] Shen H, McHale C M, Haider S I, et al. Identification of genes that modulate susceptibility to formaldehyde and imatinib by functional genomic screening in human haploid KBM7 cells [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2016, 154(1): 194
[59] Lee C C, Carette J E, Brummelkamp T R, et al. A reporter screen in a human haploid cell line identifies CYLD as a constitutive inhibitor of NF-κB [J]. PLoS One, 2013, 8(7): e70339
[60] Elling U, Taubenschmid J, Wirnsberger G, et al. Forward and reverse genetics through derivation of haploid mouse embryonic stem cells [J]. Cell Stem Cell, 2011, 9(6): 563-574
[61] Li W, Shuai L, Wan H F, et al. Androgenetic haploid embryonic stem cells produce live transgenic mice [J]. Nature, 2012, 490(7420): 407-411
[62] Carette J E, Pruszak J, Varadarajan M, et al. Generation of iPSCs from cultured human malignant cells [J]. Blood, 2010, 115(20): 4039-4042
[63] Rana T M. Illuminating the silence: Understanding the structure and function of small RNAs [J]. Nature Reviews Molecular Cell Biology, 2007, 8(1): 23-36
[64] Willingham A T, Deveraux Q L, Hampton G M, et al. RNAi and HTS: Exploring cancer by systematic loss-of-function [J]. Oncogene, 2004, 23(51): 8392-8400
[65] Fennell M, Xiang Q, Hwang A, et al. Impact of RNA-guided technologies for target identification and deconvolution [J]. Journal of Biomolecular Screening, 2014, 19(10): 1327-1337
[66] Park H R, Oh R, Wagner P, et al. New Insights into Cellular Stress Responses to Environmental Metal Toxicants [M]// Galluzzi L. Ed. International Review of Cell and Molecular Biology. Elsevier B.V., 2017, 331: 55-82
[67] Oh R S, Pan W C, Yalcin A, et al. Functional RNA interference (RNAi) screen identifies system A neutral amino acid transporter 2 (SNAT2) as a mediator of arsenic-induced endoplasmic reticulum stress [J]. The Journal of Biological Chemistry, 2012, 287(8): 6025-6034
[68] Berns K, Bernards R. Understanding resistance to targeted cancer drugs through loss of function genetic screens [J]. Drug Resistance Updates, 2012, 15(5-6): 268-275
[69] Swanton C, Marani M, Pardo O, et al. Regulators of mitotic arrest and ceramide metabolism are determinants of sensitivity to paclitaxel and other chemotherapeutic drugs [J]. Cancer Cell, 2007, 11(6): 498-512
[70] Singel S M, Cornelius C, Batten K, et al. A targeted RNAi screen of the breast cancer genome identifies KIF14 and TLN1 as genes that modulate docetaxel chemosensitivity in triple-negative breast cancer [J]. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research, 2013, 19(8): 2061-2070
[71] Schmidt E E, Pelz O, Buhlmann S, et al. GenomeRNAi: A database for cell-based and in vivo RNAi phenotypes, 2013 update [J]. Nucleic Acids Research, 2013, 41(Database issue): D1021-D1026
[72] Ngo V N, Davis R E, Lamy L, et al. A loss-of-function RNA interference screen for molecular targets in cancer [J]. Nature, 2006, 441(7089): 106-110
[73] Bassik M C, Lebbink R J, Churchman L S, et al. Rapid creation and quantitative monitoring of high coverage shRNA libraries [J]. Nature Methods, 2009, 6(6): 443-445
[74] Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems [J]. Science, 2013, 339(6121): 819-823
[75] Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells [J]. eLife, 2013, 2: e00471
[76] Blanchard A P, Hood L. Sequence to array: Probing the genome’s secrets [J]. Nature Biotechnology, 1996, 14(13): 1649
[77] Sander J D, Joung J K. CRISPR-Cas systems for editing, regulating and targeting genomes [J]. Nature Biotechnology, 2014, 32(4): 347-355
[78] Wang T, Wei J J, Sabatini D M, et al. Genetic screens in human cells using the CRISPR-Cas9 system [J]. Science, 2014, 343(6166): 80-84
[79] Sanjana N E, Shalem O, Zhang F. Improved vectors and genome-wide libraries for CRISPR screening [J]. Nature Methods, 2014, 11(8): 783-784
[80] Koike-Yusa H, Li Y L, Tan E P, et al. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library [J]. Nature Biotechnology, 2014, 32(3): 267-273
[81] Joung J, Konermann S, Gootenberg J S, et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening [J]. Nature Protocols, 2017, 12(4): 828-863
[82] Bredesen D E, Rao R V, Mehlen P. Cell death in the nervous system [J]. Nature, 2006, 443(7113): 796-802
[83] Baltazar M T, Dinis-Oliveira R J, de Lourdes Bastos M, et al. Pesticides exposure as etiological factors of Parkinson’s Disease and other neurodegenerative diseases: A mechanistic approach [J]. Toxicology Letters, 2014, 230(2): 85-103
[84] Dinwiddie M T, Terry P D, Chen J G. Recent evidence regarding triclosan and cancer risk [J]. International Journal of Environmental Research and Public Health, 2014, 11(2): 2209-2217
[85] Kalloo G, Calafat A M, Chen A M, et al. Early life triclosan exposure and child adiposity at 8 Years of age: A prospective cohort study [J]. Environmental Health: A Global Access Science Source, 2018, 17(1): 24
[86] Sobh A, Loguinov A, Stornetta A, et al. Genome-wide CRISPR screening identifies the tumor suppressor candidate OVCA2 as a determinant of tolerance to acetaldehyde [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2019, 169(1): 235-245
[87] Sobh A, Loguinov A, Yazici G N, et al. Functional profiling identifies determinants of arsenic trioxide cellular toxicity [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2019, 169(1): 108-121
[88] Panganiban R A, Park H R, Sun M Y, et al. Genome-wide CRISPR screen identifies suppressors of endoplasmic reticulum stress-induced apoptosis [J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(27): 13384-13393
[89] Liao Y J, Hao Y M, Chen H, et al. Mitochondrial calcium uniporter protein MCU is involved in oxidative stress-induced cell death [J]. Protein & Cell, 2015, 6(6): 434-442
[90] Gogvadze V, Orrenius S, Zhivotovsky B. Analysis of mitochondrial dysfunction during cell death [J]. Methods in Molecular Biology, 2015, 1264: 385-393
[91] Braun R J. Mitochondrion-mediated cell death: Dissecting yeast apoptosis for a better understanding of neurodegeneration [J]. Frontiers in Oncology, 2012, 2: 182
[92] Tangamornsuksan W, Lohitnavy O, Sruamsiri R, et al. Paraquat exposure and Parkinson’s Disease: A systematic review and meta-analysis [J]. Archives of Environmental & Occupational Health, 2019, 74(5): 225-238
[93] Orth M. Mitochondrial involvement in Parkinson’s Disease [J]. Neurochemistry International, 2002, 40(6): 533-541
[94] Coelho M A, de Braekeleer E, Firth M, et al. CRISPR GUARD protects off-target sites from Cas9 nuclease activity using short guide RNAs [J]. Nature Communications, 2020, 11(1): 4132
[95] Concordet J P, Haeussler M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens [J]. Nucleic Acids Research, 2018, 46(W1): W242-W245
[96] Liu M S, Gong S Z, Yu H H, et al. Engineered CRISPR/Cas9 enzymes improve discrimination by slowing DNA cleavage to allow release of off-target DNA [J]. Nature Communications, 2020, 11(1): 3576
[97] Liu Y, Chang J S, Yang C F, et al. Genome-wide CRISPR-Cas9 screening in Bombyx mori reveals the toxicological mechanisms of environmental pollutants, fluoride and cadmium [J]. Journal of Hazardous Materials, 2021, 410: 124666
[98] Chen Y C, Tsai Y H, Wang C C, et al. Epigenome-wide association study on asthma and chronic obstructive pulmonary disease overlap reveals aberrant DNA methylations related to clinical phenotypes [J]. Scientific Reports, 2021, 11: 5022
[99] Ren X W, Wen W, Fan X Y, et al. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas [J]. Cell, 2021, 184(23): 5838