细菌耐药已经成为人类健康的一大威胁,具有抗生素抗性的细菌引起的感染每年导致全球约70万人死亡,估计到2050年,每年有超过1 000万人死亡[1]。耐药问题之所以发展得如此迅速,主要原因之一是抗生素抗性基因(antibiotics resistance genes, ARGs)可以通过水平转移的方式进行传播。水平转移可以发生在任何环境中,特别是当细菌密度较高时,例如,在土壤、污水处理厂以及人和动物的肠道微生物群中[2-7]。水平转移方式包括转化、转导和接合转移[8],其中接合转移是水平转移的主要方式[9]。
自20世纪80年代以来,由于粪肠球菌的耐药性不断增强,粪肠球菌菌株越来越多地与医院获得性感染有关[10-12]。粪肠球菌在环境中分布也十分广泛,养殖场废水、生活污水、自然水源、土壤、饲料和粪便中都有分布[13],其对多种抗菌药物具有耐药性,也容易被诱导产生获得性耐药[14]。粪肠球菌间的ARGs主要通过信息素诱导的接合质粒进行转移传播,典型的信息素响应质粒有pCF10、pAD1、pAM373和pMG2200,其中pCF10质粒是最有代表性的一种[15]。其转移机制是由受体菌分泌一种cCF10信息素,供体菌识别到信息素后开始启动质粒的接合转移过程[16]。pCF10质粒的转移频率可以达到10-2~10-1[17],主要发生在可引起严重院内感染的肠球菌间,能够使肠球菌获得对被称为最后一种抗生素的万古霉素的耐药性[15]。粪肠球菌耐药性的传播不仅给临床中细菌感染的治疗带来巨大挑战,也严重影响到生态安全。经过抗生素治疗的动物的污水和粪便污染水源,可导致ARGs传播,可以传播到水产生物,并通过食物链逐级传递,最终影响整个生态环境[18]。
水环境是ARGs的巨大储存库[19],在水环境中还会有一些纳米材料[20]。已有研究表明纳米材料可以促进ARGs的传播[21]。一些新兴纳米材料的生物效应和生态安全性有待深入研究。纳米二硫化钼(molybdenum disulfide, MoS2)是一种新兴的过渡金属二硫化物,由1层钼和2层硫组成,是类似石墨烯的二维材料。因其具有高的比表面积、优异的光热转换性能、易于功能化等优点,已在催化、传感、载药和疾病治疗等领域得到广泛应用[22]。随着纳米MoS2应用的增多,其在环境中的残留水平可能增加,与其他纳米粒子一样,纳米MoS2的积累对生态环境和人类健康构成了威胁。本文研究了纳米MoS2对粪肠球菌中信息素诱导的耐药质粒接合转移的影响,并初步研究了其机制。为研究纳米MoS2的生物效应及其安全应用提供理论指导。
1 材料与方法(Materials and methods)
1.1 菌株、质粒及培养条件
本实验中用到的粪肠球菌OG1RF[23](ATCC 47077,NCBI:txid474186)购自美国典型培养物保藏中心(ATCC),信息素诱导质粒pCF10由美国明尼苏达大学Gary Dunny教授惠赠。受体菌为粪肠球菌OG1RS,由粪肠球菌OG1RF经本实验室诱导获得链霉素抗性,并命名为OG1RS;供体菌为粪肠球菌OG1RF(pCF10),其携带信息素调控质粒pCF10[24],该质粒编码四环素抗性基因。供体菌、受体菌均利用BHI液体培养基培养,并添加相应的抗生素(受体菌:3 000 μg·mL-1链霉素(S8290,Solarbio),供体菌:10 μg·mL-1四环素(A500731,Sangon Biotech))。菌液在37 ℃恒温恒湿培养箱中培养10 h后,将菌液按1∶10的比例转接至新鲜的BHI培养基中,置于37 ℃恒温恒湿培养箱中4~5 h生长至对数期后备用。
1.2 接合转移实验
菌液在4 ℃条件下6 000 r·min-1转速离心5 min(Eppendorf Centrifuge 5804R,Eppendorf,德国),弃上清后用含2 mmol·L-1乙二胺四乙酸(ethylene diamine tetraacetic acid, EDTA)的磷酸盐缓冲液(phosphate buffer solution, PBS)重悬菌液并洗涤3次,最后用LB液体培养基重悬菌液并调整菌液浓度至1.5×108 CFU·mL-1。供、受体菌按1∶1的比例混合后加入纳米MoS2悬液(NM000310,Solarbio),使其终浓度为1、5、10、25、50、100和200 mg·L-1,并用振荡器振荡混匀,每组3个重复。将接合菌液放入37 ℃恒温恒湿培养箱培养。2 h后取100 μL菌液利用含2 mmol·L-1 EDTA的PBS溶液梯度稀释,取合适稀释梯度的10 μL菌液分别滴在含有10 μg·mL-1四环素和3 000 μg·mL-1链霉素的双抗BHI琼脂培养基上筛选接合子,含有3 000 μg·mL-1链霉素BHI琼脂培养基上筛选受体菌、含有10 μg·mL-1四环素的BHI琼脂培养基上筛选供体菌,37 ℃倒置培养36 h左右计数形成的单菌落,并计算接合频率(接合频率=接合子数量/受体菌数量)。
1.3 不同因素对接合转移的影响
分别研究不同时间、不同温度和不同pH条件下的接合转移规律。将纳米MoS2加入接合体系中,使终浓度为25 mg·L-1,在培养箱放置不同时刻然后对接合子计数,计算接合频率;将接合体系在不同温度下(16、26、30和37 ℃)接合2 h,计算接合频率;用NaOH溶液和HCl溶液调节接合体系的初始pH,使pH分别为6、7和8,在37 ℃条件下接合2 h,计算接合频率。
1.4 纳米MoS2作用后活性氧水平检测
对数生长期的菌液用PBS洗涤2次,再用液体LB培养基重悬,并稀释至108 CFU·mL-1。装载DCFH-DA探针(CA1410,Solarbio)终浓度为2 μmol·mL-1,37 ℃放置20 min后用PBS洗掉多余探针,加入纳米MoS2使终浓度为25 mg·L-1,作用2 h,用酶标仪(Molecular Devices,SpectraMax M5,美国)检测荧光强度(激发光488 nm,发射光525 nm)。
1.5 纳米MoS2作用后细胞膜通透性检测
对数生长期的菌液用PBS洗涤2次,再用液体LB培养基重悬,并稀释至108 CFU·mL-1。供、受体菌按1∶1的比例混合后加入纳米MoS2悬液,使终浓度为25 mg·L-1(空白对照组加入等量双蒸水),作用2 h后,稀释至107 CFU·mL-1。取1 mL加入5 μL的PI染液(E607306,Sangon Biotech),37 ℃培养箱避光放置20 min,用流式细胞仪(Cell Sorter S3e,BioRad,美国)检测。
1.6 实时定量聚合酶链反应定量基因表达
实验组和对照组接合2 h后收集菌体用RNA提取试剂盒(DP430,天根)提取总RNA,再用反转录试剂盒(KR116-02,天根)将RNA反转录得到cDNA。以cDNA作为模板,用PowerUp SYBR Master Mix(A25742,Thermo)在CFX96 BioRad qPCR系统(CFX96 Optics Module,BioRad,新加坡)上进行实时定量聚合酶链反应(qPCR),定量基因的表达。qPCR反应体系如下:10 μL PowerUp SYBR Master Mix,7 μL ddH2O,上下游引物各1 μL,模板1 μL;qPCR反应条件如下:95 ℃预变性5 min,40个循环的95 ℃变性10 s、60 ℃退火30 s、72 ℃延伸10 s,72 ℃终止10 min,熔解曲线65 ℃升至95 ℃。qPCR中所有引物使用DNAStar 7.1设计,由上海生工合成,序列如表1所示。
实验中基因的表达量由绝对定量的方法计算获得,并利用16S RNA作为内参基因修正体系中的细胞含量。相关基因的标准曲线如图1所示。
1.7 扫描电镜观察细菌形态
实验组和对照组接合2 h后在4 ℃条件下6 000 r·min-1离心5 min,弃上清后加入2.5%戊二醛(PH9003,Phygene)在4 ℃条件下固定48 h,PBS离心洗涤3次,依次用30%、50%、70%、80%、90%、95%和100%无水乙醇梯度脱水,其中100%无水乙醇脱水2次。每次脱水10 min,然后6 000 r·min-1下离心3 min,最后置于冷冻干燥机(FDU-1200,EYELA,上海,中国)中冻干,用扫描电镜(Sigma300,ZEISS,德国)观察并用能谱仪(Xplore 30,OXFORD,英国)进行元素扫描分析。
2 结果(Results)
2.1 纳米MoS2浓度对pCF10质粒接合转移的影响
接合2 h后,空白对照组的接合频率为3.7×10-3,与空白对照组比较,经过纳米MoS2处理后接合频率都有所提高,最高可以达到2.4×10-2。纳米MoS2的作用具有浓度效应,促进效果呈现先增大再减小的趋势,其中25 mg·L-1纳米MoS2的促进效果最明显,接合频率可以达到空白对照组的5倍~8倍(图2(a))。但对供、受体菌的生长无影响(图2(b)),说明纳米MoS2只影响了接合转移过程。
2.2 纳米MoS2作用时间对pCF10质粒接合转移的影响
将纳米MoS2加入接合体系中,使终浓度为25 mg·L-1,对不同时刻的接合子计数,计算接合频率。空白对照组在4 h到达接合终点,接合频率最高,达到1.4×10-2。纳米MoS2作用后与对照组比较,1~8 h的接合频率都有所提高,最高可以达到3.7×10-2,并且将接合终点由4 h提前到2 h。综合来看纳米MoS2在不同时刻的促进效果不同,其中2 h时促进效果最明显,可以将接合频率提高6倍(图3)。
2.3 纳米MoS2作用下温度对pCF10质粒接合转移的影响
将接合体系在不同温度下接合2 h,计算接合频率。对照组在16 ℃的接合频率较低为7×10-6,26 ℃接合频率提高到1.3×10-3,26~37 ℃温度升高后接合频率基本不变。纳米MoS2作用后随着温度升高接合频率增加,37 ℃接合频率最高,达到4×10-3。实验组与对照组比较,37 ℃时促进效果最显(图4)。
2.4 纳米MoS2作用下pH对pCF10质粒接合转移的影响
调节接合体系的pH,在37 ℃条件下接合2 h,pH=6~8范围内,随着pH的升高,实验组与对照组的接合频率都增加,分别为2.3×10-3和3.7×10-3。pH为8.0时促进效果最明显(图5)。
2.5 纳米MoS2对细胞膜通透性的影响
质粒的接合转移需要供体菌和受体菌细胞膜间的接触融合,这一过程与细胞膜状态直接相关。为了确定纳米MoS2影响接合转移的机制,对纳米MoS2处理后的细胞膜通透性进行了测定。PI染液是一种细胞核染色试剂,不能通过完整的细胞膜,但可以通过破损的细胞膜对核染色。通过检测对照组与实验组的荧光细胞的比例,可以反映细胞膜通透性的变化。经检测,空白对照组中荧光细胞比例为0.55%;MoS2作用2 h后荧光细胞比例为0.66%(图6)。荧光细胞比例只发生微小变化,二者没有统计学差异,说明纳米MoS2没有对细胞膜通透性产生明显影响。
2.6 纳米MoS2对细胞活性氧水平的影响
纳米材料的小粒径及高生物活性,可以使细菌产生羟基自由基(·OH),进而产生活性氧(ROS)反应,利用DCFH-DA探针可以检测细胞内活性氧水平。DCFH-DA探针进入细胞后,在活性氧存在的条件下,被氧化生成荧光物质,通过检测对照组与实验组的荧光强度,可以反映细胞内活性氧水平。经检测,MoS2作用2 h后,供体菌和受体菌中荧光强度值都在800左右(图7),说明纳米MoS2没有对细胞活性氧水平产生明显影响。
2.7 纳米MoS2对pCF10质粒接合转移调控基因表达的影响
纳米MoS2可以促进pCF10质粒接合转移的发生,浓度为25 mg·L-1的纳米MoS2,在37 ℃、pH=8.0的条件下作用于接合体系2 h后pCF10质粒的接合转移频率可升高5倍~8倍。因为接合转移过程受到一系列基因的调控,用荧光定量PCR的方法对调控基因的表达情况进行定量。ccfA和prgQ分别是编码信息素cCF10和iCF10的基因,prgA和prgB是编码黏附蛋白的基因,prgZ是负责信息素导入的基因,prgY是供体自诱导的调控基因。25 mg·L-1的纳米MoS2作用于接合体系2 h后,与空白对照组比较,这些基因的表达都无明显变化(图8),说明纳米MoS2不会影响基因的表达,这是因为纳米MoS2是惰性材料,有良好的生物相容性。
表1 引物序列
Table 1 Primer sequence

基因Gene引物Primer序列Sequence功能Function产物长度/bpProducts size/bpprgAprgBprgA-FGTTTGATGATGGCCGCTTAGGTTAprgA-RCACTTCGCCGTTTTGGTATTGACTprgB-FAGCGGATGGAAAATTTTACTCACCprgB-RGCGCACTAGATACAGGCACATTA编码黏附蛋白Coding adhesive protein编码黏附蛋白Coding adhesive protein175197prgYprgY-FTTTGGAAGGTGCGGAAGAATCprgY-RGTAAATGCGCCTGCCCAATA调控供体自诱导Regulating self-induction202prgZprgZ-FGACTTTGACTGCAGGGACACCprgZ-RAGCGCCATCTCTAATCACAAT负责信息素转运Transporting pheromone 202prgQprgQ-FTATAGGAGGGGTGTAAATGAAAACprgQ-RCCGTCCTATGGCAGTCG编码iCF10信息素Coding iCF10 pheromone143ccfAccfA-FAGATATACGAAGGCCCGCTGAAccfA-RAATTAGTTATCCGCTGCTCCTGA编码cCF10信息素Coding cCF10 pheromone24316S16S-F16S-RGCGGCGTGCCTAATACACCGCGGGTCCATCC内参基因Reference genes219
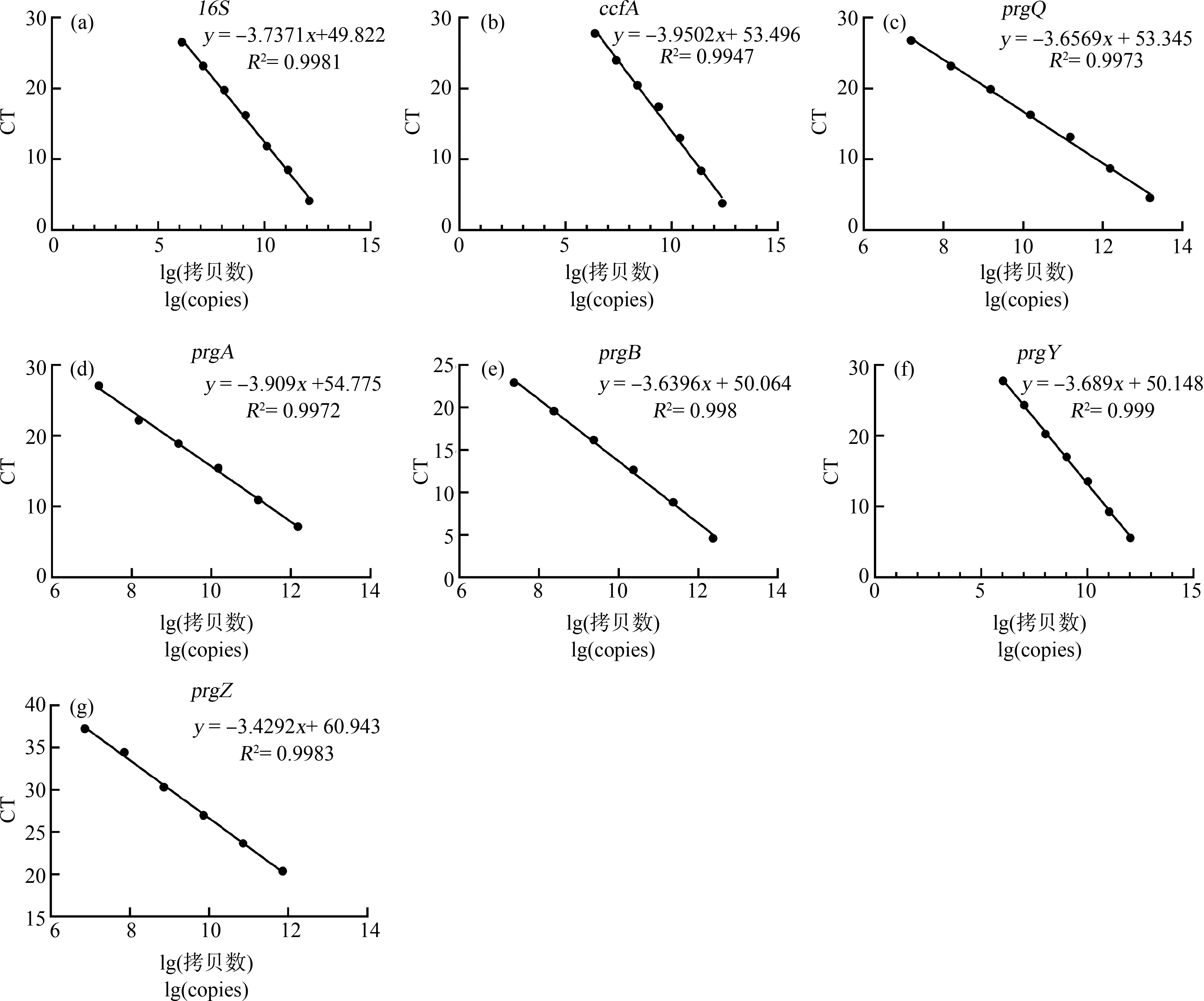
图1 qPCR实验中基因的标准曲线
注:CT值表示扩增程中,扩增产物的荧光信号达到设定的荧光阈值时所对应的扩增循环数(cycle threshold)。
Fig. 1 Standard curves of genes in qPCR experiments
Note: CT represents the corresponding amplification cycle threshold when the fluorescence signal of the amplification product reaches the set
fluorescence threshold during the amplification process.
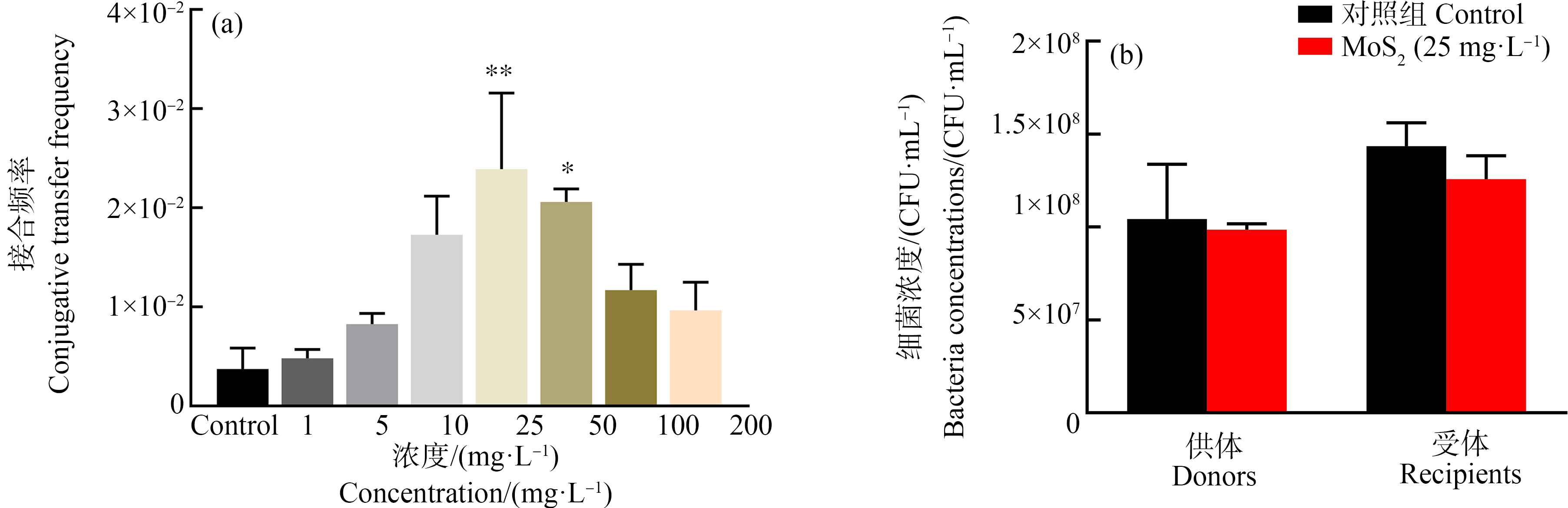
图2 纳米MoS2对接合转移的影响
注:(a)不同浓度纳米MoS2对pCF10质粒接合转移的影响规律;(b)纳米MoS2对供体菌和受体菌的影响;MoS2表示二硫化钼,
*表示P<0.05,**表示P<0.01。
Fig. 2 Effect of nano-MoS2 on conjugation transfer
Note: (a) Effects of different concentrations of nano-MoS2 on pCF10 plasmid conjugation and transfer; (b) Effects of nano-MoS2
on donor bacteria and recipient bacteria; MoS2 stands for molybdenum disulfide, *represents P<0.05, **represents P<0.01.
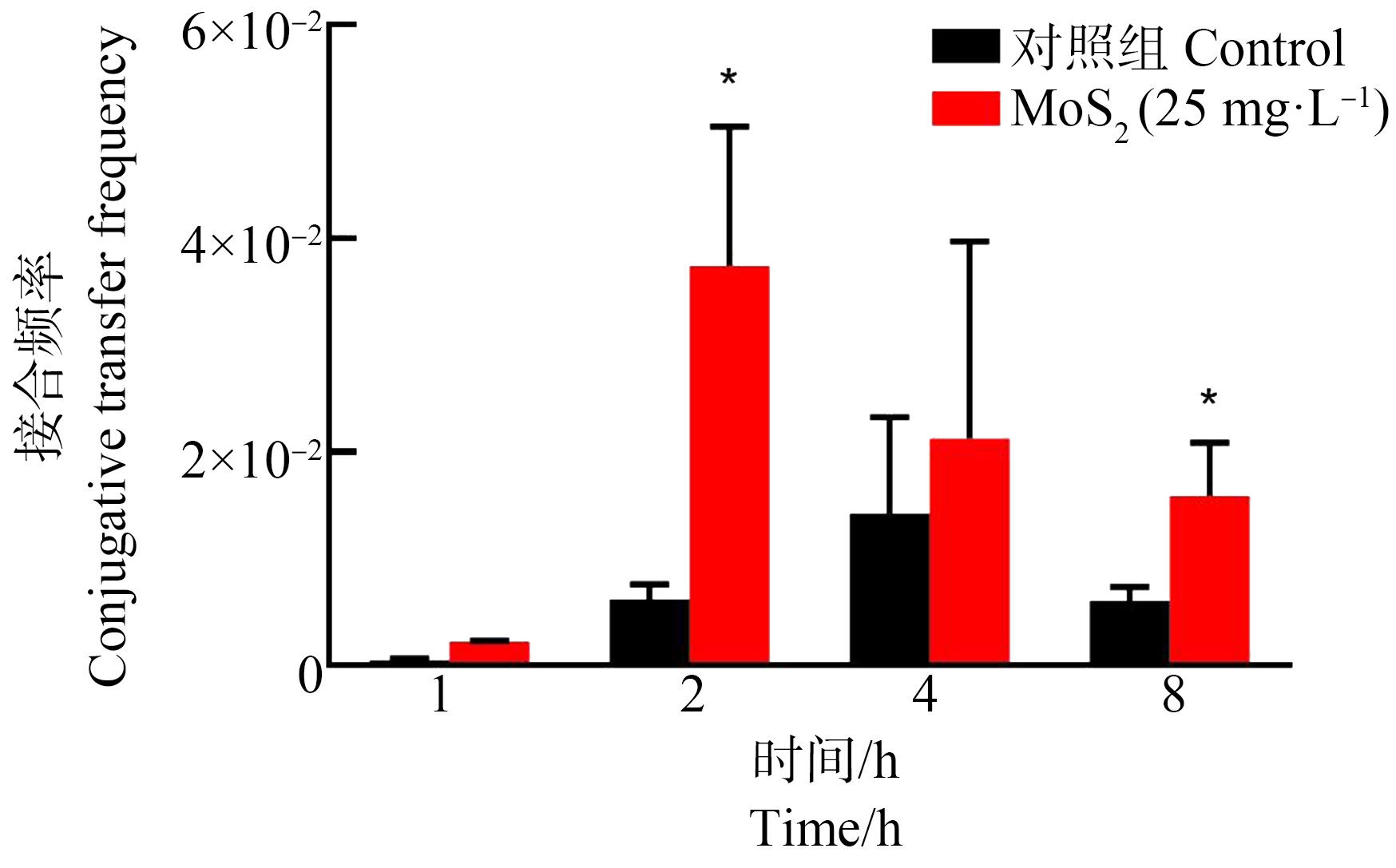
图3 纳米MoS2作用时间对pCF10质粒接合转移的影响
注:*表示P<0.05。
Fig. 3 Effect of MoS2 action time on pCF10 plasmid
conjugation transfer
Note: *represents P<0.05.
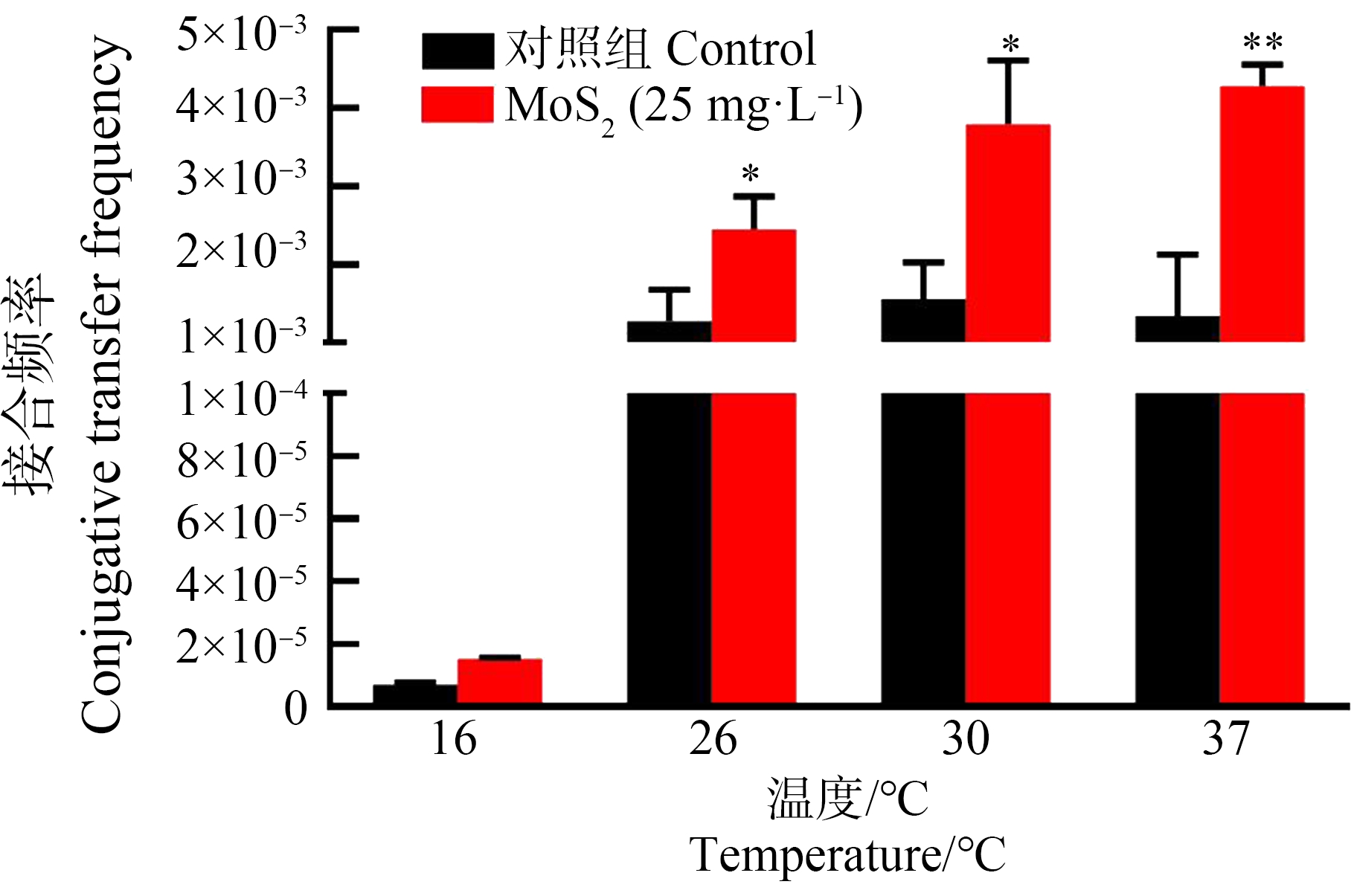
图4 纳米MoS2作用下温度对pCF10质粒接合转移的影响
注:*表示P<0.05,**表示P<0.01。
Fig. 4 Effect of temperature on pCF10 plasmids conjugation
transfer under nano-MoS2
Note: *represents P<0.05, **represents P<0.01.
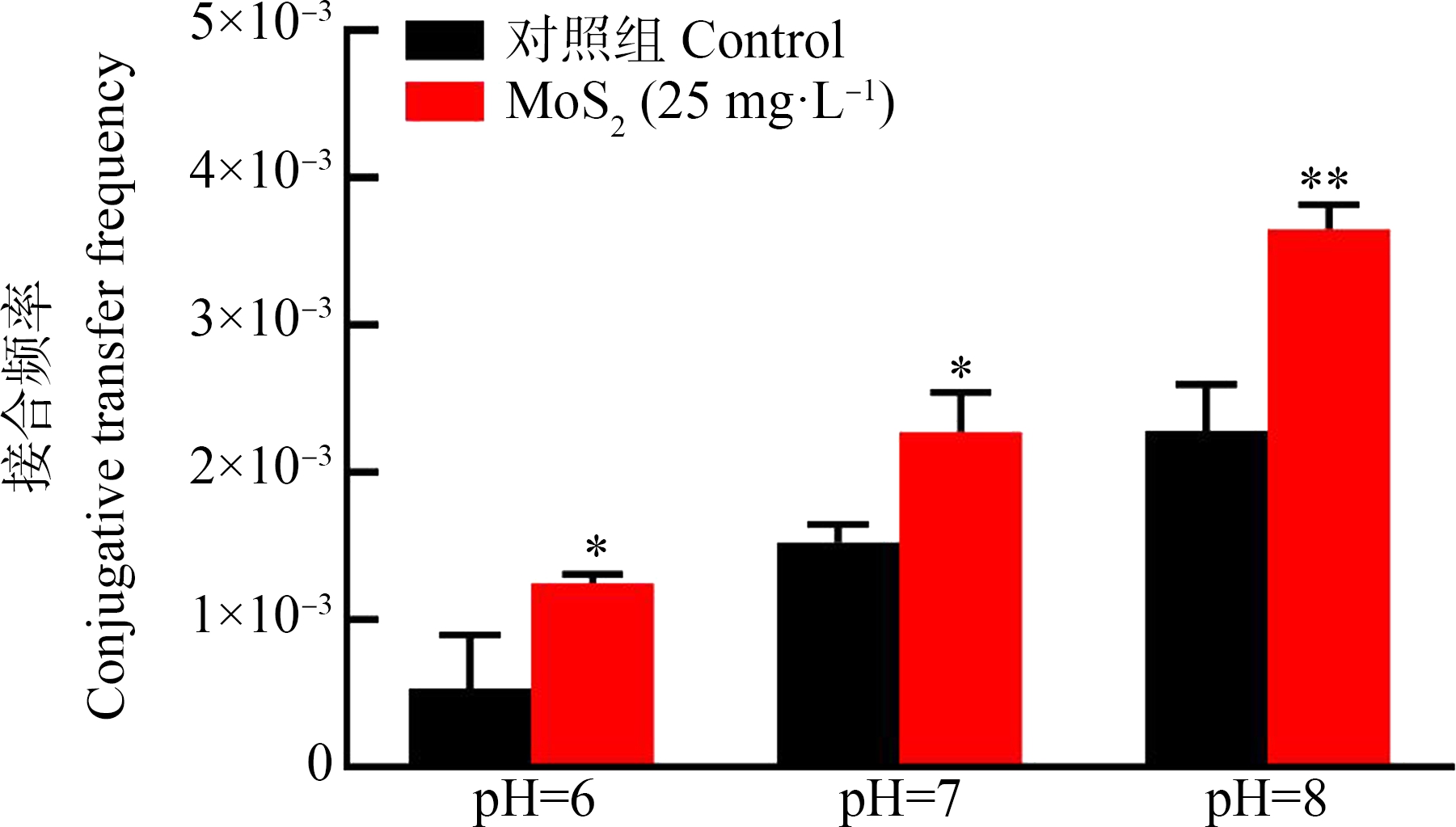
图5 纳米MoS2作用下pH对pCF10质粒接合转移的影响
注:*表示P<0.05,**表示P<0.01。
Fig. 5 Effect of pH on pCF10 plasmids conjugation transfer
under nano-MoS2
Note: *represents P<0.05, **represents P<0.01.
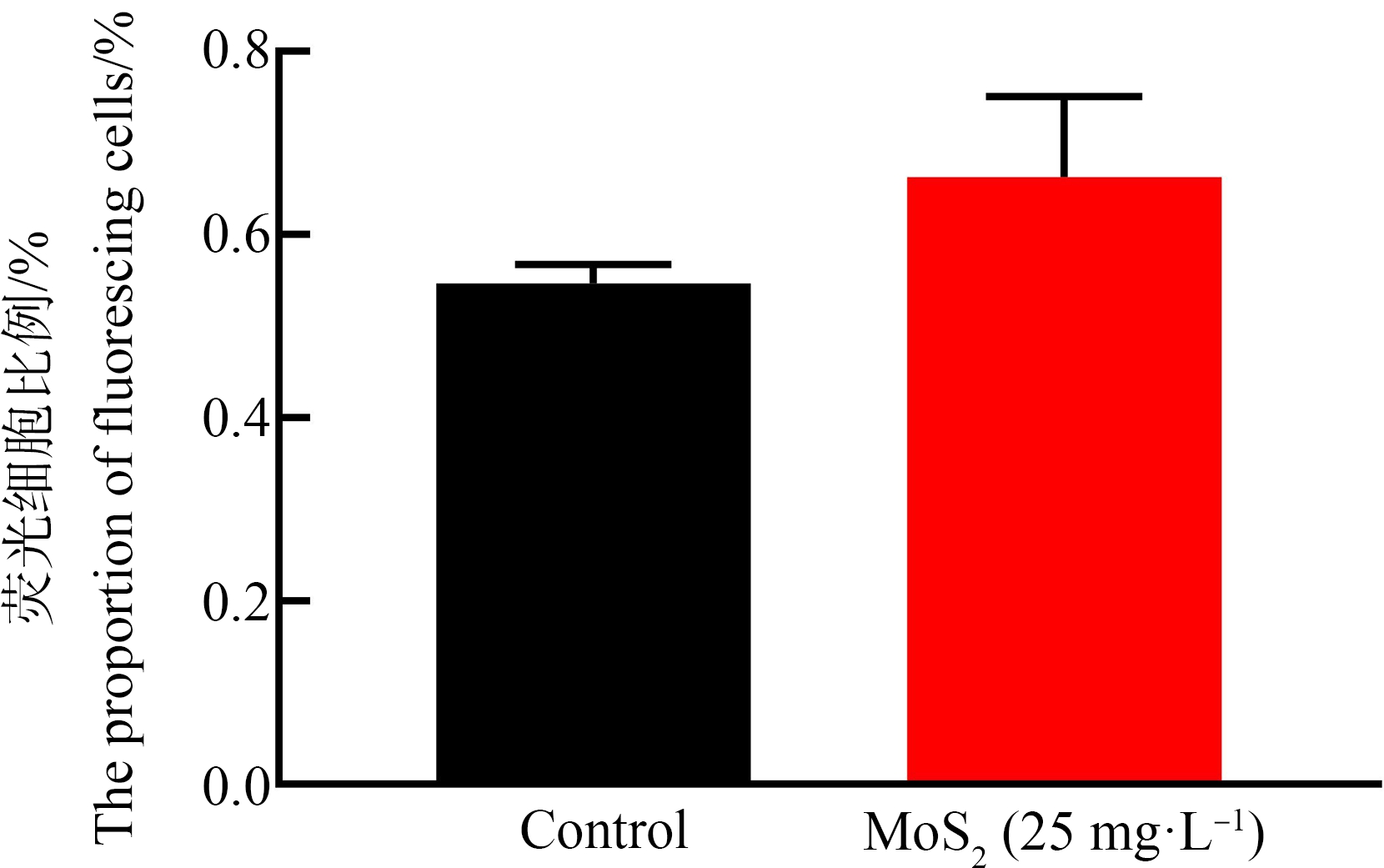
图6 纳米MoS2对细胞膜通透性的影响
Fig. 6 Effect of nano-MoS2 on cell membrane permeability
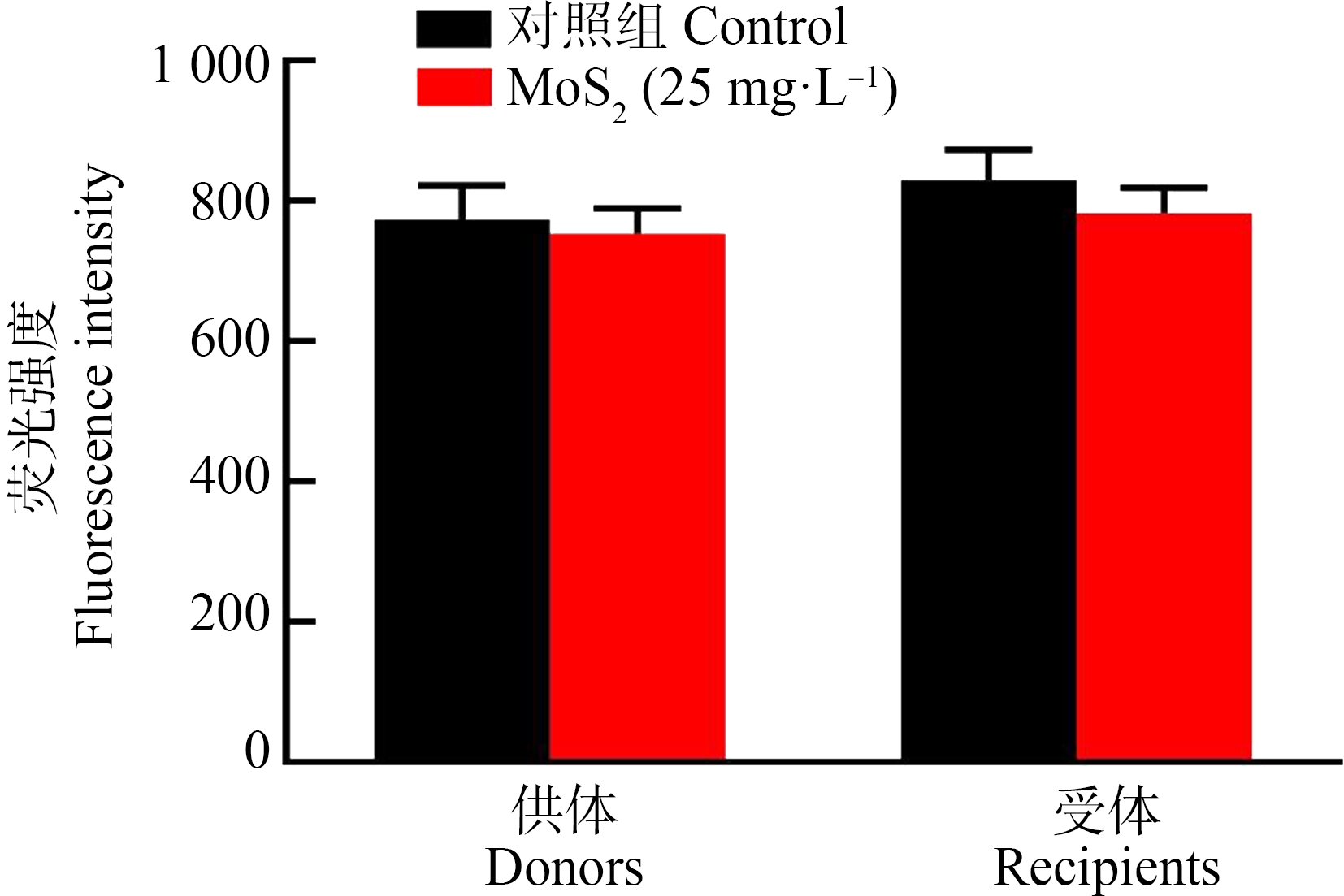
图7 纳米MoS2对细胞活性氧水平的影响
Fig. 7 Effect of nano-MoS2 on the level of reactive
oxygen species in cells
2.8 纳米MoS2促进细菌聚集
细菌聚集是接合转移发生的重要前提,粪肠球菌自身的黏附蛋白可以促进细菌的黏附和聚集,但纳米MoS2作用后并没有使黏附蛋白编码基因上调。通过扫描电镜发现,纳米MoS2加入到接合体系中会附着在细菌表面(图9),能谱仪元素分析的结果也显示在团聚的细菌表面有钼(molybdenum, Mo)元素和硫(sulfur, S)元素分布(图10)。因为纳米MoS2本身具有吸附性能,而且MoS2是片层状的结构,不容易进入细胞内部,这使得纳米MoS2会吸附在细菌表面。与空白对照组比较,表面吸附了纳米MoS2的细菌会变得更加聚集(图9),进而促进了接合转移的发生,使接合频率提高。
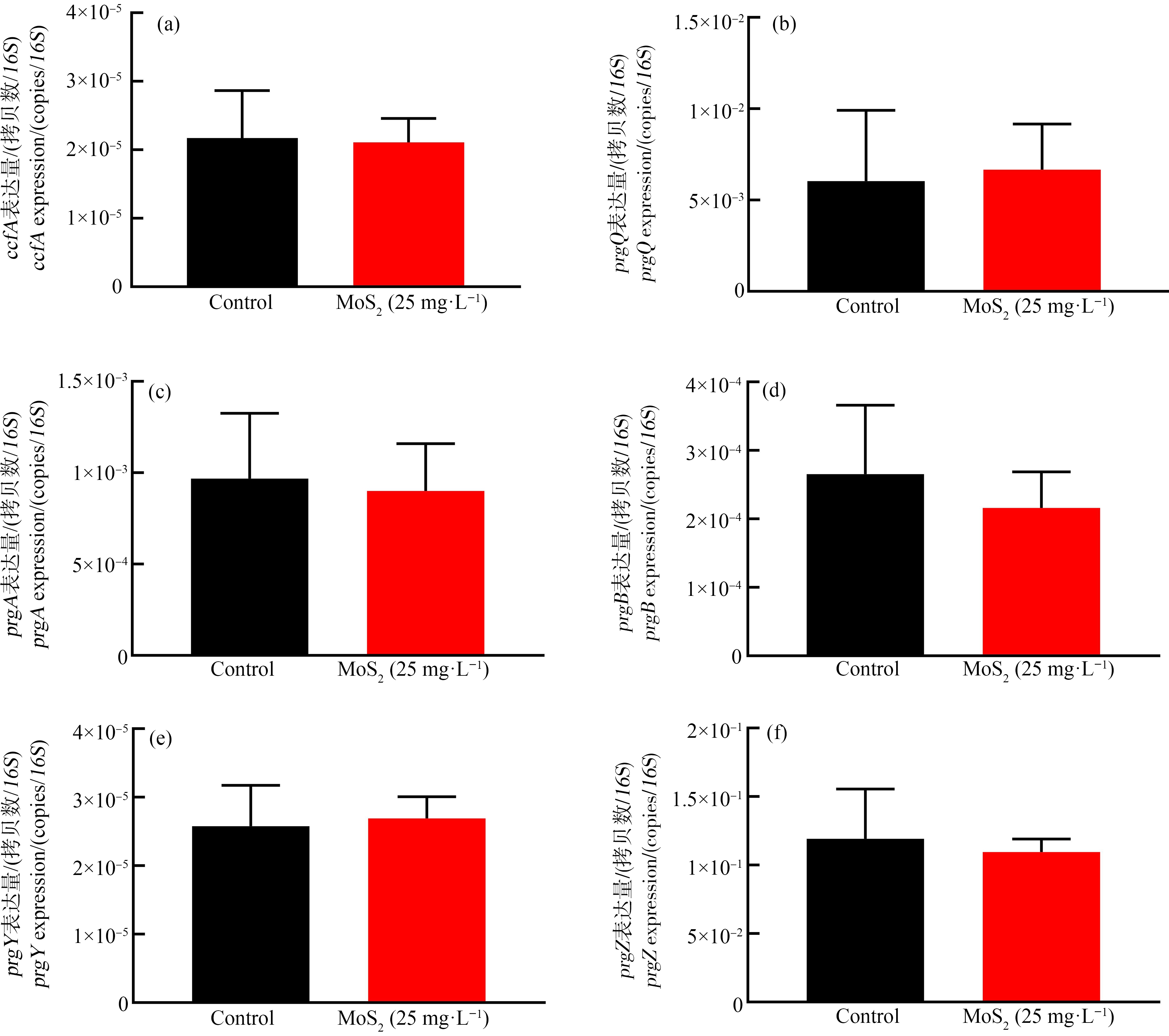
图8 纳米MoS2对质粒接合转移调控基因表达的影响
注:(a) 纳米MoS2对编码cCF10信息素的ccfA基因的影响;
(b) 纳米MoS2对编码iCF10信息素的prgQ基因的影响;(c) 纳米MoS2对编码黏附蛋白的prgA基因的影响;
(d) 纳米MoS2对编码黏附蛋白的prgB基因的影响;(e) 纳米MoS2对供体自诱导调控基因prgY的影响;(f) 纳米MoS2对负
责信息素导入的prgZ基因的影响。
Fig. 8 Effect of nano-MoS2 on gene expression of plasmid conjugation transfer
Note: (a) Effect of nano-MoS2 on ccfA gene encoding cCF10 pheromone;
(b) Effect of nano-MoS2 on prgQ gene encoding iCF10 pheromone; (c) Effect of nano-MoS2 on prgA gene encoding
adhesive protein; (d) Effect of nano-MoS2 on prgB gene encoding adhesive protein; (e) Effect of nano-MoS2 on donor
self-induced regulatory gene prgY; (f) Effect of nano-MoS2 on prgZ gene responsible for pheromone import.
3 讨论(Discussion)
粪肠球菌是一种通常生活在人类和其他动物胃肠道中的细菌,也会作为益生菌添加到饲料中[25]。由于粪便污染和饲料浪费,最终会导致环境中粪肠球菌的数量增加。同时革兰氏阳性菌中的肠球菌、链球菌和葡萄球菌是引起医院感染最常见的病原体,在公共卫生方面需要特别关注致病性革兰氏阳性菌中耐药性的传播。例如万古霉素的耐药性,这在肠球菌中很常见,虽然肠球菌感染一般不会危及生命,但万古霉素耐药基因一旦传播给金黄色葡萄球菌等细菌就会导致致命疾病[26]。临床上肠球菌已成为引起医院感染的第二大致病菌[27],并可以通过接合转移的方式传播ARGs[16],使其对人类健康和环境安全的风险日益增加。然而,目前人们往往关注于革兰氏阴性菌,如大肠杆菌中耐药基因的接合转移研究,而忽视了革兰氏阳性菌中耐药基因的接合转移。因此本研究中建立了革兰氏阳性菌粪肠球菌中pCF10质粒介导的耐药基因接合转移模型。
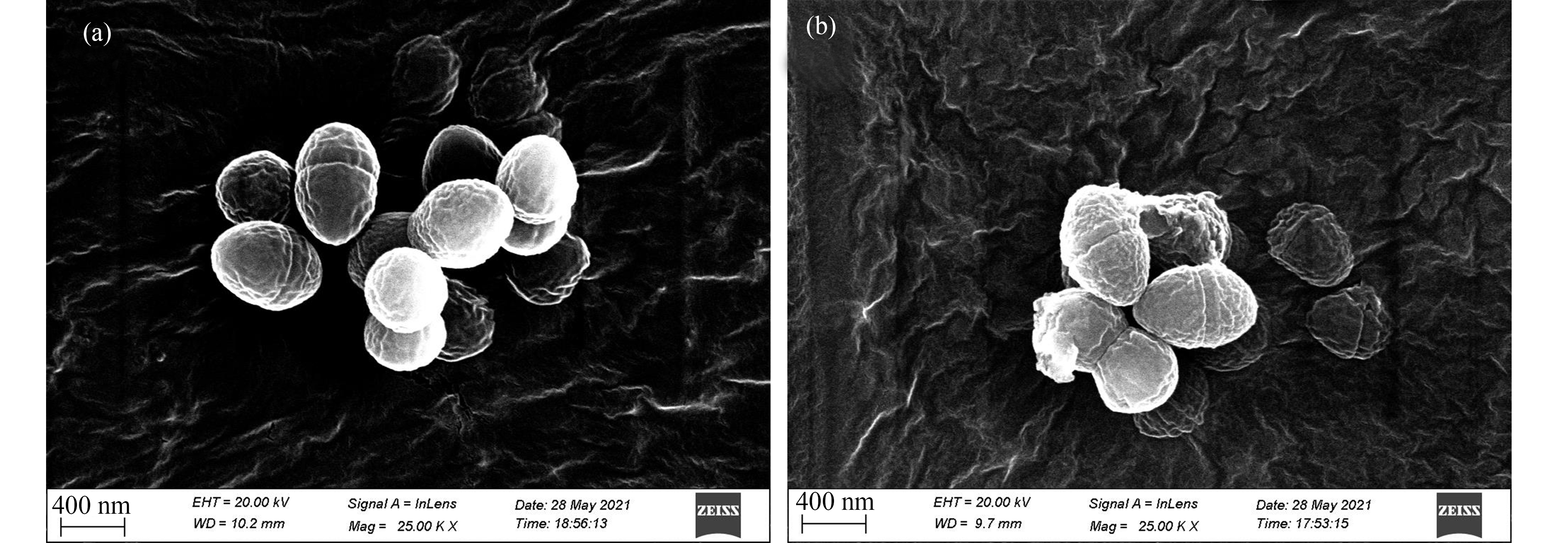
图9 纳米MoS2促进细菌聚集
注:(a) 空白对照组;(b) 加入纳米MoS2的实验组。
Fig. 9 Bacterial aggregation promoted by nano-MoS2
Note: (a) Blank control group; (b) Added nano-MoS2.

图10 纳米MoS2附着在细菌表面
注:(a) SEM图像;(b) Mo元素分布;(c) S元素分布;Mo表示钼元素,S表示硫元素。
Fig. 10 Nano-MoS2 attached to the surface of bacteria
Note: (a) SEM images; (b) Distribution of Mo element; (c) Distribution of S element; Mo stands for molybdenum element,
and S stands for sulfur element.
pCF10质粒是信息素诱导质粒,接合转移过程受到信息素的调节[16],对编码信息素的ccfA和prgQ基因,以及与信息素相关的prgY和prgZ基因的表达进行定量分析,发现纳米MoS2并没有对这些基因的表达产生影响,说明纳米MoS2没有影响信息素的产生和转运。另外黏附蛋白在pCF10质粒的接合转移过程中也发挥重要作用,它使供体菌与受体菌可以紧密地结合在一起。对编码黏附蛋白的prgA和prgB基因的表达进行定量分析,发现纳米MoS2同样没有对这些基因的表达产生影响。
一些纳米材料会通过影响活性氧系统和改变细胞膜的通透性影响质粒的接合转移。例如纳米氧化铝可以使革兰氏阴性菌中羟基自由基的生成增加,同时细菌的总抗氧化能力、过氧化氢酶活性、超氧化物歧化酶活性和谷胱甘肽还原酶活性也随着纳米氧化铝水平的升高而升高。另外还会破化细胞膜结构并进入细菌细胞[21]。对本实验中纳米MoS2作用后粪肠球菌的活性氧水平和细胞膜的通透性进行了测定,发现纳米MoS2对活性氧系统和细胞膜的通透性均没有影响,这一结果与基因表达的结果一致。这可能是由于粪肠球菌是革兰氏阳性菌,细胞壁较厚,纳米材料不易进入细菌细胞,另外纳米MoS2是片层状的二维结构,比纳米颗粒更难进入细胞;纳米MoS2还具有良好的生物相容性,这些性状使得纳米MoS2不会影响细菌生长和细菌结构,因此分子水平和细胞膜水平没有发生变化。
扫描电镜和能谱分析结果表明,纳米MoS2会黏附在细菌表面,促进细菌聚集,为细菌之间的接合转移提供良好的条件,最终导致接合转移频率增加。也有报道称在转化过程中细菌的黏附和运动会使细菌摄入外界DNA[28],接合过程也是受体菌获取受体菌之外的DNA,而且接合过程需要供体菌与受体菌接触,然后通过Ⅳ型分泌系统将供体中的质粒传递给受体[29],所以细菌的聚集为接合转移的发生提供了很好的环境。纳米MoS2正是因为促进了细菌聚集才导致接合转移频率增加。
作为一种类石墨烯材料,MoS2的规模化生产和市场化发展迅速[30]。可能会有大量的MoS2从实验室中排出,此外,清洁产品和润滑剂的应用会将数以吨计的纳米MoS2释放到环境中[31],MoS2在环境中的释放将对生物体和人类健康构成潜在风险[32]。虽然纳米MoS2具有良好的生物相容性,但是较高的浓度会对生物体表现出显著的毒性。目前关于纳米MoS2的环境残留浓度还未见报道,有一些关于纳米MoS2生物毒性的研究发现:50 mg·L-1的MoS2对A549细胞活力的抑制率为25.6%[33];>25 mg·L-1的MoS2纳米片通过干扰细胞表面受体和mTOR途径诱导LC3-GFP U87细胞自噬[34];饲料中150 mg·kg-1 MoS2可引起肠道炎症,扰乱小鼠肠道氨基酸代谢和肠道微生物群落[31]。本研究通过研究纳米MoS2对粪肠球菌中接合转移的影响,进一步完善了纳米MoS2的生态环境风险评价,为纳米MoS2的应用提供了更丰富的理论指导。
通讯作者简介:邱志刚(1979—),男,博士,研究员,主要研究方向为纳米材料生物安全性及细菌耐药基因。
[1] Huemer M, Mairpady Shambat S, Brugger S D, et al. Antibiotic resistance and persistence-Implications for human health and treatment perspectives [J]. EMBO Reports, 2020, 21(12): e51034
[2] Forsberg K J, Reyes A, Wang B, et al. The shared antibiotic resistome of soil bacteria and human pathogens [J]. Science, 2012, 337(6098): 1107-1111
[3] Karkman A, Do T T, Walsh F, et al. Antibiotic-resistance genes in waste water [J]. Trends in Microbiology, 2018, 26(3): 220-228
[4] Che Y, Xia Y, Liu L, et al. Mobile antibiotic resistome in wastewater treatment plants revealed by nanopore metagenomic sequencing [J]. Microbiome, 2019, 7(1): 44
[5] Thanner S, Drissner D, Walsh F. Antimicrobial resistance in agriculture [J]. mBio, 2016, 7(2): e02227-e02215
[6] San Millan A. Evolution of plasmid-mediated antibiotic resistance in the clinical context [J]. Trends in Microbiology, 2018, 26(12): 978-985
[7] Huddleston J R. Horizontal gene transfer in the human gastrointestinal tract: Potential spread of antibiotic resistance genes [J]. Infection and Drug Resistance, 2014, 7: 167-176
[8] Furuya E Y, Lowy F D. Antimicrobial-resistant bacteria in the community setting [J]. Nature Reviews Microbiology, 2006, 4(1): 36-45
[9] Pal C, Bengtsson-Palme J, Kristiansson E, et al. Co-occurrence of resistance genes to antibiotics, biocides and metals reveals novel insights into their co-selection potential [J]. BMC Genomics, 2015, 16: 964
[10] George R C, Uttley A H. Susceptibility of enterococci and epidemiology of enterococcal infection in the 1980s [J]. Epidemiology and Infection, 1989, 103(3): 403-413
[11] Mundy L M, Sahm D F, Gilmore M. Relationships between enterococcal virulence and antimicrobial resistance [J]. Clinical Microbiology Reviews, 2000, 13(4): 513-522
[12] Sievert D M, Ricks P, Edwards J R, et al. Antimicrobial-resistant pathogens associated with healthcare-associated infections summary of data reported to the national healthcare safety network at the centers for disease control and prevention, 2009—2010 [J]. Infection Control & Hospital Epidemiology, 2013, 34(1): 1-14
[13] 游选旺, 唐毕华, 黄健. 214株肠球菌的耐药性分析[J]. 实验与检验医学, 2009, 27(6): 627-628
[14] Raza T, Ullah S R, Mehmood K, et al. Vancomycin resistant Enterococci: A brief review [J]. Journal of the Pakistan Medical Association, 2018, 68(5): 768-772
[15] Price V J, Huo W W, Sharifi A, et al. CRISPR-cas and restriction-modification act additively against conjugative antibiotic resistance plasmid transfer in Enterococcus faecalis [J]. mSphere, 2016, 1(3): e00064-e00016
[16] Breuer R J, Hirt H, Dunny G M. Mechanistic features of the enterococcal pCF10 sex pheromone response and the biology of Enterococcus faecalis in its natural habitat [J]. Journal of Bacteriology, 2018, 200(14): e00733-e00717
[17] Hirt H, Greenwood-Quaintance K E, Karau M J, et al. Enterococcus faecalis sex pheromone cCF10 enhances conjugative plasmid transfer in vivo [J]. mBio, 2018, 9(1): e00037-e00018
[18] Witte W. Ecological impact of antibiotic use in animals on different complex microflora: Environment [J]. International Journal of Antimicrobial Agents, 2000, 14(4): 321-325
[19] 俞道进, 曾振灵, 陈杖榴. 四环素类抗生素残留对水生态环境影响的研究进展[J]. 中国兽医学报, 2004, 24(5): 515-517
[20] Mackevica A, Olsson M E, Hansen S F. The release of silver nanoparticles from commercial toothbrushes [J]. Journal of Hazardous Materials, 2017, 322(Pt A): 270-275
[21] Qiu Z G, Yu Y M, Chen Z L, et al. Nanoalumina promotes the horizontal transfer of multiresistance genes mediated by plasmids across genera [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(13): 4944-4949
[22] Wang X, Mansukhani N D, Guiney L M, et al. Differences in the toxicological potential of 2D versus aggregated molybdenum disulfide in the lung [J]. Small, 2015, 11(38): 5079-5087
[23] Dunny G M, Brown B L, Clewell D B. Induced cell aggregation and mating in Streptococcus faecalis: Evidence for a bacterial sex pheromone [J]. Proceedings of the National Academy of Sciences of the United States of America, 1978, 75(7): 3479-3483
[24] Dunny G, Funk C, Adsit J. Direct stimulation of the transfer of antibiotic resistance by sex pheromones in Streptococcus faecalis [J]. Plasmid, 1981, 6(3): 270-278
[25] 刘静慧, 吴峰洋, 陈宝江, 等. 粪肠球菌的益生特性和毒副作用研究[C]. 中国新疆和田: 第九届(2019)中国兔业发展大会, 2019
[26] Grohmann E, Muth G, Espinosa M. Conjugative plasmid transfer in gram-positive bacteria [J]. Microbiology and Molecular Biology Reviews, 2003, 67(2): 277-301
[27] Hidron A I, Edwards J R, Patel J, et al. NHSN annual update: Antimicrobial-resistant pathogens associated with healthcare-associated infections: Annual summary of data reported to the National Healthcare Safety Network at the Centers for Disease Control and Prevention, 2006—2007 [J]. Infection Control and Hospital Epidemiology, 2008, 29(11): 996-1011
[28] Bakkali M. Could DNA uptake be a side effect of bacterial adhesion and twitching motility? [J]. Archives of Microbiology, 2013, 195(4): 279-289
[29] Li F, Alvarez-Martinez C, Chen Y Q, et al. Enterococcus faecalis PrgJ, a VirB4-like ATPase, mediates pCF10 conjugative transfer through substrate binding [J]. Journal of Bacteriology, 2012, 194(15): 4041-4051
[30] Huang C C, Al-Saab F, Wang Y D, et al. Scalable high-mobility MoS2 thin films fabricated by an atmospheric pressure chemical vapor deposition process at ambient temperature [J]. Nanoscale, 2014, 6(21): 12792-12797
[31] Zou W, Li X Y, Li C H, et al. Influence of size and phase on the biodegradation, excretion, and phytotoxicity persistence of single-layer molybdenum disulfide [J]. Environmental Science & Technology, 2020, 54(19): 12295-12306
[32] Guiney L M, Wang X, Xia T, et al. Assessing and mitigating the hazard potential of two-dimensional materials [J]. ACS Nano, 2018, 12(7): 6360-6377
[33] Chng E L K, Sofer Z, Pumera M. MoS2 exhibits stronger toxicity with increased exfoliation [J]. Nanoscale, 2014, 6(23): 14412-14418
[34] Zhou X F, Jia J B, Luo Z, et al. Remote induction of cell autophagy by 2D MoS2 nanosheets via perturbing cell surface receptors and mTOR pathway from outside of cells [J]. ACS Applied Materials & Interfaces, 2019, 11(7): 6829-6839