有机磷类化合物(organophosphorus compounds, OPs)包括有机磷阻燃剂(organophosphorus flame retardants, OPFRs)、有机磷杀虫剂(organophosphorus pesticides, OPPs)和其他含磷的有机化合物。工业生产上,OFPRs作为溴化阻燃剂的低毒替代品被应用于家电、建筑和电子领域;农业上,OPPs则被广泛用于杀虫和害虫控制[1-2]。此外在医学领域,一些抗病毒的药物中也含有OPs[3]。因此,OPs容易通过磨损、挥发和溶解等方式进入各种环境介质中。目前科研人员已在诸如河流、地下水、海水、空气和室内等环境介质中检测到了OPs[4-6]。甚至在东亚地区的人类母乳样本中检测到10余种OPs,最高浓度为70 ng·g-1[7]。越来越广泛存在的OPs可能造成的危害和健康风险引起了研究者们的关注。
OPs具有神经毒性[8-9]、内分泌毒性[10]和致癌性[11]。研究表明,接触OPPs(如毒死蜱)会引起人类的神经系统发育缺陷[12]。OPFRs如磷酸三甲苯(TMPP)、磷酸三苯酯(TPHP)和磷酸三(2,3-二溴丙基)酯(TDBPP)则显示出对核受体(nuclear receptors, NRs)的亲和力和内分泌的干扰作用[13-14]。上述研究及更多OPs的毒性毒理研究往往都是通过传统的体内(in vivo)或者体外(in vitro)实验进行的,这些方法需要耗费大量的时间与金钱。此外,实验手段无法完整覆盖一整类化合物的毒性数据,并且测试在实验室间存在误差[15]。因此,迫切需要一种有效的替代方法,对化合物毒性数据进行评估。
随着计算科学的发展,计算毒理学已经成为一种具有成本效益且快速的实验替代方法[16]。计算毒理学整合各种来源的数据,开发基于数学、统计科学和机器学习的模型,对化学品进行高效和高通量的定性或定量预测[17]。定量结构活性关系(quantitative structure-activity relationship, QSAR)模型便是计算毒理学领域被广泛利用的模型之一,其原理是建立化合物的化学结构与各种毒理学终点效应和生物活性之间的函数关系[18]。QSAR模型被研究者们广泛应用于药物发现、毒理学和生物学等领域[19]。除了建立化学结构与毒性数据之间的关系模型外,QSAR模型还能通过分子描述符的解释从一定程度进行毒理机制的阐述。Alves等[20]使用QSAR方法为COVID-19病毒建立了抑制性药物预测模型,并应用该模型从小分子数据库中高通量地筛选了11个候选药物,其中3个被美国转化科学推动中心(National Center for Advancing Translational Sciences, NCATS)实验证实为有效的病毒抑制剂。Jeon等[21]则收集大量实验数据,为鱼胚胎急性毒性建立了一个通用的基线毒性QSAR模型,并使用该模型筛选毒性化合物。大量的研究表明QSAR模型用于化合物的毒性预测行之有效。
分子对接是研究化合物与目标蛋白结合部位行为的工具,能够通过计算手段探究化合物与特定靶点蛋白的结合特异性,从而从一定程度上反映化合物的毒性。Zou等[22]使用分子对接得到的配体-受体相互作用能量(binding energy)来反映化学品的毒性作用。Li等[23]通过对接羟基多溴联苯醚(HO-PBDEs)与人类甲状腺激素受体,从一定程度上揭示了HO-PBDEs的毒性机制。
现有的一些针对大鼠急性口服毒性的QSAR模型存在没有使用测试集[24]、选用的分子描述符数量不规范[25]等问题。在本研究中,完善地建立了基于逐步算法的线性回归(SW-MLR)和基于遗传算法的线性回归(GA-MLR)QSAR模型用于预测大鼠的急性口服毒性。研究收集的数据集中含有9种没有毒性实验数据的OPs,最终开发完成的QSAR模型将用于预测这9种OPs的急性毒性。分子对接也将作为一个辅助工具来评估这9种OPs潜在的神经毒性。
本研究首先收集了OPs对大鼠急性口服毒性的数据。检索并从文献中提取了62种环境中常见的OPs清单[26-27]。通过查询EPA CompTox数据库(https://comptox.epa.gov/dashboard),收集了同一实验条件下获得的53种OPs急性毒性数据LD50(导致一组实验动物中刚好死亡一半的化合物浓度)。获取的OPs的急性毒性数值使用log函数进行规范化处理。整个数据集按照4∶1的比例随机划分为训练集(n=43)和测试集(n=10),没有毒性实验数据的9种OPs作为最后的预测集(n=9)。在之后的计算实验中,训练集仅用于建立模型,测试集则用于评估模型的预测能力,这种划分方式有利于验证QSAR模型的拟合和泛化能力。
数据集中所有OPs的结构文件(.sdf)来自PubChem(https://pubchem.ncbi.nlm.nih.gov/)数据库。并使用PaDEL开源软件[28]计算所有化合物的分子结构描述符,共得到1 444个分子结构描述符,包括(1)一维结构描述符:如原子和化学键的数量、分子量等;(2)二维拓扑描述符:如拓扑指数、拓扑电荷和自相关系数等;(3)电荷描述符、原子电状态描述符等。本研究中使用了PaDEL计算的一维和二维描述符,因为这些类型的描述符计算速度快,可重复性好,并且不需要分子结构能量最小化过程,该过程中不同实验室间可能会在计算上产生误差。计算结束后,我们对得到的分子描述符数据进行了预处理,剔除了方差为0的单列分子描述符数据,相关系数>0.99的多组描述符最终仅保留一组。经过预处理后,最终保留了833组分子描述符用于后续分析。
分别使用逐步算法(SW)和遗传算法(GA)进行分子描述符的选择。2种算法选择的分子描述符变量通过基于普通最小二乘法(OLS)的多元线性回归(MLR)方法关联毒性终点并建立QSAR模型,之后进行模型的内部和外部评估。研究中使用SPSS 21.0软件建立了SW-MLR模型,使用DTC-QSAR开源软件[29]建立了GA-MLR模型。所有参数的统计分析过程在EXCEL软件中完成。本研究详细的QSAR模型构建及OPs的LD50预测工作流程如图1所示。
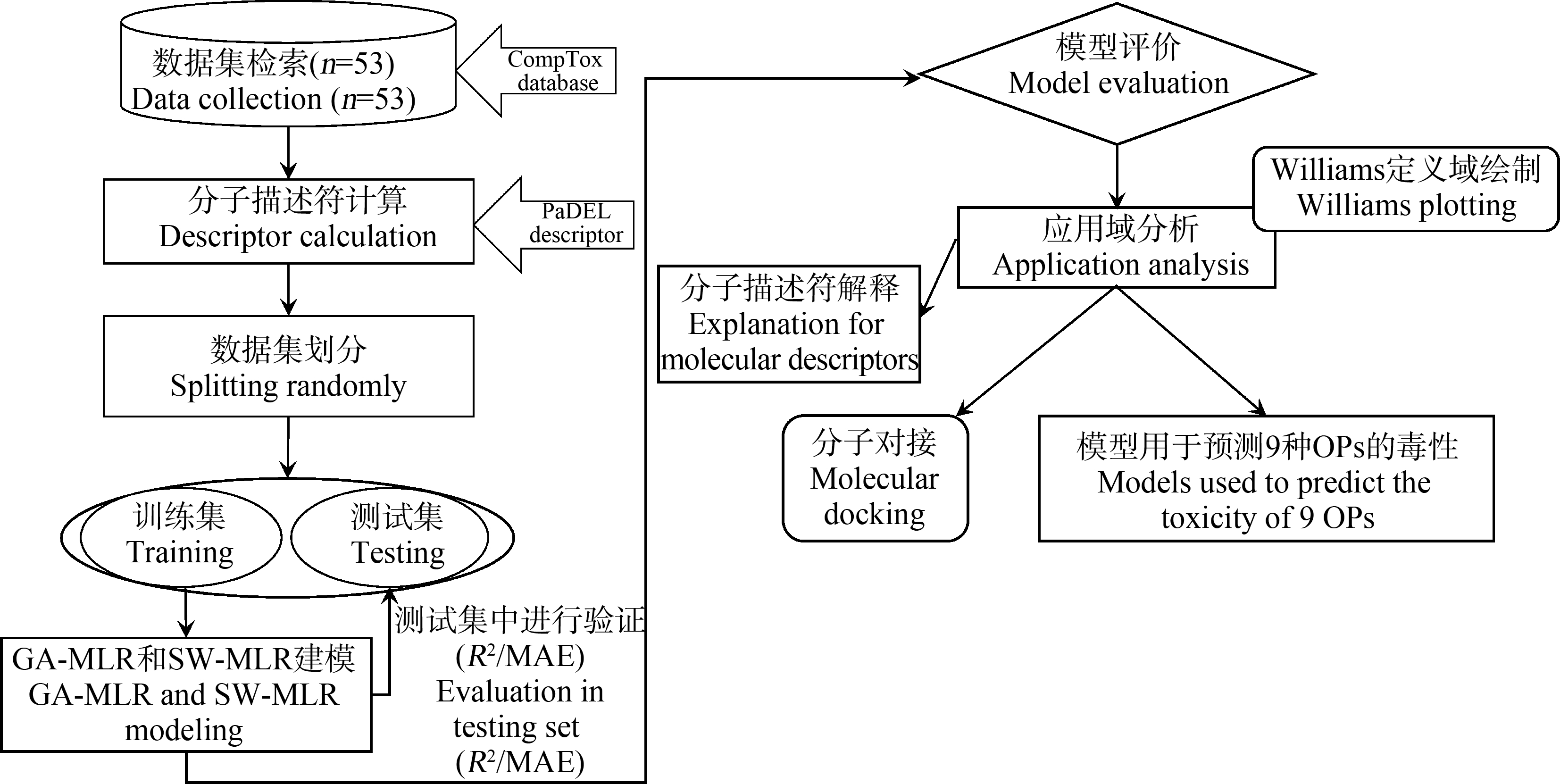
图1 定量结构活性关系(QSAR)模型构建及有机磷类化合物(OPs)的急性毒性半致死量(LD50)预测的工作流程图
注:GA-MLR表示基于遗传算法的多元线性回归模型;SW-MLR表示基于逐步算法的多元线性回归模型;
R2表示决定系数;MAE表示平均绝对误差。
Fig. 1 Workflow diagram for quantitative structure-activity relationship (QSAR) models construction and
acute toxicity semi-lethal dose (LD50) prediction of organophosphorus compounds (OPs)
Note: GA-MLR represents the multiple linear regression model based on genetic algorithm; SW-MLR represents the multiple linear regression
model based on stepwise algorithm; R2 represents the coefficient of determination; MAE represents mean absolute error.
模型建立后,使用训练集的统计参数评估模型的拟合程度和误差,使用测试集的统计参数评估模型的泛化能力与预测能力。本研究中基于训练集的统计参数有决定系数平方(R2)、估计的标准误差(SEE)和指定自由度(df)下的方差比(F)等。测试集的统计参数有平均绝对误差(MAE)、外部相关系数![]() 协调相关系数(CCC)等。此外,还针对模型的训练使用了留一法交叉验证
协调相关系数(CCC)等。此外,还针对模型的训练使用了留一法交叉验证![]() 和Y随机(Y-randomized testing)测试进行模型鲁棒性的评估。相应的统计参数与计算公式如表1所示。
和Y随机(Y-randomized testing)测试进行模型鲁棒性的评估。相应的统计参数与计算公式如表1所示。
使用Williams图(又称杠杆方法)[30]对建立的QSAR模型进行应用域分析。基于帽子矩阵(一类投影矩阵)得出的值(h)与标准化残差(δ)的比较。h大于警告杠杆值(h*)的化合物被认为是结构异常值,标准化残差>2.5的化合物则被认为是计算异常值。Williams图的公式定义如下:
(1)
表1 本研究中用于模型评价的公式及参数
Table 1 Equations and parameters used for model evaluation in this study

序号No.参数Parameters公式Formula训练集参数 Internal train parameters1决定系数(R2)Coefficient of determination (R2)R2=1-∑ni=1(Yexp-Ypred)2∑ni=1(Yexp-Y—2train)éëêêê〛2标准偏差(SEE)Standard error of estimation (SEE)SEE=(∑ni=1(ε^i)2N-p-1)1/23方差比(F)Critical value (F)F=∑(Ypred -Y—train)2p/∑(Ypred -Yexp)2N-p-1测试集参数 External test parameters4平均绝对误差(MAE)Mean absolute error (MAE)1m∑mi=1|(Yexp-Ypred )|5外部相关系数(Q2F1)External correlation coeffiicient (Q2F1)Q2F1=1-∑mi=1(Yexp-Ypred)2∑mi=1(Yexp-Y—train)2éëêêê〛6外部相关系数(Q2F2)External correlation coeffiicient (Q2F2)Q2F2=1-∑mi=1(Yexp-Ypred)2∑mi=1(Yexp-Y—test)2éëêêê〛7一致性相关系数(CCC)Concordance correlation coefficient (CCC)CCC=2∑mi=1(Yexp-Y—test)(Ypred-Y—test)/YYYYYY=∑mi=1(Yexp-Y—test)2+∑mi=1(Ypred-Y—pred)2+m(Y—test-Y—pred)2
注:Yexp表示有机磷类化合物的实验LD50值;Ypred表示特定模型预测的化合物LD50值;p表示分子描述符的数量;N表示训练集样本数常量;n表示训练集样本数;m表示测试集样本数;i表示第i个样本;![]() 表示训练集实验值的平均值;
表示训练集实验值的平均值;![]() 表示回归残差;
表示回归残差;![]() 表示测试集实验数据的平均值;
表示测试集实验数据的平均值;![]() 表示模型计算数据的平均值;YYY表示假设实验值与模型计算值值独立时的正交平方距离。
表示模型计算数据的平均值;YYY表示假设实验值与模型计算值值独立时的正交平方距离。
Note: Yexp means experimental LD50 data for OPs; Ypred means predictive LD50 data calculated by specific model; p means number of molecular descriptors; N means constant number of samples in training set; n means the number of samples in the training set; m means the number of samples in test set; i means the i-th sample; ![]() means the average value of model computed data; YYY means the orthogonal squared distance assuming the experimental values are independent of the model computed data.
means the average value of model computed data; YYY means the orthogonal squared distance assuming the experimental values are independent of the model computed data.
![]()
(2)
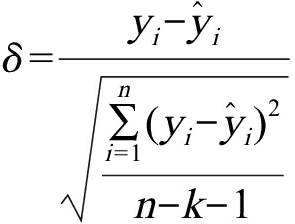
(3)
式中:xi是第i个化合物的分子描述符;X是分子描述符矩阵;k是描述符的数量;yi是模型响应值,即本研究中大鼠急性毒性的logLD50值;![]() 是预测值。
是预测值。
我们先前的实验研究证实了稀有鮈鲫的丁酰胆碱酯酶(BChE)能够被一些OPs显著抑制,从而引起神经毒性[31]。这项研究中,借助分子对接工具探究预测集中9种OPs对哺乳动物类的BChE的结合与抑制能力。从蛋白质数据库中检索到BChE的人类蛋白质结构(PDB ID: 5K5E)作为对接蛋白。通过AutoDockTools4程序进行预处理,去除水分、添加氢并分配电荷。对接网格的大小设置为52 Å×52 Å×52 Å,并使用AutoDock Vina程序[32]进行分子对接。对接前,从BChE蛋白中提取了共结晶化合物并重新对接至原蛋白进行对接程序的验证。最终使用PLIP工具[33]分析了对接后化合物与蛋白质的结合模式。
逐步算法的参数设置为进入F值:<0.010,逐出F值:>0.050。建立的SW-MLR模型如下:
logLD50=-0.154(±1.5150)+0.008(±0.0018)ATS3s-2.783(±0.5247)GATS3e+2.054(±0.4083)MATS8i-88.853(±13.1459)JGI7-2.871(±0.6230)MATS3i+1.257(±0.2907)maxssO-(±0.0553)0.180topoDiameter
遗传算法的参数设置为最大迭代次数:100,方程参数数量:7,初始方程数:100,每代方程数:30,适应函数:MAE。建立的GA-MLR模型如下;
logLD50=5.608(±0.7354)+0.0067(±0.0049)AATSC7m-0.012(±0.0088)ATSC3i+0.0133(±0.0087)VR3_Dzp-3.1038(±0.9454)AVP_4-85.9765(±14.6601)JGI7+0.263(±0.1021)nsssCH-1.2293(±0.3834)GATS4p
对于每个模型还执行了2 000次Y-随机测试。随机测试结果显示SW-MLR和GA-MLR的最大伪R2值分别为0.488和0.504,最大伪Q2值分别为0.299和0.234。所有的伪R2和伪Q2都很低,这表明原始模型的结果并不是偶然关联的。SW-MLR和GA-MLR模型的拟合关系结果如图2所示。
在训练集的统计参数上,SW-MLR模型优于GA-MLR模型,![]() 和F分别为0.879、0.455、0.817和36.248,表明SW-MLR建模方法能够更好地拟合训练集的数据。在测试集的统计参数上,GA-MLR模型优于SW-MLR模型,
和F分别为0.879、0.455、0.817和36.248,表明SW-MLR建模方法能够更好地拟合训练集的数据。在测试集的统计参数上,GA-MLR模型优于SW-MLR模型,![]() 和CCC分别为0.331、0.831、0.828和0.907,表明GA-MLR建模方法有着更强的泛化和预测能力。
和CCC分别为0.331、0.831、0.828和0.907,表明GA-MLR建模方法有着更强的泛化和预测能力。
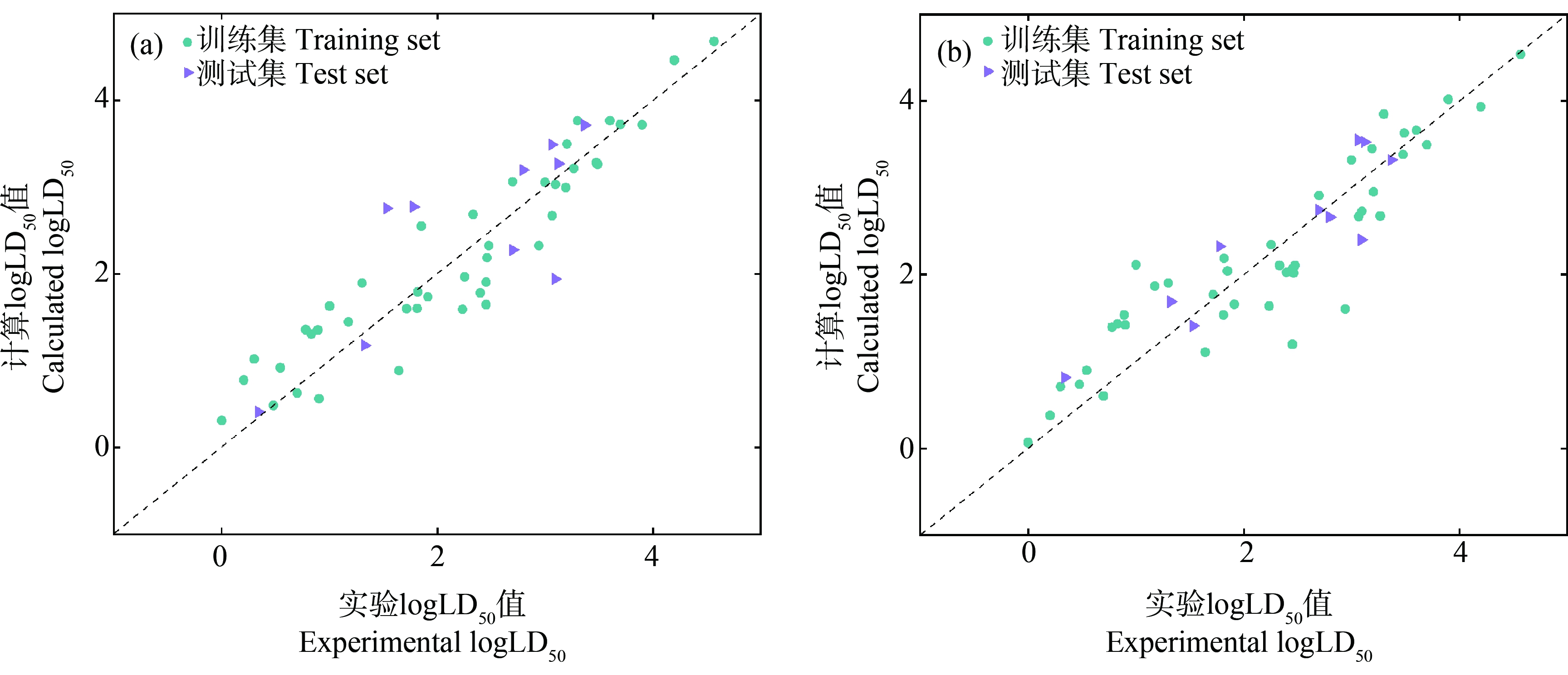
图2 SW-MLR(a)和GA-MLR(b)模型中OPs实验值与预测值拟合关系
Fig. 2 Fitting relationship between experimental and predicted values of OPs in SW-MLR (a) and GA-MLR (b) models
在SW-MLR方程中,分子描述符ATS3s、MATS8i和maxssO与毒性终点logLD50正相关。ATS3s、MATS8i分别是基于原子的内在状态和电离电位来表征化合物拓扑结构的分子描述符[34]。maxssO则代表分子结构中—O—基团的最大电性拓扑状态,前人的研究中也选用maxssO来描述一些无毒的抗疟化合物[35],这与我们研究中maxssO描述符在SW-MLR方程中的正系数一致。分子描述符GATS3e、JGI7、MATS3i和topoDiameter则与毒性终点logLD50负相关。GATS3e和JGI7描述符与化合物中高电负性的原子相关[36-37]。MATS3i和topoDiameter是特定拓扑结构的分子描述符。
在GA-MLR方程中,分子描述符AATSC7m、nsssCH和VR3_Dzp与毒性终点logLD50正相关。AATSC7m和nsssCH分别对应OPs分子结构中R基上支链的长度[38]、支链或取代基烷基化的程度[39]。VR3_Dzp则是比较抽象的二维描述符,代表极化率加权的Barysz矩阵的特征向量[40]。分子描述符ATSC3i、AVP_4、JGI7和GATS4p则与毒性终点logLD50负相关。这4个描述符与分子的疏水性、极性和原子的电负性相关[36,41]。对于2个模型分子描述符的解释从一定程度上揭示了OPs的化学结构与毒性相关的内在因素,对于识别OPs的毒性起到关键作用。
建立的SW-MLR模型和GA-MLR模型应用域表征Willimas图如图3所示。SW-MLR模型中,所有OPs类化合物都很好地落入了应用域范围内。GA-MLR模型中,仅有一种化合物(dimethoate, CAS: 60-51-5, h=0.570>h*=0.558)为离群点。结论表明本研究的2种模型能够较好地预测OPs类化合物对大鼠的急性毒性数据。表2中给出了使用2种QSAR模型预测的没有实验数据的9种OPs的急性毒性结果。
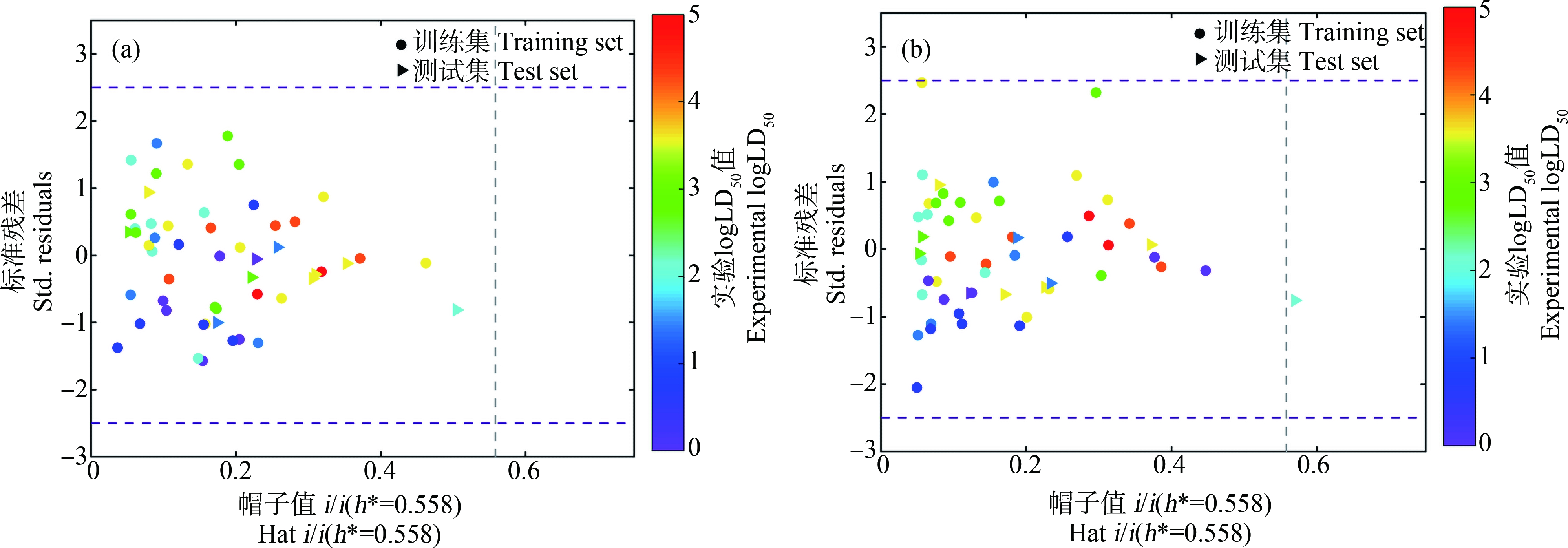
图3 SW-MLR(a)和GA-MLR(b)的Williams应用域图
Fig. 3 Williams plotting for SW-MLR (a) and GA-MLR (b)
模型建立完成后,将研究的模型与其他一些大鼠急性口服毒性的QSAR模型进行了比对。Hanane等[24]建立了高度拟合的人工神经网络![]() 模型,但没有通过外部测试集进行验证。Guo等[42]建立了拟合优度良好的MLR模型
模型,但没有通过外部测试集进行验证。Guo等[42]建立了拟合优度良好的MLR模型![]() 但在研究中对于30个化合物的数据集选用了多达8个的分子描述符(k=8),违反了Topliss-costello规则[25],其模型的良好性能可能来源于偶然关联。与其他研究相比,我们收集了更多关于OPs对大鼠口服急性毒性的数据(n=53),并使用了符合Topliss-costello规则数量的分子描述符
但在研究中对于30个化合物的数据集选用了多达8个的分子描述符(k=8),违反了Topliss-costello规则[25],其模型的良好性能可能来源于偶然关联。与其他研究相比,我们收集了更多关于OPs对大鼠口服急性毒性的数据(n=53),并使用了符合Topliss-costello规则数量的分子描述符![]() 进行模型的构建。构建的2种QSAR方程更具合理性且有更大的应用范围。
进行模型的构建。构建的2种QSAR方程更具合理性且有更大的应用范围。
AutoDock Vina重新对接BChE(5K5E)的原配体的结合能结果为-11.9 kcal·mol-1,均方根偏差(RMSD)为0.89 Å,说明对接程序适用于进一步的对接与研究。使用AutoDock Vina软件对接预测集(9种OPs)与BChE(5K5E)的最低对接能量结果如表2所示。PLIP工具对OPs与BChE的结合模式可视化如图4所示。
AutoDock Vina对接的结果显示其中8个OPs的对接结合能低于阈值[43]-6 kcal·mol-1,表明了潜在的结合能力以及进一步可能造成的神经毒性。此外,取代基或支链烷基化程度较高的化合物(如磷酸三(3-氯丙基)酯)对接结合能也较高,这表明更低的结合能力与更低的神经毒性,这一结果与GA-MLR模型中分子描述符nsssCH的含义一致。因此我们认为OPs的R基及取代基的烷基化能够降低毒性。由图4可知,BChE与OPs结合的关键氨基酸残基有Gly116和Ser198(氢键);His428、Trp332和Phe329(盐桥);Trp231、Phe329、Trp82、Tyr332和His438(π-堆叠)。这些关键氨基酸残基与前人的研究较为一致[44-45]。
研究采用了SW和GA算法,基于PaDEL分子描述符,对OPs化合物的大鼠口服急性毒性建立了多元线性回归QSAR模型,SW-MLR模型具有更好的拟合能力,GA-MLR模型有着更好的预测能力。对模型分子描述符的分析显示了OPs的R基的高烷基化和支链的延长能够降低毒性。此外,通过模型预测了数据集中没有实验数据的9种OPs的毒性,并以分子对接方法检测其与BChE的潜在结合能力。所构建模型能够方便有效地预测OPs类化合物的毒性,并为新化学品的合成与环境保护政策的制定提供帮助。
表2 SW-MLR和GA-MLR对9种OPs的LD50预测值、没有实验值的9种OPs与
丁酰胆碱酯酶(BChE)对接的能量与关键残基
Table 2 Predicted LD50 values by SW-MLR and GA-MLR for nine OPs, docking energy and key
residue results by docking to butyrylcholinesterase (BChE)
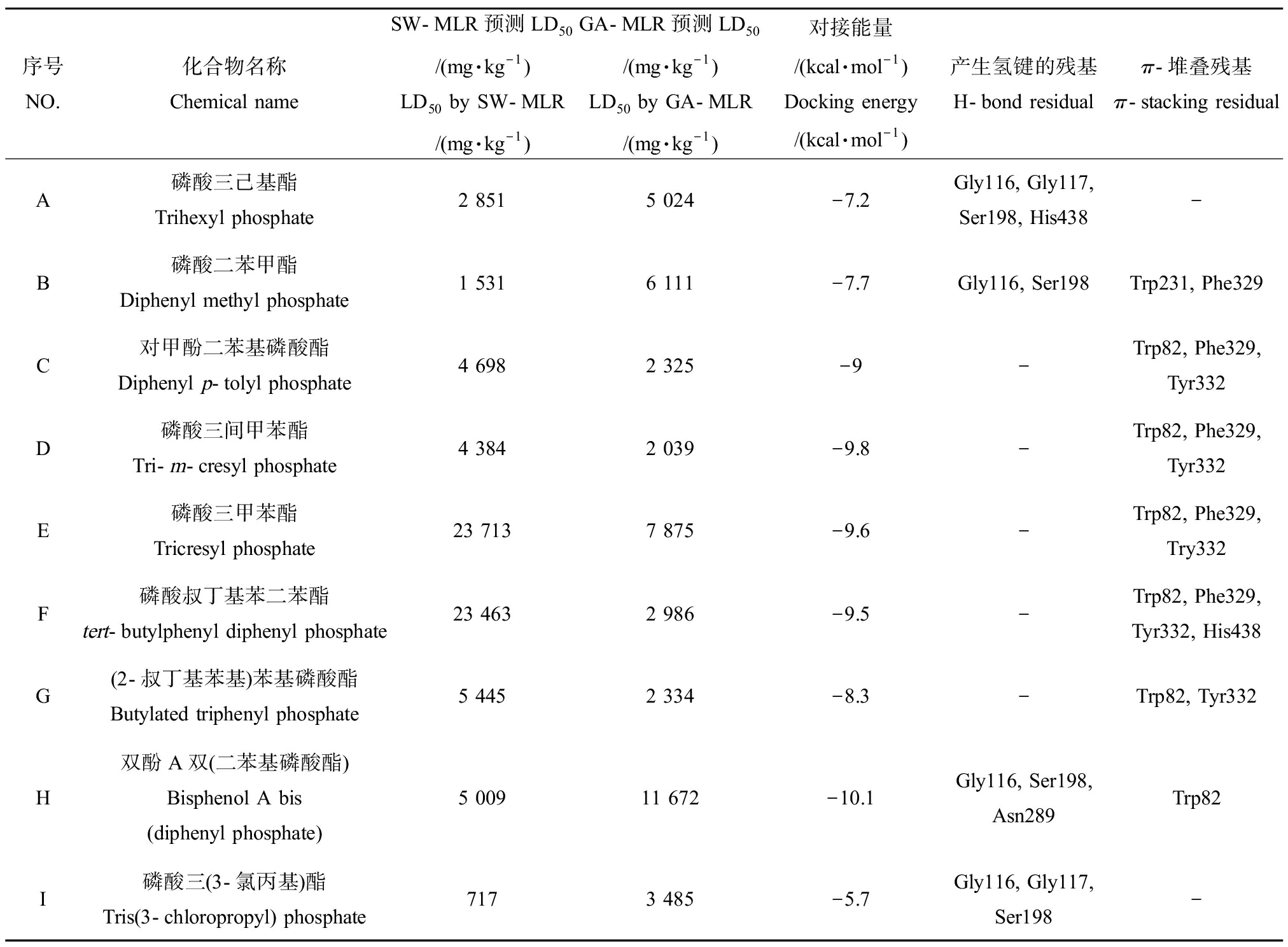
序号NO.化合物名称Chemical nameSW-MLR预测LD50/(mg·kg-1)LD50 by SW-MLR/(mg·kg-1)GA-MLR预测LD50/(mg·kg-1)LD50 by GA-MLR/(mg·kg-1)对接能量/(kcal·mol-1)Docking energy/(kcal·mol-1)产生氢键的残基H-bond residualπ-堆叠残基π-stacking residualA磷酸三己基酯Trihexyl phosphate2 8515 024-7.2Gly116, Gly117, Ser198, His438-B磷酸二苯甲酯Diphenyl methyl phosphate1 5316 111-7.7Gly116, Ser198Trp231, Phe329C对甲酚二苯基磷酸酯Diphenyl p-tolyl phosphate4 6982 325-9-Trp82, Phe329, Tyr332D磷酸三间甲苯酯Tri-m-cresyl phosphate4 3842 039-9.8-Trp82, Phe329, Tyr332E磷酸三甲苯酯Tricresyl phosphate23 7137 875-9.6-Trp82, Phe329, Try332F磷酸叔丁基苯二苯酯tert-butylphenyl diphenyl phosphate23 4632 986-9.5-Trp82, Phe329, Tyr332, His438G(2-叔丁基苯基)苯基磷酸酯Butylated triphenyl phosphate5 4452 334-8.3-Trp82, Tyr332H双酚A双(二苯基磷酸酯)Bisphenol A bis (diphenyl phosphate)5 00911 672-10.1Gly116, Ser198, Asn289Trp82I磷酸三(3-氯丙基)酯Tris(3-chloropropyl) phosphate7173 485-5.7Gly116, Gly117, Ser198-
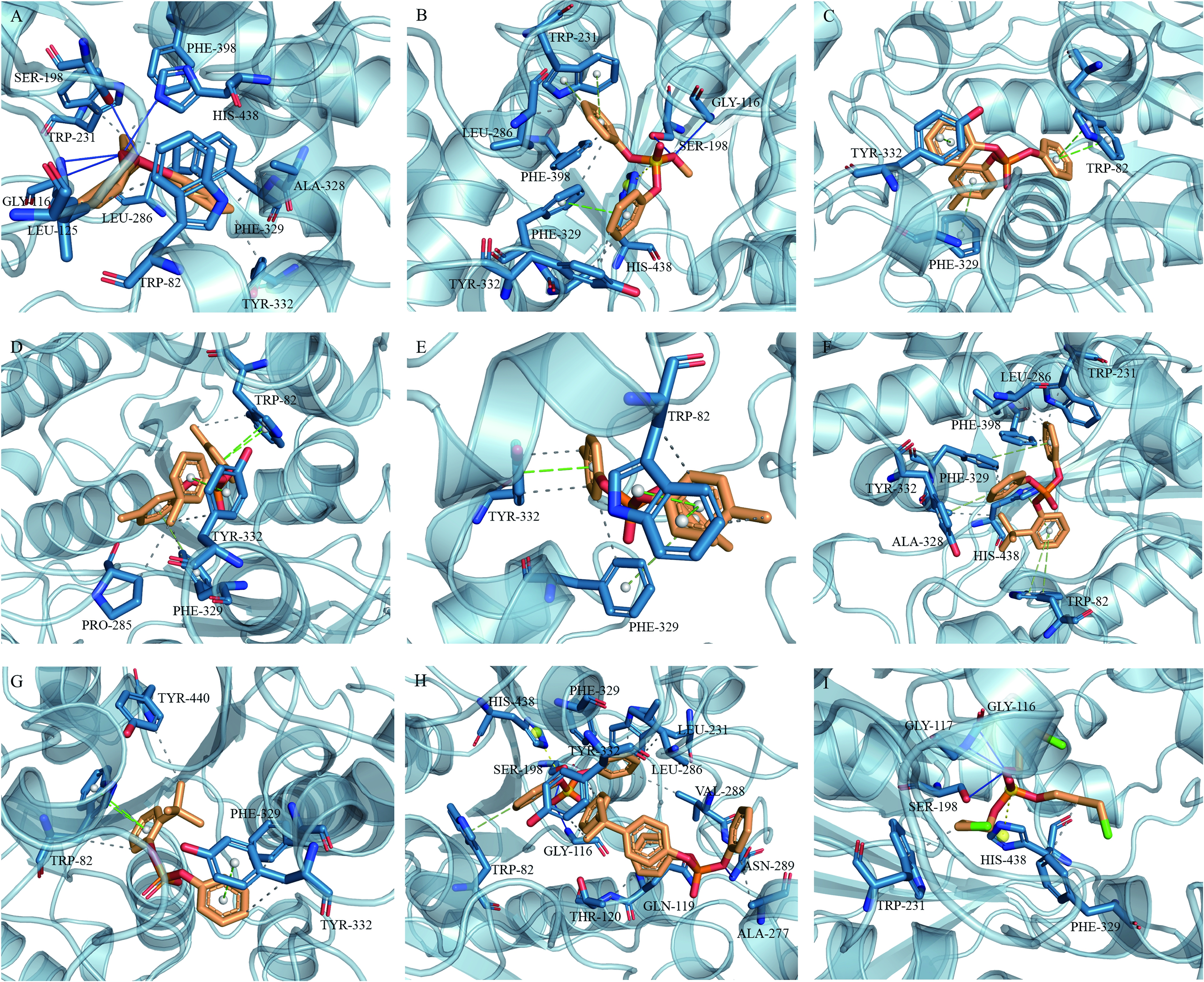
图4 9种OPs与BChE(5K5E)分子对接结果的可视化
注:A~I对应表2中化合物序号;疏水性相互作用为灰色虚线,平行π-堆叠为亮绿色虚线,
垂直π-堆叠为深绿色虚线,氢键为蓝色实线,盐桥为黄色虚线。
Fig. 4 Ligand interaction diagram of the main interactions of nine OPs in the active site of BChE (5K5E)
Note: A~I represents the compounds in Table 2; hydrophobic interactions are represented by gray dashes;
π-stackings are represented by bright green (parallel) and dark green (perpendicular) dashes;
hydrogen bonds are represented by blue solid lines; salt bridges are represented by yellow dashes.
通讯作者简介:查金苗(1975—),男,博士,研究员,主要研究方向为水生模型生物体系的构建与发展、水环境生物毒性测试方法、环境内分泌干扰物的筛选技术研究、环境污染物对水生生物分子毒理机制和水生态系统完整性评估方法等。
共同通讯作者简介:闫赛红(1988—),女,博士,助理研究员,主要研究方向为水生态毒理学。
[1] Pantelaki I, Voutsa D. Organophosphate flame retardants (OPFRs): A review on analytical methods and occurrence in wastewater and aquatic environment [J]. The Science of the Total Environment, 2019, 649: 247-263
[2] Pundir C S, Malik A, Preety. Bio-sensing of organophosphorus pesticides: A review [J]. Biosensors and Bioelectronics, 2019, 140: 111348
[3] Clercq E D. Clinical potential of the acyclic nucleoside phosphonates cidofovir, adefovir, and tenofovir in treatment of DNA virus and retrovirus infections [J]. Clinical Microbiology Reviews, 2003, 16(4): 569-596
[4] Tan X X, Luo X J, Zheng X B, et al. Distribution of organophosphorus flame retardants in sediments from the Pearl River Delta in South China [J]. The Science of the Total Environment, 2016, 544: 77-84
[5] Hou L, Jiang J Y, Gan Z W, et al. Spatial distribution of organophosphorus and brominated flame retardants in surface water, sediment, groundwater, and wild fish in Chengdu, China [J]. Archives of Environmental Contamination and Toxicology, 2019, 77(2): 279-290
[6] Zainuddin A H, Wee S Y, Aris A Z. Occurrence and potential risk of organophosphorus pesticides in urbanised Linggi River, Negeri Sembilan, Malaysia [J]. Environmental Geochemistry and Health, 2020, 42(11): 3703-3715
[7] Kim J W, Isobe T, Muto M, et al. Organophosphorus flame retardants (PFRs) in human breast milk from several Asian countries [J]. Chemosphere, 2014, 116: 91-97
[8] Mangas I, Vilanova E, Estévez J, et al. Neurotoxic effects associated with current uses of organophosphorus compounds [J]. Journal of the Brazilian Chemical Society, 2016, 27(5): 809-825
[![]() M. Neurotoxic effects of organophosphorus pesticides and possible association with neurodegenerative diseases in man: A review [J]. Toxicology, 2018, 410: 125-131
M. Neurotoxic effects of organophosphorus pesticides and possible association with neurodegenerative diseases in man: A review [J]. Toxicology, 2018, 410: 125-131
[10] Yang F W, Zhao G P, Ren F Z, et al. Assessment of the endocrine-disrupting effects of diethyl phosphate, a nonspecific metabolite of organophosphorus pesticides, by in vivo and in silico approaches [J]. Environment International, 2020, 135: 105383
[11] Al-Salem A M, Saquib Q, Siddiqui M A, et al. Organophosphorus flame retardant (tricresyl phosphate) trigger apoptosis in HepG2 cells: Transcriptomic evidence on activation of human cancer pathways [J]. Chemosphere, 2019, 237: 124519
[12] Burke R D, Todd S W, Lumsden E, et al. Developmental neurotoxicity of the organophosphorus insecticide chlorpyrifos: From clinical findings to preclinical models and potential mechanisms [J]. Journal of Neurochemistry, 2017, 142(Suppl.2): 162-177
[13] Zhang Q, Lu M Y, Dong X W, et al. Potential estrogenic effects of phosphorus-containing flame retardants [J]. Environmental Science & Technology, 2014, 48(12): 6995-7001
[14] Zhang Q, Wang J H, Zhu J Q, et al. Potential glucocorticoid and mineralocorticoid effects of nine organophosphate flame retardants [J]. Environmental Science & Technology, 2017, 51(10): 5803-5810
[15] Busquet F, Strecker R, Rawlings J M, et al. OECD validation study to assess intra- and inter-laboratory reproducibility of the zebrafish embryo toxicity test for acute aquatic toxicity testing [J]. Regulatory Toxicology and Pharmacology, 2014, 69(3): 496-511
[16] 于洋, 郑玉婷, 张丽丽, 等. 面向我国化学物质环境管理的计算毒理学模型评估思路及建设路径[J]. 生态毒理学报, 2021, 16(5): 24-32
Yu Y, Zheng Y T, Zhang L L, et al. Evaluation thoughts of computational toxicological models and constructional path for environmental management of chemicals in China [J]. Asian Journal of Ecotoxicology, 2021, 16(5): 24-32 (in Chinese)
[17] Brad R, Mayeno A N. What is computational toxicology? [J]. Methods in Molecular Biology, 2012, 929: 3-7
[18] Schultz T W, Cronin M T D, Walker J D, et al. Quantitative structure-activity relationships (QSARs) in toxicology: A historical perspective [J]. Journal of Molecular Structure: THEOCHEM, 2003, 622(1-2): 1-22
[19] Vilar S, Cozza G, Moro S. Medicinal chemistry and the molecular operating environment (MOE): Application of QSAR and molecular docking to drug discovery [J]. Current Topics in Medicinal Chemistry, 2008, 8(18): 1555-1572
[20] Alves V M, Bobrowski T, Melo-Filho C C, et al. QSAR modeling of SARS-CoV Mpro inhibitors identifies sufugolix, cenicriviroc, proglumetacin, and other drugs as candidates for repurposing against SARS-CoV-2 [J]. Molecular Informatics, 2021, 40(1): e2000113
[21] Jeon H K, Sarma S N, Kim Y J, et al. Toxicokinetics and metabolisms of benzophenone-type UV filters in rats [J]. Toxicology, 2008, 248(2-3): 89-95
[22] Zou X M, Lin Z F, Deng Z Q, et al. The joint effects of sulfonamides and their potentiator on Photobacterium phosphoreum: Differences between the acute and chronic mixture toxicity mechanisms [J]. Chemosphere, 2012, 86(1): 30-35
[23] Li F, Xie Q, Li X H, et al. Hormone activity of hydroxylated polybrominated diphenyl ethers on human thyroid receptor-beta: in vitro and in silico investigations [J]. Environmental Health Perspectives, 2010, 118(5): 602-606
[24] Hanane F, Taoufiq F, Mohamed M. Evaluation of the model prediction toxicity (LD50) for series of 42 organophosphorus pesticides [J]. Journal of Engineering Studies and Research, 2019, 25(1): 30-35
[25] Topliss J G, Edwards R P. Chance factors in studies of quantitative structure-activity relationships [J]. Journal of Medicinal Chemistry, 1979, 22(10): 1238-1244
[26] Knaak J B, Dary C C, Power F, et al. Physicochemical and biological data for the development of predictive organophosphorus pesticide QSARs and PBPK/PD models for human risk assessment [J]. Critical Reviews in Toxicology, 2004, 34(2): 143-207
[27] Liagkouridis I, Cousins A P, Cousins I T. Physical-chemical properties and evaluative fate modelling of ‘emerging’ and ‘novel’ brominated and organophosphorus flame retardants in the indoor and outdoor environment [J]. Science of the Total Environment, 2015, 524-525: 416-426
[28] Yap C W. PaDEL-descriptor: An open source software to calculate molecular descriptors and fingerprints [J]. Journal of Computational Chemistry, 2011, 32(7): 1466-1474
[29] Ambure P, Aher R B, Gajewicz A, et al. “NanoBRIDGES” software: Open access tools to perform QSAR and nano-QSAR modeling [J]. Chemometrics and Intelligent Laboratory Systems, 2015, 147: 1-13
[30] Gramatica P. Principles of QSAR models validation: Internal and external [J]. QSAR & Combinatorial Science, 2007, 26(5): 694-701
[31] Yuan L L, Li J S, Zha J M, et al. Targeting neurotrophic factors and their receptors, but not cholinesterase or neurotransmitter, in the neurotoxicity of TDCPP in Chinese rare minnow adults (Gobiocypris rarus) [J]. Environmental Pollution, 2016, 208(Pt B): 670-677
[32] Trott O, Olson A J. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading [J]. Journal of Computational Chemistry, 2010, 31(2): 455-461
[33] Adasme M F, Linnemann K L, Bolz S N, et al. PLIP 2021: Expanding the scope of the protein-ligand interaction profiler to DNA and RNA [J]. Nucleic Acids Research, 2021, 49(W1): W530-W534
[34] Hollas B. An analysis of the autocorrelation descriptor for molecules [J]. Journal of Mathematical Chemistry, 2003, 33(2): 91-101
[35] Ibrahim Z Y, Uzairu A, Abechi S. Quantum Modelling of Some Potent, Non-Toxic Antimalarial Compounds [M]. Chisinau: Scholar Press, 2019: 161-166
[36] Galvez J, Garcia R, Salabert M T, et al. Charge indexes. New topological descriptors [J]. Journal of Chemical Information and Computer Sciences, 1994, 34(3): 520-525
[37] Caballero J, Garriga M, Fernández M. 2D autocorrelation modeling of the negative inotropic activity of calcium entry blockers using Bayesian-regularized genetic neural networks [J]. Bioorganic & Medicinal Chemistry, 2006, 14(10): 3330-3340
[38] Adedirin O, Uzairu A, Shallangwa G A, et al. QSAR and molecular docking based design of some n-benzylacetamide as γ-aminobutyrate-aminotransferase inhibitors [J]. The Journal of Engineering and Exact Sciences, 2018, 4(1): 65-84
[39] Shi Q P, Guo W, Shen Q C, et al. In vitro biolayer interferometry analysis of acetylcholinesterase as a potential target of aryl-organophosphorus flame-retardants [J]. Journal of Hazardous Materials, 2021, 409: 124999
[40] Todeschini R, Consonni V. Molecular Descriptors for Chemoinformatics: Volume Ⅰ: Alphabetical Listing/Volume Ⅱ: Appendices, References [M]. John Wiley & Sons, 2009: 41
[41] García-Domenech R, Galvez J, de Julian-Ortiz J V, et al. Some new trends in chemical graph theory [J]. Chemical Reviews, 2008, 108(3): 1127-1169
[42] Guo J X, Wu J J, Wright J B, et al. Mechanistic insight into acetylcholinesterase inhibition and acute toxicity of organophosphorus compounds: A molecular modeling study [J]. Chemical Research in Toxicology, 2006, 19(2): 209-216
[43] Shityakov S, Förster C. In silico predictive model to determine vector-mediated transport properties for the blood-brain barrier choline transporter [J]. Advances and Applications in Bioinformatics and Chemistry, 2014, 7: 23-36
[44] Bennion B J, Lau E Y, Fattebert J L, et al. Modeling the binding of CWAs to AChE and BuChE [J]. Military Medical Science Letters, 2013, 82(3): 102-114
[45] Jończyk J, Kuku owicz J,
owicz J,  ątka K, et al. Molecular modeling studies on the multistep reactivation process of organophosphate-inhibited acetylcholinesterase and butyrylcholinesterase [J]. Biomolecules, 2021, 11(2): 169
ątka K, et al. Molecular modeling studies on the multistep reactivation process of organophosphate-inhibited acetylcholinesterase and butyrylcholinesterase [J]. Biomolecules, 2021, 11(2): 169
Corresponding author), E-mail: jmzha@rcees.ac.cn
# 共同通讯作者(Co-corresponding author), E-mail: shyan@rcees.ac.cn