原生生物是由单细胞组成的微生物[1],分布广泛,以细菌、藻类等为食,并被后生生物等捕食,在水生食物网中占有重要地位[2]。而且,部分原生生物以水体中的碎屑或腐殖质为食,担任着净化水体的重要功能,是食物链中不可或缺的一环[3]。原生生物生长周期短,对水环境变化极为敏感,是水体富营养化重要的指示生物之一。例如富营养的湖泊与水质较好的湖泊中纤毛虫的丰度存在巨大差异,在富营养化湖泊中纤毛虫丰度更高[4]。
目前,原生生物监测主要基于自微型生物群落监测方法(PFU法)[5]。将泡沫塑料块放入监测的水体中富集原生生物,带回实验室后使用显微镜人工观察原生生物群落的组成,判断监测水体区域的受污染状况。虽然此方法操作易上手、成本较低,但是重污染地区原生生物聚集较慢,影响评估结果[6],具有一定的局限性。监测沉积物中原生生物则需要将环境样品长时间风干,使用培养皿对孢子(原生生物在干旱条件下存活方式)进行培养[7],在显微镜下人工计数需要耗费大量时间与精力且保存不易。同时原生生物易受到环境因子的影响而改变自身形态这一特性对人工鉴定造成了很大的困难[8]。
环境DNA(eDNA)宏条形码技术为监测沉积物原生生物多样性提供了新的方法。生物参与生态过程时,会向环境中释放含有DNA的分泌物,例如血液、粪便、脱落的组织结构等[9]。这些游离于环境中的生物DNA片段被称为环境DNA[10]。随着测序技术发展,高通量测序(high-throughput sequencing)因其可以一次性将数据量巨大的DNA条形码碱基信息进行分类,使获取环境中物种DNA信息成为了可能[11]。结合聚合酶链式反应技术(PCR)可以识别环境中存在的低丰度的物种信息[12]。该技术最大的特点是能够将环境中微量存在的DNA条形码碱基序列片段几何数量级的扩增,从而获得环境中生物多样性的信息[13]。eDNA宏条形码比传统方法具有更高的敏感度[14],不需要目标物种在采样位点被观测到[15]。eDNA技术检测物种不直接对生物体进行采样,具有对稀有和难以捕捉物种的实地调查能力[16]。目前环境DNA宏条形码技术已经被用于监测鱼类、浮游等水生生物中[17],但在我国湖泊沉积物研究中尚不多见。因此本项目拟以太湖流域为对象:(1)建立基于环境DNA宏条形码技术监测沉积物原生生物多样性的方法;(2)评估沉积物原生生物完整性指数与水生态健康的关系。
1 材料与方法(Materials and methods)
1.1 研究区域
太湖流域的河流、水库和湖荡等设置82个点位,每个点位一共采取3份等体积沉积物样本,共采集246份样品。分别在2019年3月和8月采集沉积物用于原生生物群落分析,采样位点图如图1所示。
1.2 样品的采集与处理
每个点位用尺寸为300 mm×208 mm×150 mm(长×宽×高)的皮特森抓泥器采集4次,抓取表层深度约5 cm的沉积物,累计采集0.125~0.25 m2,采样过程中避免水体的扰动。将底泥混匀后取50 g装入离心管中。每个位点采集3个平行样品。现场用干冰保存,实验室内用-80 ℃冰箱保存。
1.3 实验方法
用冻干机将样品中的水分冻干后拍打混匀,每份样品取约0.3 mg,使用QIAGEN power soil kit 100试剂盒提取沉积物中DNA。使用18S RNA V9引物(上游引物序列CCTTCYGCAGGTTCACCTAC,下游引物序列TCCCTGCCHTTTGTACACAC)进行PCR扩增。PCR在96孔板中进行,每个96孔板均设有3个阴性对照(无菌水)。PCR产物用1.5%的琼脂糖凝胶电泳检测是否存在非特异性扩增。PCR反应体系如下:2×Vazyme taq酶12.5 μL、上游引物0.5 μL、下游引物0.5 μL、DNA样品1 μL、无酶水10.5 μL。反应条件为:(1)在95 ℃条件下预变性3 min;(2)95 ℃变性30 s;(3)62 ℃退火30 s;(4)72 ℃延伸15 s循环;(5)重复2到4步骤循环28次;(6)末次延伸72 ℃ 5 min。

图1 采样位点分布图
Fig. 1 Sampling sites map
PCR产物利用Ion torrent Proton平台进行高通量测序[18]。在EcoView软件中去除Q<20的序列,保留读长130~170 bp的序列;使用UCHIME软件筛查嵌合子序列信息,避免嵌合子污染去除重复、低频序列,保留unique序列并排除测序过程中的随机误差;使用USEARCH程序类聚相似度97%以上的分类单元(OTU),类聚阈值参考18S rRNA基因通用类聚标准;在Protist Ribosomal Reference database(PR2)数据库(版本号4.14.0)进行物种信息注释,并将注释为古菌、细菌、叶绿体、线粒体和未知的序列去除,注释结果从属关系混乱的物种按照域界门纲目科属种顺序重新划分,3份平行样本中物种OTU出现2次及以上保留,数据四舍五入取整数均值。以上操作在Linux系统中完成。基于物种OTU水平、门水平和属水平上分析eDNA技术区分物种alpha-多样性的季节差异,使用T值检验来判定;基于OTU与属水平上分析beta-多样性不同季节的差异,将OTU注释文件在R语言中进行数据统计分析。
1.4 指数计算方法
基于OTU分类单元水平与属水平分析,使用R语言Vegan包计算alpha-多样性与beta-多样性。alpha-多样性计算公式如下。
丰富度(Richness)指数计算公式:Richness即为OTU分类单元数
香农(Shannon)指数计算公式:H=-∑(Pi)×(ln Pi),Pi代表个体数与总体数之比
均匀度(Pielou)指数计算公式:![]() 为均匀度指数,H为香农指数,S代表物种数
为均匀度指数,H为香农指数,S代表物种数
基于OTU分类单元水平与属水平计算方法重复性指数,公式如下所示[19]:
3个平行样本OTU的交叉率=3×共有的OTU数目/(平行1的OTU数目+平行2的OTU数目+平行3的OTU数目)。
考虑权重的OTU交叉率=(平行1共有的序列数+平行2共有的序列数+平行3共有的序列数)/(平行1的序列数+平行2的序列数+平行3的序列数)。
至少2个平行样本出现的OTU交叉率=(2×只在2个平行中出现的OTU数目+3×3个平行共有的OTU数目)/(平行1的OTU数目+平行2的OTU数目+平行3的OTU数目)。
1.5 原生生物完整性评估
根据上述步骤得到的数据构建原生生物完整性指数,该方法主要是筛选对环境干扰有明显变化的原生生物多样性指数来定量描述生物特性与环境影响因子的关系。首先根据原生生物特征筛选指标;计算出多样性指数的分布并做相关性分析筛选合适的指数;计算原生生物指标值以及确定生物完整性指数的计算方法;确定评价标准。按照以下步骤进行构建:首先确定参考点与受损点,在以往的研究中水库位点常被认为是生境较好的地带,太湖流域北部的湖泊与河流均长期处于水污染影响的状态,水质处于中国地表水标准3类水以下,故参考点选取水库位点,受损点选取太湖北部地区位点,本研究取T1、T2、T3和T6水库位点为参考点,取T46、T48、T72和T75河流位点为受损点,取相同位点3份平行样本中多样性指数均值计算原生生物完整性指数。随着参考点与受损点变化,能够明显区分参考点与受损点的参数称为显著性差异指数,呈现上升趋势的以5%分位数作为参考值,下降趋势的以95%分位数作为参考值。统一的参数量纲分值计算:下降的参数分数为参考值/参数值;上升的参数分数为(最大值-参考值)/(最大值-参考值)。最后统一进行分数汇总,按照25%、50%和75%人为划分太湖流域生态健康状态等级。
2 结果(Results)
2.1 原生生物群落组成
利用环境DNA技术检测出82个位点共有6 215个OTU,其中原生生物有2 468个OTU,占总序列数的13%(图2),隶属13门44纲(表1)。其中有70%的分类单元能注释到种,79%的分类单元能注释到属,91%的分类单元能注释到科,97%的分类单元能注释到目,98%的分类单元能注释到纲,99%的分类单元能注释到门。OTU序列数优势类群为纤毛虫门、丝足虫门、锥足亚门、甲藻门和顶复门等(图2),其中纤毛虫门分类单元占原生生物总分类单元的37%,丝足虫门分类单元占原生生物总分类单元的30%,锥足亚门分类单元占原生生物总分类单元的10%,甲藻门分类单元占原生生物总分类单元的9%,顶复门分类单元占原生生物总分类单元的5%。
2.2 方法重复性研究
选取2次采样位点中97个位点的样本,样本包含2个季度采样重合位点,每个位点样本包含3个平行样本,共计291个样本。
基于OTU计算与基于属多样性指数CV值分布绝大部分均处于0.15以下,可知环境DNA宏条形码技术具有很好的方法重复性。随机抽取1个点位(共3份平行样本)的方法重复性研究示意图如图3所示。3个平行样本均检出的OTU占总OTU的21.7%,至少在2个平行样本检出的OTU占总OTU的47%。若考虑序列权重,3个平行样本OTU交叉率达到63.50%,至少在2个平行样本检出的OTU所占比例达到84.25%。3个平行样本出现的属平均值的交叉率平均值为37.70%,至少在2个平行样本出现的属交叉率均值为62.15%。基于权重3个平行样本出现的属交叉率均值为91.33%,基于权重至少在2个平行样本出现的属交叉率均值为97.13%。基于OTU的多样性指数丰富度CV值均值为12.5%;香农指数CV值均值为4.9%;均匀度CV值均值为4.5%。基于属的多样性指数丰富度CV值均值为9.3%;香农指数CV值均值为4%;均匀度CV值均值为3.7%,alpha-多样性CV值分布如图4所示。

图2 原生生物OTU分布
Fig. 2 Distribution of OTU in protist
表1 物种注释表
Table 1 Taxa annotation table

界Kingdom门Phylum纲Class囊泡虫Alveolata顶复门Apicomplexa纤毛虫门CiliophoraApicomplexaArmophoreaCiliophora-6Ciliophora-8Colpodea异毛纲Heterotrichea 核残迹纲Karyorelictea LitostomateaNassophorea寡膜纲Oligohymenophorea 叶咽纲Phyllopharyngea Plagiopylea前口类Prostomatea 旋毛纲Spirotrichea甲藻门 Dinophyta PerkinseaDinophyceae甲藻纲Syndiniales Perkinsida变形虫门Amoebozoa Breviatea锥足亚门Conosa 变形虫亚门 Lobosa Breviatea始变形虫下门Archamoebea 黏菌纲Mycetozoa-Myxogastrea 叶足亚门Variosea Discosea-FlabelliniaDiscosea-LongamoebiaLobosaLobosa-G1变形虫纲Tubulinea无根虫门ApusozoaApusomonadidaeHilomonadeaApusomonadidae_Group-1Planomonadida古虫界Excavata盘嵴亚界 Discoba 眼虫门Euglenozoa渗养门Heterolobosea 雅各巴虫Jakobida后滴门 MetamonadaParabasalia副基体纲Preaxostyla 有孔虫界Rhizaria丝足虫门 Cercozoa EndomyxaEndomyxa-PhytomyxeaFilosaFilosa-GranofiloseaFilosa-ImbricateaFilosa-SarcomonadeaFilosa-ThecofiloseaNovel-CladeGlobothalamea有孔虫门 Foraminifera Monothalamids

图3 方法重复性研究示意图
注:A1~A3代表相同位点的3个平行样本。
Fig. 3 Schematic diagram of method repeatability study
Note: A1~A3 represent three parallel samples of the same samples.
2.3 原生生物群落alpha-多样性季节差异
原生生物alpha-多样性在不同季节存在显著差异。对比2个季度的丰富度指数、香农指数、均匀度指数、分类单元数优势物种门相对丰度,基于OTU分类单元水平与属水平进行分析。T值检验分析整体性差异结果显示,基于OTU与基于属计算的丰富度指数在时间尺度上具有显著差异,而香农指数与均匀度则未见显著性差异(图5)。基于OTU计算的原生生物优势物种丰富度指数,在不同季节中具有显著差异类群为纤毛虫门、甲藻门、顶复门和盘嵴亚界(图6)。
2.4 原生生物群落beta-多样性
进行春季(3月)和秋季(8月)原生生物群落主成分(PCA)分析,区分时间尺度上群落结构的变化。不同季节原生生物群落结构差异不显著(图7)。
2.5 利用原生生物完整性指数评价水生态健康
不同季节指标筛选:本研究筛选了2个季度15个评价参数(表2)。甲藻门丰富度、甲藻门香农指数、甲藻门相对丰度和纤毛虫相对丰度4个指数在3月的参考点与受损点之间具有显著性差异;纤毛虫香农指数、丝足虫丰富度、基于OTU的香农指数和基于OTU的丰富度在8月的参考点与受损点之间存在显著性差异(图8)。
3月的各类指数中,参考点与受损点之间甲藻门丰富度、甲藻门香农指数和甲藻门相对丰度呈下降趋势,纤毛虫相对丰度呈上升趋势。8月的各类指数中,参考点与受损点之间基于OTU的丰富度、香农指数、丝足虫丰富度和纤毛虫香农指数均呈下降趋势。生物完整性指数校正表如表3所示,春季(3月)与秋季(8月)水生态健康评分标准如表4所示。评价结果显示,3月与8月采样位点生态健康等级具有一致性(图9)。

图4 alpha-多样性指数CV值
Fig. 4 CV value of alpha diversity index
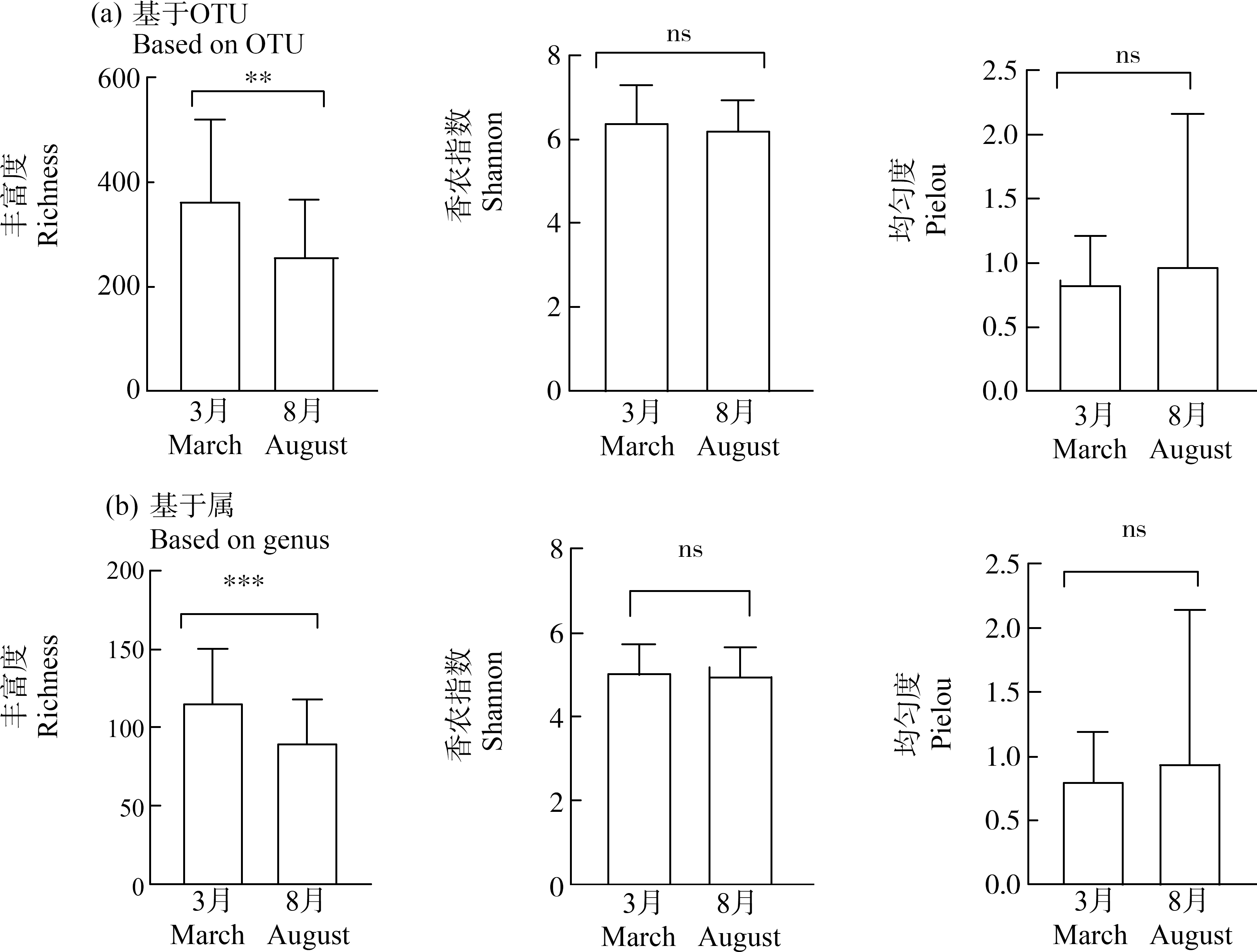
图5 多样性指数非独立样本T值检验
注:**代表P<0.001;***代表P<0.0001;ns表示不显著。
Fig. 5 T value test of non independent samples based on OTU diversity index
Note: **represents P<0.001; ***represents P<0.0001; ns represents not significant.
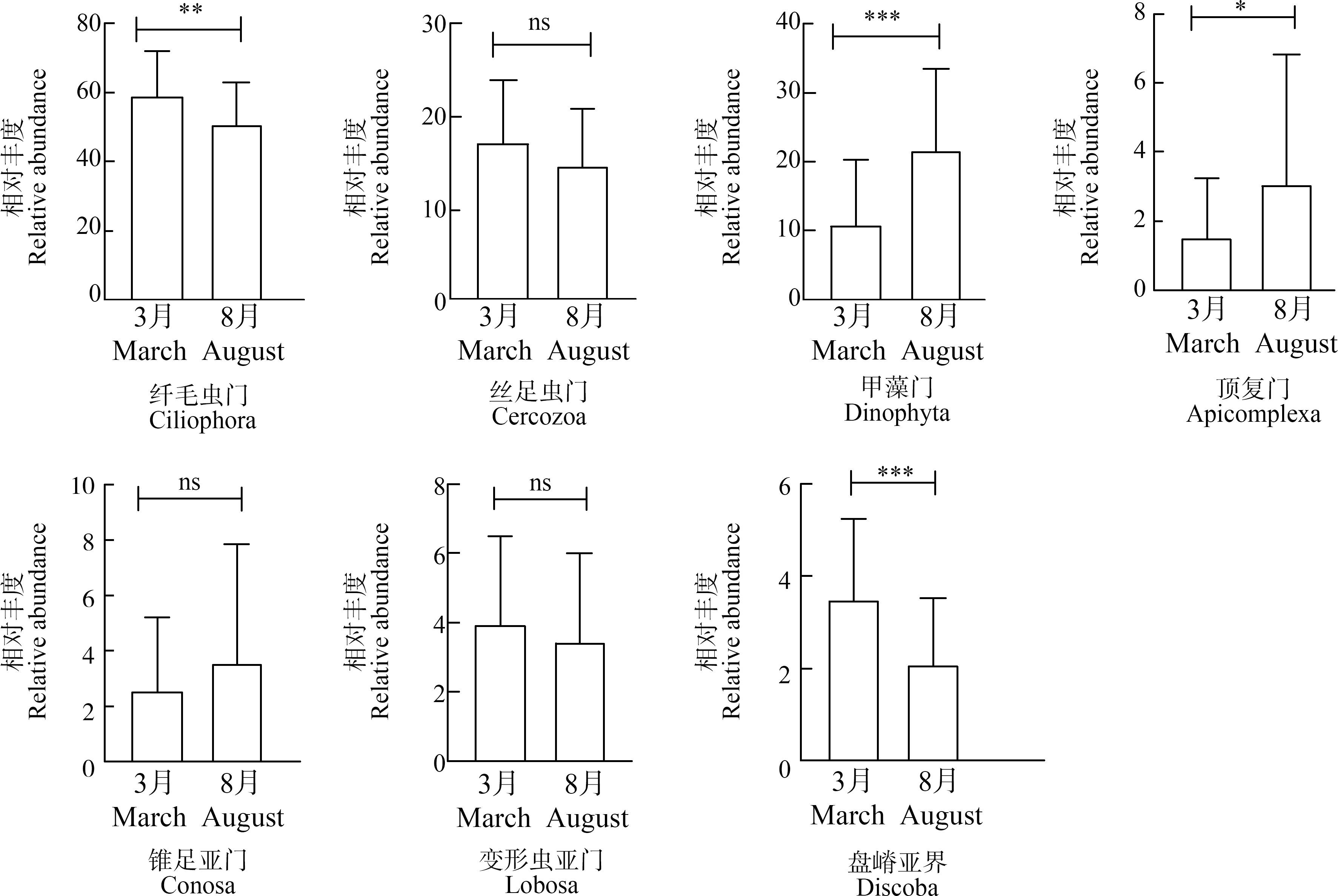
图6 不同采样季节对原生生物优势物种的影响
注:*代表P<0.05,**代表P<0.001,***代表P<0.0001;ns表示不显著。
Fig. 6 Effects of different sampling seasons on dominant protist species
Note: *represents P<0.05; **represents P<0.001; ***represents P<0.0001; ns represents not significant.
表2 指数筛选表
Table 2 Index screening table
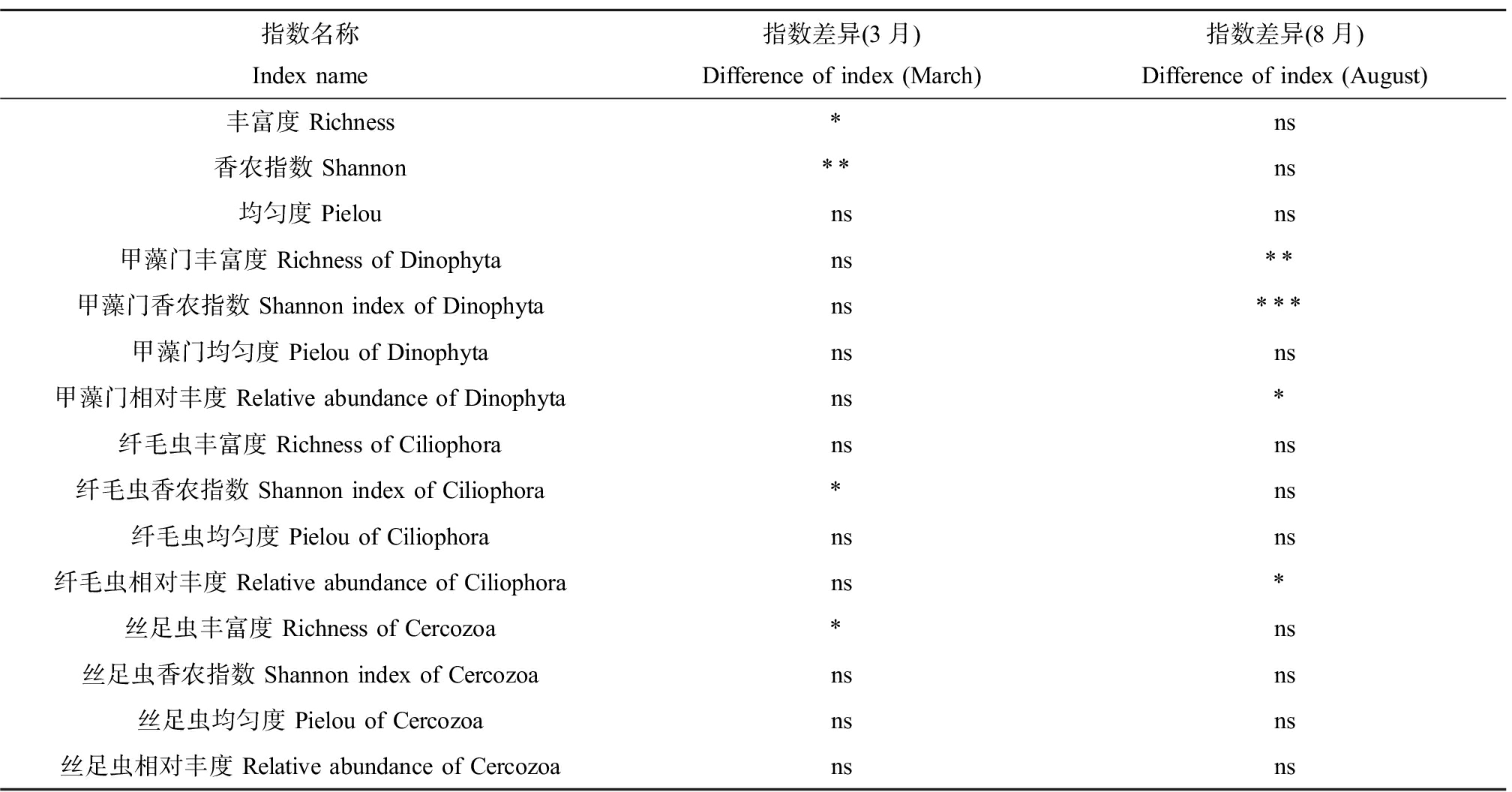
指数名称Index name指数差异(3月)Difference of index (March)指数差异(8月)Difference of index (August)丰富度 Richness*ns香农指数 Shannon**ns均匀度 Pielounsns甲藻门丰富度 Richness of Dinophytans**甲藻门香农指数 Shannon index of Dinophytans***甲藻门均匀度 Pielou of Dinophytansns甲藻门相对丰度 Relative abundance of Dinophytans*纤毛虫丰富度 Richness of Ciliophoransns纤毛虫香农指数 Shannon index of Ciliophora*ns纤毛虫均匀度 Pielou of Ciliophoransns纤毛虫相对丰度 Relative abundance of Ciliophorans*丝足虫丰富度 Richness of Cercozoa*ns丝足虫香农指数 Shannon index of Cercozoansns丝足虫均匀度 Pielou of Cercozoansns丝足虫相对丰度 Relative abundance of Cercozoansns
注:*表示差异显著。
Note: *represents significant.
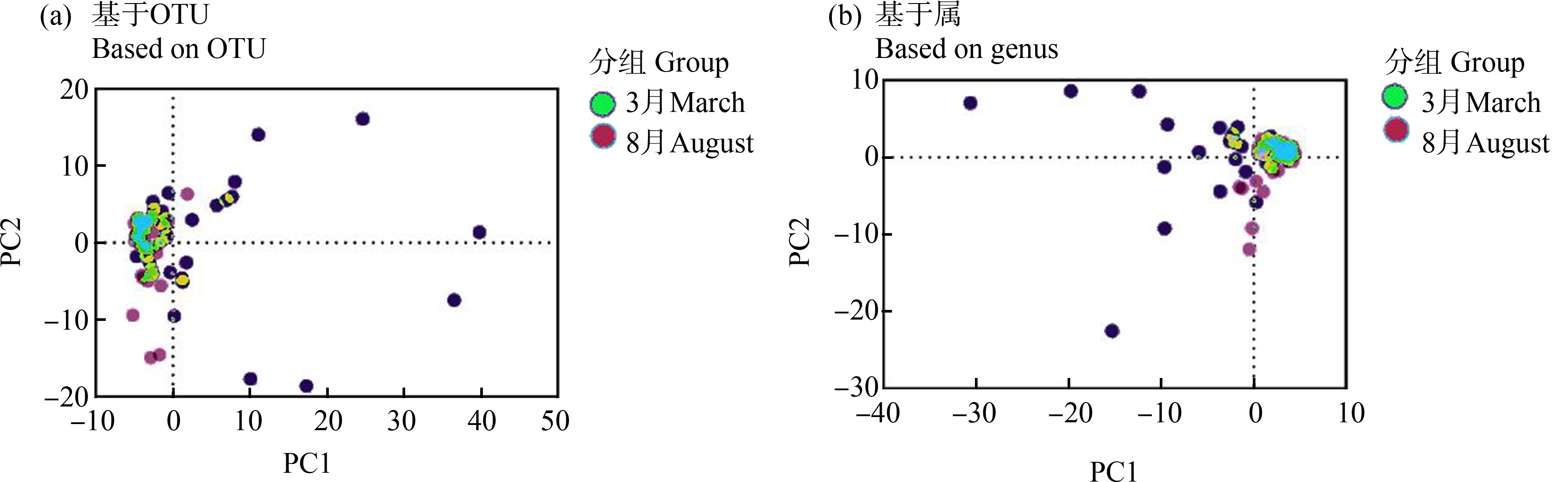
图7 原生生物群落PCA分析
Fig. 7 PCA analysis of protist community
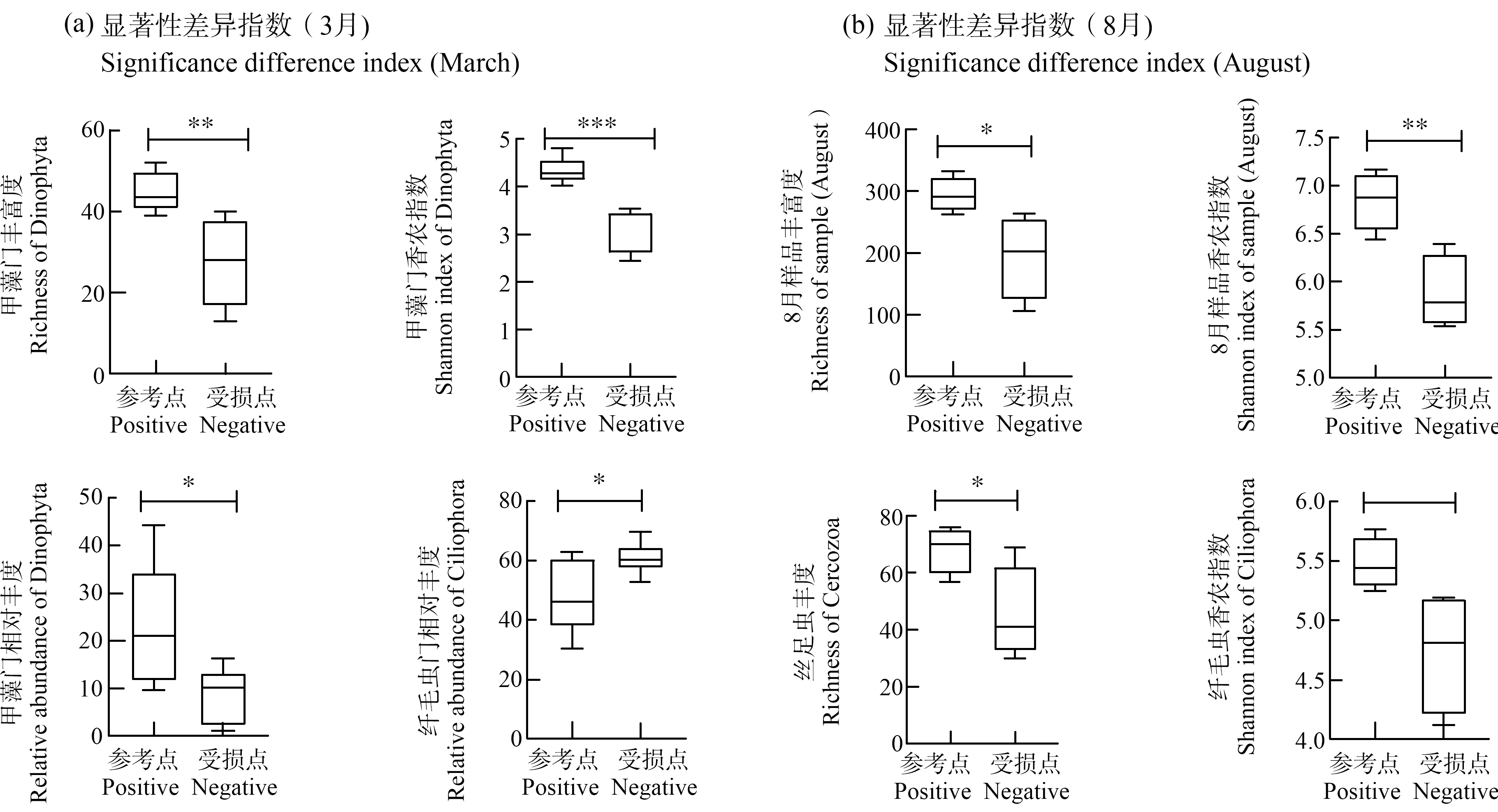
图8 原生生物完整性差异指数
Fig. 8 Protist community index screening
表3 指数校正表
Table 3 Index correction table

指数名称 Index name校正公式 Updating formula生物多样性指数(3月) Biodiversity index (March)甲藻门丰富度 Richness of Dinophyta(52-49)/(42-X)甲藻门香农指数 Shannon index of Dinophyta(4.808-4.479)/(4.808-X)甲藻门相对丰度 Relative abundance of Dinophyta(0.439-0.307)/(0.439-X)纤毛虫相对丰度 Relative abundance of Ciliophora30.53%/X生物多样性指数(8月) Biodiversity index (August)丰富度 Richness(396-379)/(396-X)香农指数 Shannon index(7.25-7.17)/(7.25-X)纤毛虫香农指数 Shannon index of Ciliophora(6.36-5.92)/(6.36-X)丝足虫丰富度 Richness of Cercozoa(102-95)/(102-X)
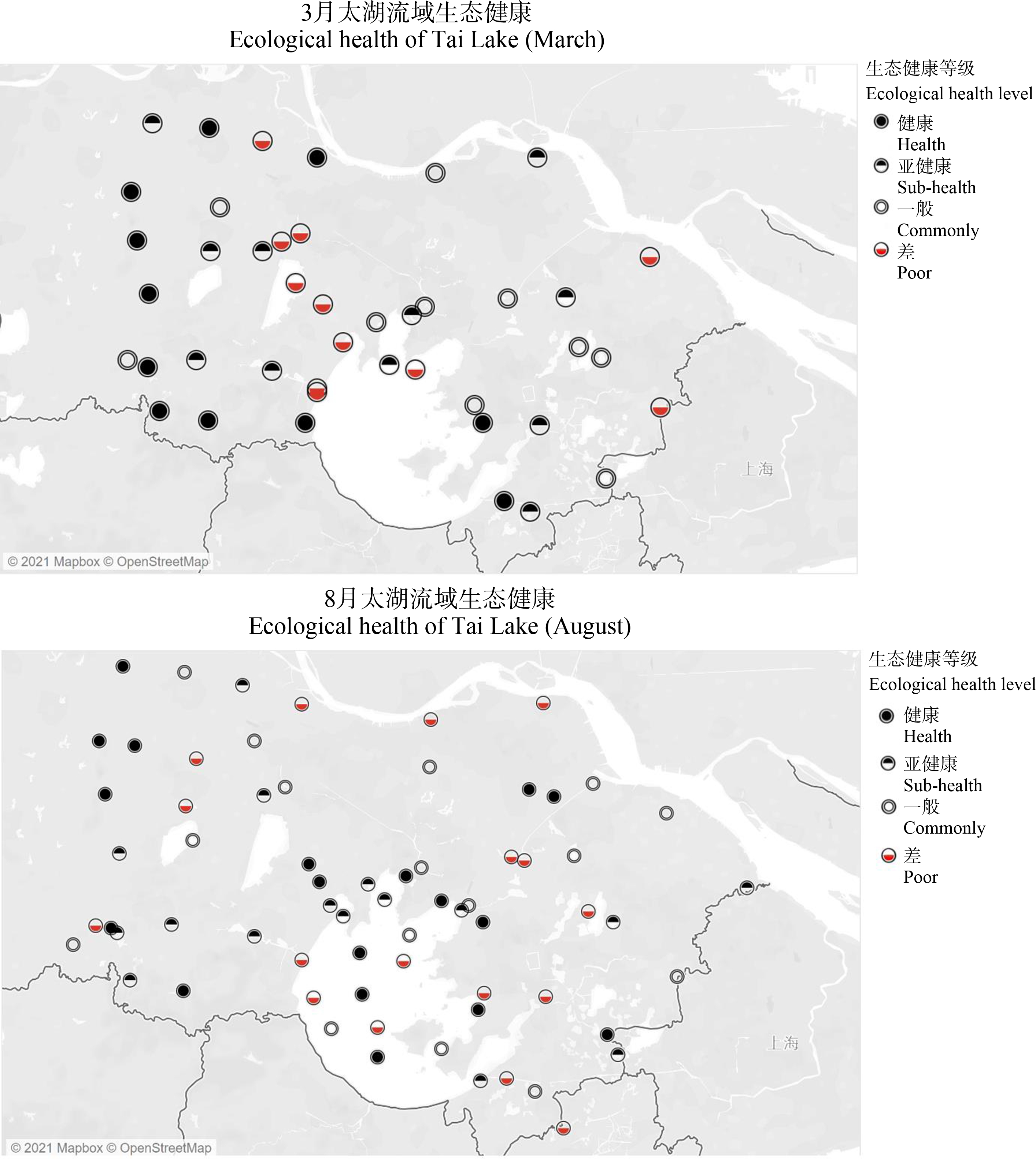
图9 太湖流域生态健康等级分布
Fig. 9 Distribution of ecological health grades in Tai Lake Basin
表4 原生生物完整性指数标准
Table 4 Integrity index standard of benthic protist

百分位数Percentile健康状态Health status完整性指数范围(3月)Integrity index range (March)完整性指数范围(8月)Integrity index range (August)>75%健康 Health>1.436>0.9250%~75%亚健康 Sub-health1.3~1.4360.675~0.9225%~50%一般 Commonly1.14~1.30.47~0.675<25%差 Poor<1.14<0.47
3 讨论(Discussion)
3.1 生物信息数据库
准确的原生生物监测仍然需要进一步完善本土物种DNA条形码数据库。由于我国还未建立本土原生生物物种注释数据库,本次原生生物的注释采用的是国际通用的PR2数据库,仍然存在无法注释的OTU,注释结果并不能完全包含太湖流域沉积物原生生物信息,未来尚需开展大规模的本土物种DNA信息建库的研究工作。
3.2 eDNA技术的重复性评价
环境DNA宏条形码技术在监测与评价生物系统多样性方面体现出了极大的应用潜力[20],但是关于该技术对沉积物原生生物监测的重复性与稳定性的研究较少[21]。精准度会影响监测结果在时间尺度与空间尺度上的比较[22],重复性是一个重要的衡量依据,体现了一项监测技术的稳定性与可行度[23]。使用环境DNA技术分析的对象主要是OTU[24],因此基于分类单元的技术重复性分析结果举足轻重。本研究中发现虽然至少在2个平行样本中检出的OTU的平均交叉率只有47%,但是基于序列权重的交叉率达到84.25%,说明平行样本间共有的OTU覆盖了绝大多数的优势物种,某一个平行中特有的OTU主要为低丰度OTU,在环境中分布不均匀,并不影响技术重复性研究的最终结论,这一现象侧面反映了环境DNA技术具有极高的获取环境中物种信息的能力。
3.3 沉积物原生生物多样性的季节差异
基于OTU与基于属计算的原生生物alpha-多样性指数在丰富度上存在着显著性差异,然而其他指数并不存在类似差异,丰富度指数表示OTU的分类单元数,说明3月原生生物群落比8月具有更多的种,而8月原生生物群落在丰度、分布上与3月差别并不明显。beta-多样性在季节上差异不显著,这可能是由于沉积物中的环境DNA存在随着时间的累积过程,反映的是近段时间内生物组成,另外,相比水样中的DNA,沉积物DNA受外界温度、光照、水流等因素影响更小。
3.4 环境DNA技术监测沉积物原生生物的展望
利用水环境中的各种生物多样性参数的指标来评价生境整体的好坏,这些指标被称为水质生物评价参数[25]。国内使用该方法评价生态健康已有60多年,相比理化参数,生物指数可以更加直接地指示生态健康。水体中的原生生物主要生活在沉积物中,迁移活动依靠水流等被动作用,因此生物活动位置较其他水体生物稳定[26]。沉积物是污染物长期汇集的场所,易受污染的影响而改变群落结构与生物多样性[27],所以沉积物原生生物群落的各种生物多样性指数可以反映长期以来水环境生态健康状态[28]。
太湖流域一直以来是环境监测评价研究的热点地区,大多数研究基于浮游动植物[29]、水体大型生物如鱼类[30]开展研究,鲜有研究关注沉积物中的原生生物群落[31]。在环境DNA技术的支撑下,构建基于原生生物完整性指数用于评价水生态健康[32],使用原生生物完整性指数来评价水生态健康具有一定意义上的方法学补充与数据参考价值[33],但参考点与受损点的选取不可避免存在着研究人员的主观因素,不能完全代表监测区域的水生态健康状态。常规监测中,建议使用沉积物原生生物群落来构建生物完整性指数用于评价长期的生态健康状态。沉积物中污染物浓度与组成更多反映了水环境长期的变化趋势,起伏较低、更为稳定,同时不受光照等影响,底层水温主要受季节变化的影响,温度变化波动较小。
环境DNA技术为弥补了传统监测方法的不足[34],不依赖于专业物种鉴定专家,更加快速高效且可标准化,不需要对沉积物原生生物进行培养,降低了时间成本[35],避免了因培养条件限制而遗漏重要物种的可能性。未来通过进一步的技术研发,如数据库完善、方法的标准化,可实现在实际的水生态环境监测和评估上的广泛应用。
[1] Geisen S. Thorough high-throughput sequencing analyses unravels huge diversities of soil parasitic protists [J]. Environmental Microbiology, 2016, 18(6): 1669-1672
[2] Titeux N, Henle K, Mihoub J B, et al. Biodiversity scenarios neglect future land-use changes [J]. Global Change Biology, 2016, 22(7): 2505-2515
[3] Plutzer J, Karanis P. Neglected waterborne parasitic protozoa and their detection in water [J]. Water Research, 2016, 101: 318-332
[4] 李静, 卢文轩, 张雷燕, 等. 夏季蓝藻水华期间太湖河口区和敞水区纤毛虫群落组成及水平分布[J]. 水生生物学报, 2014, 38(5): 860-867
Li J, Lu W X, Zhang L Y, et al. Community compositions and horizontal distribution of ciliates in Lake Taihu during the cyanobacterial bloom in summer [J]. Acta Hydrobiologica Sinica, 2014, 38(5): 860-867 (in Chinese)
[5] 沈韫芬, 顾曼如, 龚循矩, 等. 微型生物监测新技术[J]. 北京: 中国建筑工业出版社, 1994: 36-38
[6] 马正学, 宋玉珍, 刘红岩. 用原生动物评价黄河兰州段的水质[J]. 中国环境科学, 1994, 14(6): 401-406
Ma Z X, Song Y Z, Liu H Y. Assessment of pollution in Lanzhou reach of Huanghe River by using protozoan communities [J]. China Environmental Science, 1994, 14(6): 401-406 (in Chinese)
[7] Robertson L J, Clark C G, Debenham J J, et al. Are molecular tools clarifying or confusing our understanding of the public health threat from zoonotic enteric protozoa in wildlife? [J]. International Journal for Parasitology: Parasites and Wildlife, 2019, 9: 323-341
[8] Rakshit D, Sahu G R, Mohanty A K, et al. Bioindicator role of tintinnid (Protozoa: Ciliophora) for water quality monitoring in Kalpakkam, Tamil Nadu, south east coast of India [J]. Marine Pollution Bulletin, 2017, 114(1): 134-143
[9] Yang J H, Zhang X W. eDNA metabarcoding in zooplankton improves the ecological status assessment of aquatic ecosystems [J]. Environment International, 2020, 134: 105230
[10] Xie Y W, Wang J Z, Yang J H, et al. Environmental DNA metabarcoding reveals primary chemical contaminants in freshwater sediments from different land-use types [J]. Chemosphere, 2017, 172: 201-209
[11] Zhang L J, Yang J H, Li H Z, et al. Development of the transcriptome for a sediment ecotoxicological model species, Chironomus dilutus [J]. Chemosphere, 2020, 244: 125541
[12] Li J Y, Su L, Wei F H, et al. Bioavailability-based assessment of aryl hydrocarbon receptor-mediated activity in Lake Tai Basin from Eastern China [J]. The Science of the Total Environment, 2016, 544: 987-994
[13] Xie Y W, Wang J Z, Wu Y, et al. Using in situ bacterial communities to monitor contaminants in river sediments [J]. Environmental Pollution, 2016, 212: 348-357
[14] Wu W X, Liu H B. Disentangling protist communities identified from DNA and RNA surveys in the Pearl River-South China Sea Continuum during the wet and dry seasons [J]. Molecular Ecology, 2018, 27(22): 4627-4640
[15] Sweeney B W, Battle J M, Jackson J K, et al. Can DNA barcodes of stream macroinvertebrates improve descriptions of community structure and water quality? [J]. Journal of the North American Benthological Society, 2011, 30(1): 195-216
[16] Martins F M S, Galhardo M, Filipe A F, et al. Have the cake and eat it: Optimizing nondestructive DNA metabarcoding of macroinvertebrate samples for freshwater biomonitoring [J]. Molecular Ecology Resources, 2019, 19(4): 863-876
[17] 李飞龙, 杨江华, 杨雅楠, 等. 环境DNA宏条形码监测水生态系统变化与健康状态[J]. 中国环境监测, 2018, 34(6): 37-46
Li F L, Yang J H, Yang Y N, et al. Using environmental DNA metabarcoding to monitor the changes and health status of aquatic ecosystems [J]. Environmental Monitoring in China, 2018, 34(6): 37-46 (in Chinese)
[18] 高旭, 杨江华, 张效伟. 浮游动物DNA宏条形码标志基因比较研究[J]. 生态毒理学报, 2020, 15(2): 61-70
Gao X, Yang J H, Zhang X W. Study on the selection of marker genes in zooplankton DNA metabarcoding monitoring [J]. Asian Journal of Ecotoxicology, 2020, 15(2): 61-70 (in Chinese)
[19] 张丽娟, 徐杉, 赵峥, 等. 环境DNA宏条形码监测湖泊真核浮游植物的精准性[J]. 环境科学, 2021, 42(2): 796-807
Zhang L J, Xu S, Zhao Z, et al. Precision of eDNA metabarcoding technology for biodiversity monitoring of eukaryotic phytoplankton in lakes [J]. Environmental Science, 2021, 42(2): 796-807 (in Chinese)
[20] 孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究[J]. 生态毒理学报, 2018, 13(5): 76-86
Sun J Y, Yang J H, Zhang X W. Identification and biomass monitoring of zooplankton Cladocera species with eDNA metabarcoding technology [J]. Asian Journal of Ecotoxicology, 2018, 13(5): 76-86 (in Chinese)
[21] van der Loos L M, Nijland R. Biases in bulk: DNA metabarcoding of marine communities and the methodology involved [J]. Molecular Ecology, 2021, 30(13): 3270-3288
[22] Sleith R S, Katz L A. Ubiquity or not ubiquity: That is the question [J]. Molecular Ecology, 2019, 28(22): 4842-4844
[23] Keeley N, Wood S A, Pochon X. Development and preliminary validation of a multi-trophic metabarcoding biotic index for monitoring benthic organic enrichment [J]. Ecological Indicators, 2018, 85: 1044-1057
[24] Jiang J G, Shen Y F. Use of the aquatic protozoa to formulate a community biotic index for an urban water system [J]. Science of the Total Environment, 2005, 346(1-3): 99-111
[25] 蔡琨, 张杰, 徐兆安, 等. 应用底栖动物完整性指数评价太湖生态健康[J]. 湖泊科学, 2014, 26(1): 74-82
Cai K, Zhang J, Xu Z A, et al. Application of a benthic index of biotic integrity for the ecosystem health assessment of Lake Taihu [J]. Journal of Lake Sciences, 2014, 26(1): 74-82 (in Chinese)
[26] Delavenne J, Keszler L, Castelin M, et al. Deep-sea benthic communities in the largest oceanic desert are structured by the presence of polymetallic crust [J]. Scientific Reports, 2019, 9(1): 6977
[27] Peralta-Maraver I, Galloway J, Posselt M, et al. Environmental filtering and community delineation in the streambed ecotone [J]. Scientific Reports, 2018, 8(1): 15871
[28] 苏玉, 曹晓峰, 黄艺. 应用底栖动物完整性指数评价滇池流域入湖河流生态系统健康[J]. 湖泊科学, 2013, 25(1): 91-98
Su Y, Cao X F, Huang Y. Stream ecosystem health assessment of inflow rivers in Lake Dianchi Catchment by using Benthic Integrity Biotic Index (B-IBI) [J]. Journal of Lake Sciences, 2013, 25(1): 91-98 (in Chinese)
[29] Dawson J, Pillay D, Roberts P J, et al. Declines in benthic macroinvertebrate community metrics and microphytobenthic biomass in an estuarine lake following enrichment by hippo dung [J]. Scientific Reports, 2016, 6: 37359
[30] Trebitz A S, Hoffman J C, Grant G W, et al. Potential for DNA-based identification of Great Lakes fauna: Match and mismatch between taxa inventories and DNA barcode libraries [J]. Scientific Reports, 2015, 5: 12162
[31] 刘霞, 陆晓华, 陈宇炜. 太湖浮游硅藻时空演化与环境因子的关系[J]. 环境科学学报, 2012, 32(4): 821-827
Liu X, Lu X H, Chen Y W. Long-term evolution of planktonic diatoms and their relationships with environmental factors in Lake Taihu [J]. Acta Scientiae Circumstantiae, 2012, 32(4): 821-827 (in Chinese)
[32] 张翔, 张效伟, 杨江华, 等. 应用物种DNA条形码识别太湖流域部分底栖无脊椎动物种类[J]. 环境监控与预警, 2016, 8(6): 18-21, 25
Zhang X, Zhang X W, Yang J H, et al. Applying DNA barcoding for identification of some invertebrate macrozoobenthos at Taihu Lake Basin [J]. Environmental Monitoring and Forewarning, 2016, 8(6): 18-21, 25 (in Chinese)
[33] Stapanian M A, Micacchion M, Adams J V. Wetland habitat disturbance best predicts metrics of an amphibian index of biotic integrity [J]. Ecological Indicators, 2015, 56: 237-242
[34] Abdullah Al M, Xue Y Y, Xiao P, et al. DNA metabarcoding reveals the significant influence of anthropogenic effects on microeukaryotic communities in urban waterbodies [J]. Environmental Pollution, 2021, 285: 117336
[35] Chen W D, Ren K X, Isabwe A, et al. Stochastic processes shape microeukaryotic community assembly in a subtropical river across wet and dry seasons [J]. Microbiome, 2019, 7(1): 138