河流可以为人类提供食物、基本生活用水,以及提供旅游、灌溉、输送和水产养殖等,因此河流对人类的发展极其重要[1]。但是由于人类活动,例如污染、过度捕捞和水利开发等[2-3],改变了原有的河流生境、影响了河流的水质状况和降低河流水生生物多样性数量甚至导致生物灭绝。获得有效的河流水生生物多样性信息可以有效进行水生生物保护的研究[4]。河流水生生物特殊的生活环境及其滞后的调查技术使得水生生物的基础资料较陆地生物少,因此要获得河流水生生物多样性信息需要更有效的方法来进行监测。
环境DNA(environmental DNA, eDNA)技术是近年来受到广泛关注的一种生物检测技术,其中eDNA是指生物与环境相互作用遗留的DNA,可能来源于生物脱落的组织、分泌物、排泄物、血液和尸体等[5-6],分布在土壤、沉积物、空气、冰芯和自然水体等环境中,是生物完整和片段化DNA的混合物。eDNA技术是通过对这些DNA的鉴别来对环境中生物的种类、群落结构及生活习性等进行定性或定量的分析。作为一种新兴的生态系统评估技术,eDNA技术比传统的生态监测技术更加便利且有效。
河流eDNA发展至今已经进行了大量研究,诸多的文献记录可为科学家寻找关联性提供了可能。文本挖掘可以通过信息检索、信息提取和数据挖掘3个步骤对大量文献进行量化分析[7],为研究者提供研究数据。知识图谱通过分析关键词、国家和机构等关键信息的相似度,构建和绘制各单元之间的联系,以寻求某研究领域的研究热点、主题演变拐点,为后续研究提供知识储备及创新方向[8-9]。该研究方法已经广泛应用于金融、生物医疗和管理等诸多领域[10],但是河流生态中的应用较少。
到目前为止,eDNA技术的不断发展并趋于成熟,逐渐成为水生生物调查的重要手段,减少了传统方法在分类学上的困难及调查的成本。河流eDNA发展至今已经有了一定的文献基础,但是国内关于河流eDNA研究的知识图谱却较少见。本文通过对近30年河流eDNA相关文献的关键词、发表年份、研究领域和国际合作等文本数据进行挖掘分析。绘制研究热点、学科方向、国际合作三大类科学图谱(图1),揭示河流eDNA的热点方向、前沿研究趋势和国际合作方向,为未来eDNA技术在河流环境中应用及研究提供参考。
1 数据与方法(Data and methods)
1.1 数据收集
本文所采集的数据来源于Web of Science(WOS)核心合集数据库,该数据库具有世界上最有影响力和最重要的文献,包括Science Citation Index-Expand (SCI-E, 1945—至今)、Social Science Citation Index (SSCI, 1956—至今)、Arts & Humanities Citation Index (A&HCI, 艺术与人文引文索引)和Emerging Sources Citation Index (ESCI, 2015—至今)的不同版本的文献。eDNA的首次提出于1987年,但该技术的研究真正得到发展是1990年之后[11],因此本次检索时间跨度设为1990—2020年。为确保检索结果的全面性,本次检索以主题“TS=(“environmental DNA” OR “eDNA” AND “river*”)”,共检索文献2 333篇。
1.2 数据处理
根据1990—2020年已发表2 333篇相关文章,从中选取2 151篇文献(研究性文章1 947篇,综述文章204篇)进行分析,并提取文献相关信息:关键词、作者、研究机构、来源出版物、文章类型和地理区域等。英格兰、苏格兰、北爱尔兰和威尔士发表的文章被归类为英国(United Kingdom)发表的文章。使用Web of Science自带的统计分析功能,对收集的文献发文量、研究方向和学科类别等进行统计分析。本文根据检索文献,从中共提取关键词9 024个,研究国家/地区110个,学科类别103个,研究方向71个。

图1 河流eDNA科学图谱流程
Fig. 1 The flowchart for scientific mapping of river eDNA
1.3 可视化分析
VOSviewer是荷兰莱顿大学科学技术研究中心(The Centre for Science and Technology Studies, CWTS)开发的科学计量网络分析软件,是一种文本挖掘工具,可用于关键词分析、聚类分析和数据处理等[12]。该软件以“共现聚类”为核心,利用VOS映射技术来计算2个标签的距离。本文采用VOSviewer软件对河流eDNA进行可视化分析,分析内容主要分为关键词聚类分析及关键词时间趋势分析,并深入挖掘可视化图形的内在含义。在共现网络视图中,关键词及国家由节点表示,节点之间的线代表两者的共现关系,节点越大说明出现的次数越多,连线密集说明2个节点之间联系越紧密。
2 结果与分析(Results and analysis)
2.1 研究热点分析
某一学科不同年份的发文量可在一定程度上反映该学科的发展速度以及发展水平。环境DNA技术在近年来得到越来越广泛的发展,在文献发表方面,其数量也在不断增加(图2)。1990—2000年间,关于eDNA的文献发表量仅占检索总数的0.86%。在该时间段,发文量较少且呈平缓的趋势,eDNA正处于萌芽发展阶段,年度发文量少于10篇。2001—2011年间发文量逐年缓慢上升,虽然上升趋势缓慢,在这期间却发表了众多对后续研究具有强大影响力的文章。2004年,Handelsman[13]发表了一篇关于宏基因组的综述,近5年其影响因子达到18.504,是本次检索文章结果中该阶段被引量最多的文献,年平均引用次数达到67.39。2003年,Baker等[14]发表了一篇关于通用引物16S rRNA的综述,该文的影响力也不容小觑。特别的是,2008年,Ficetola等[6]发表文章,将eDNA技术首次应用于水生生态系统中,检测两栖动物美国牛蛙入侵状况。2012—2020年间是eDNA飞速发展的时期,年发文量呈指数式增长。2012年,Thomsen等[15]首次利用eDNA技术对淡水中的多物种进行检测,开启了eDNA技术在淡水生物多样性检测的应用研究。
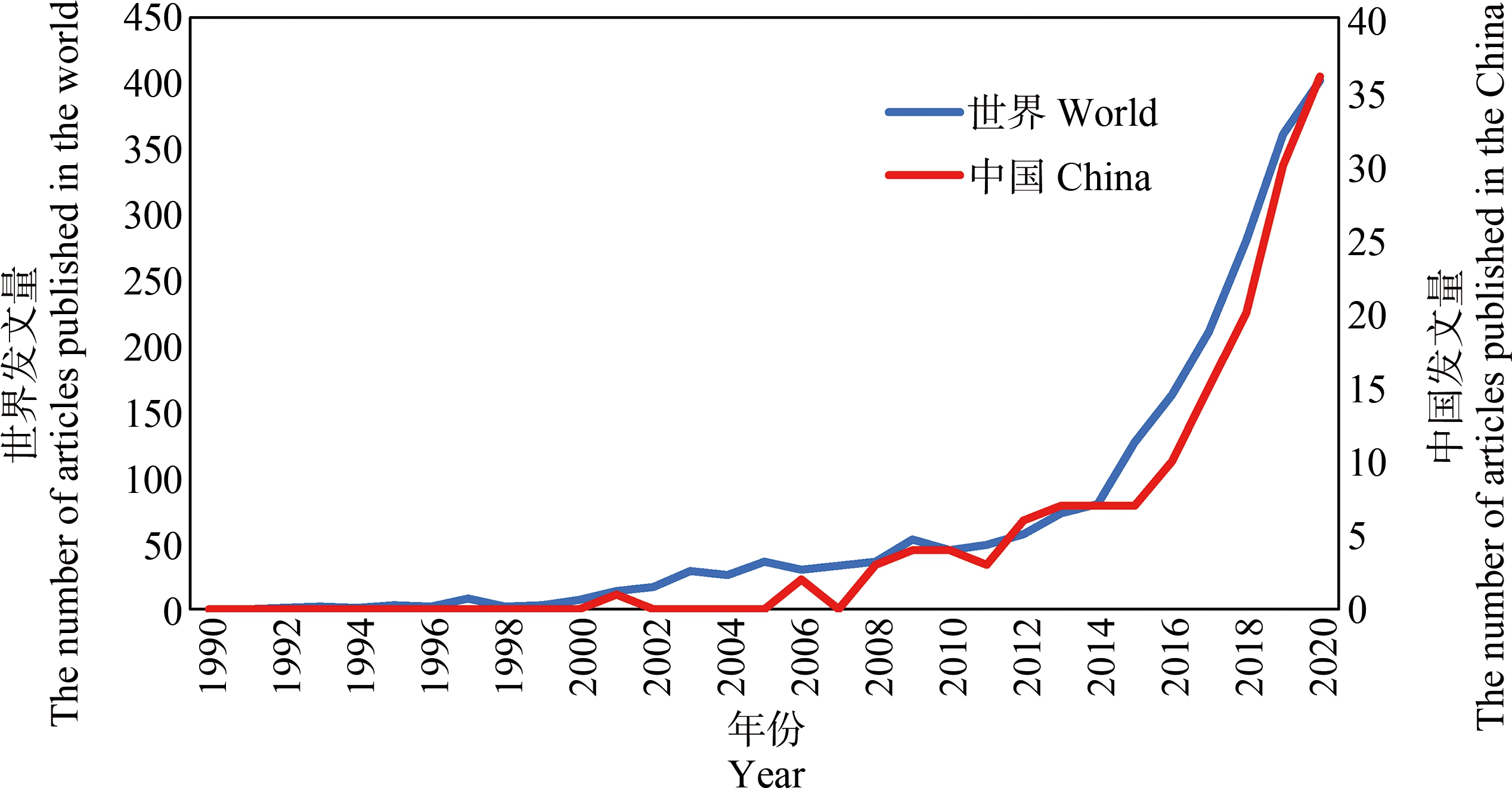
图2 1990—2020年河流eDNA发文数量
Fig. 2 Number of river eDNA publications from 1990 to 2020
高频关键词分析可以反映并分析一段时间内某研究领域的研究热点。根据VOSviewer软件所作的关键词网络分析图(图3),就颜色将50个关键词分为3个主题进行研究:宏基因组鉴别(绿色聚类)、宏条形码技术与群落生态监测(蓝色聚类)、生态保护及eDNA应用(红色聚类)。3个主题之间相互连接,层层递进。以下,对3个主题进行进一步分析。
宏基因组鉴别。绿色聚类的关键词数量少且出现频率不高,仅有19个关键词,较突显的关键词有:“diversity”“identification”“DNA”“metagenomics”。eDNA技术的发展离不开DNA测序技术。首先是DNA第一代测序技术,即1997年Maxam和Gilbert发明的化学降解法及Sanger的双末端终止法[16-17]。该方法只能对样本进行单独测序,因此不足以处理复杂的环境样本。之后的几十年里,在第一代测序技术的发展基础上,产生了条形码技术[18]、第二代测序技术(next-generation sequencing technology, NGS,即高通量测序技术)[19]、第三代测序技术[20-21]。1988年,Handelsman等[22]提出的宏基因组学概念为研究微生物提供了新的思路和方法。该技术是基因克隆文库的进一步深化,并不依赖于微生物的分离培养,可直接提取环境中的微生物基因的遗传物质,为进一步认识和解决环境问题提供了新方式。

图3 河流eDNA关键词共现网络
Fig. 3 The keyword co-occurrence network of river eDNA
宏条形码技术与群落生态监测。蓝色聚类的关键词含有11个,主要的关键词有:“environmental DNA”“metabarcoding”“communities”“ancient DNA”等。宏条形码技术(metabarcoding)是一种结合DNA分类与高通量测序2种技术的快速生物多样性评估方法[23]。该技术可以同时快速且高性价比地识别数千个样本的整个分类组成,提供比传统方法更全面的群落分析[24]。eDNA技术通过对生物群落监测可为河流环境中物种和群落的管理和保护提供必要的信息,例如:物种食性分析[25]、外来物种监测和生物多样性分析。该技术也可应用于古环境样本,其对古DNA的分析为研究者们理解气候变化及生态环境响应提供了新视角[26]。
eDNA技术的应用。红色聚类共有20个关键词,以eDNA为中心,其他关键词围绕在其周围。围绕eDNA开展了河流生态系统多样化的应用研究,生物多样性及其保护的研究占多数。多样性是生物基本的特征,而全球淡水生物多样性正在以惊人的速度下降,这可能对生态系统的结构和功能造成破坏,进而对人类福祉造成有害影响,因此降低生物多样性丧失速度及其生物多样性有害变化是国际目标[16]。除生物多样性检测及其保护外,eDNA技术在河流入侵物种调查[27-28]、生物量调查[29-30]和珍稀物种保护[31]等方面发挥了重要作用。
从上述关键词中,筛选出关联度最高10个的关键词(表1)。关键词分析可以较全面地提供某一研究的研究热点及趋势[32]。根据该表,在河流环境DNA研究领域更加注重于生物多样性的研究。在生态系统运行的过程中,生物多样性的损失和增加都会影响生态系统稳定性以及生态系统功能和服务的可持续性[33]。生物的多样性保护学科文献发文量占比的升高,说明人们已经越来越重视生物多样性的价值[34],并不断向生物多样性保护及其可持续利用的目标前进。生物多样性的保护工作大多依赖于生物监测,以获得有关生态和时间尺度上的物种分布和种群规模的精确数据。eDNA技术为监测河流环境、获取河流生物信息提供了省时省力的方法[35]。
表1 河流eDNA频次最高的10个关键词
Table 1 The 10 keywords that appear most frequently in river eDNA
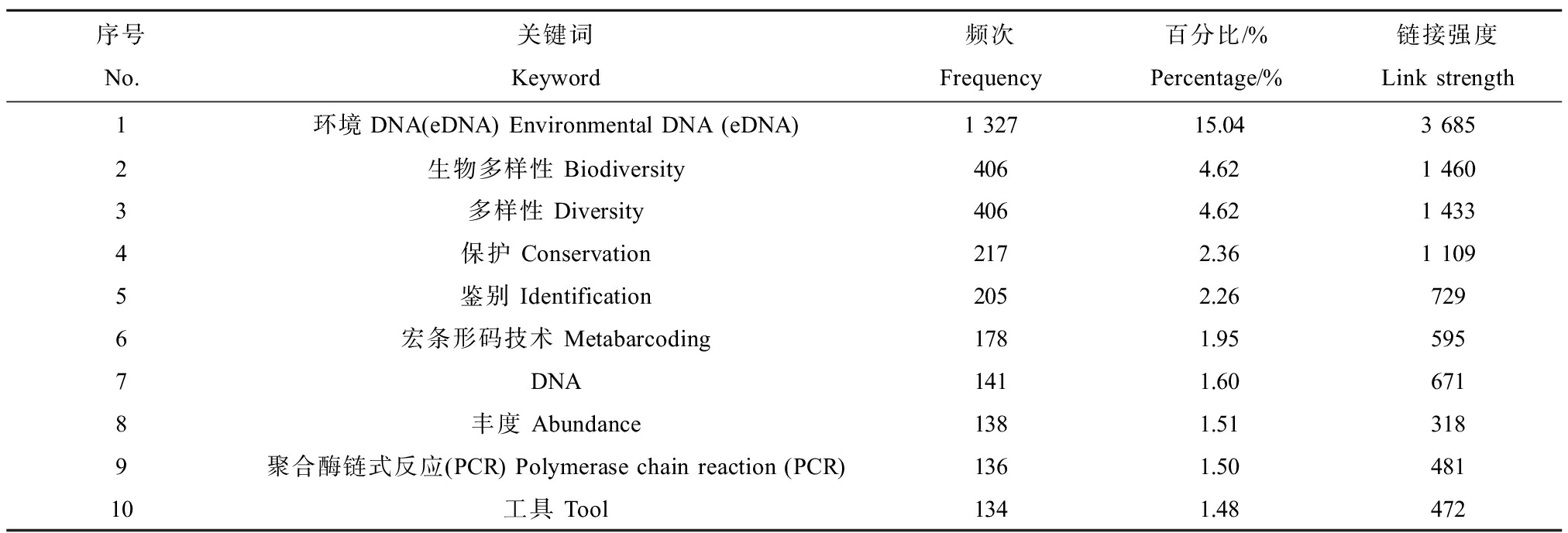
序号No.关键词Keyword频次Frequency百分比/%Percentage/%链接强度Link strength1环境DNA(eDNA) Environmental DNA (eDNA)1 32715.043 6852生物多样性 Biodiversity4064.621 4603多样性 Diversity4064.621 4334保护 Conservation2172.361 1095鉴别 Identification2052.267296宏条形码技术 Metabarcoding1781.955957DNA1411.606718丰度 Abundance1381.513189聚合酶链式反应(PCR) Polymerase chain reaction (PCR)1361.5048110工具 Tool1341.48472
根据WOS所得关键词绘制关键词共现网络,同时获得关键词出现的平均年份图(图4)。关键词出现的平均年份体现了eDNA技术在方法学及研究应用上的转变。在方法学上eDNA技术主要经历了3个阶段,在应用上不断创新的技术推动河流生物研究从定性到定量、微生物到大型水生生物、复杂到更简便发展。
河流eDNA的应用不断扩展离不开技术的发展。根据关键词随时间变化的趋势,eDNA的方法学研究经历了3个阶段。阶段1:基于一代测序的“扩增”和“克隆”。在该阶段的研究开创性地将DNA元条形码应用于浮游细菌基因多样性的分析,并使得基于克隆的宏基因组学和DNA元条形码在微生物学的研究中得到普遍应用[11, 22, 36]。阶段2:基于高通量测序的eDNA宏条形码技术。由于高通量测序技术的出现,使得从环境样本中恢复DNA序列数据成为可能,使得eDNA技术的应用不断扩展[37]。例如:推断微生物群落中的个体健康[38]、分析多样性推断生态系统健康[39]和研究生物食性[40]。阶段3:近期在主要对eDNA方法的标准化和定量化研究。在该阶段更加追求eDNA技术在应用中标准化,以消除检测错误,提高eDNA检测的准确性[41]。
随着技术的发展,河流eDNA的应用研究随着技术发展也有不同侧重点(图4)。近年来eDNA技术更加注重河流环境的生态监测、鱼类监测[42]、河流管理和生物多样性监测等方面。河流生态保护是一个长期的过程,制定可持续性的管理模式对于河流生态环境保护具有重大意义。流域生态系统健康评价是促进我国生态文明建设的重要手段,评价结果是流域规划和管理的重要依据[43]。河流生态系统作为流域生态系统的一部分,进行简单、有效的河流监测,收集有效的河流生态信息,有利于决策者在应对环境变化时采取干预与管理活动[44]。

图4 河流eDNA研究高频关键词的平均出现年份
Fig. 4 Average year of appearance of high-frequency keywords in river eDNA studies
2.2 学科方向分析
按Web of Science类别对检索文献进行归类,统计各学科方向发表论文数量,并选取1990—2020年与2015—2020年排名前十的学科类别(图5(a))分析其变化趋势。在河流eDNA研究中,比重较大的学科方向主要有生态学(26.00%)、微生物学(14.96%)、多学科科学(14.53%)和生物化学分子生物学(12.98%)等。对比2015—2020年学科占比,其中生态学、环境科学和生物多样性保护等学科类别的占比呈明显上升趋势,而微生物学、生物化学分子生物学和生物技术微生物学的占比却呈下降趋势。由此可见,在河流eDNA研究领域中,生态学、环境科学和生物多样性保护是目前较热门的学科,其发文数量在近几年来不断提升。为进一步探索河流eDNA在不同学科中的应用,选取生态学、微生物学两大学科进行深入分析(图5(b)和图5(c))。
在总体检索结果中,生态学学科的发文量位于各大学科发文量的首位,并且近年来有不断升高的趋势。以eDNA(environmental DNA)为中心的生态学学科关键词共现网络主要分为两大类,红色聚类是方法学,更注重eDNA技术在应用过程中的技术壁垒;绿色聚类注重于eDNA技术在河流生态中的应用(图5(b))。河流作为人类经济发展不可或缺的一部分,其生态问题成为21世纪全世界面临的重大挑战,也成为了政府及学术界关注的焦点[45]。生态保护工作依赖于生物多样性的评估及环境污染物生态毒理学评估。研究表明,利用eDNA技术可以通过监测水生生物群落、生态系统功能进而识别河流中化学污染压力源,有效评估化学污染的影响[46-47]。因此,在河流生态学学科中关注的主要焦点在于通过监测河流中的物种变化、生物多样性改变和生物量增减等状况进行河流生态环境的监测,为管理部门制定保护政策提供依据。

图5 排名前十的研究方向占比和学科关键词共现网络
注:(a)1990—2020年与2015—2020年排名前十的研究方向占比;(b)生态学学科关键词共现网络;(c)微生物学学科关键词共现网络。
Fig. 5 Percentage of top 10 research direction and the keyword co-occurrence network of disciplines
Note: (a) Proportion of top 10 research directions in 1990—2020 and 2015—2020; (b) The keyword co-occurrence network of ecology; (c) The keyword co-occurrence network of microbiology.
微生物学学科的发文在总的检索结果中仅次于生态学,但是近年来微生物学科文献发文量的比例呈现下降的趋势。eDNA最早的研究便是源于微生物[11, 36],在2003年之前eDNA技术的应用都一直局限在微生物的鉴定之上。eDNA宏条形码技术为探究微生物生物多样性提供了更大的可能,同时也可为渔业养殖、河流生态保护提供有利的信息。已经有多项研究验证了该技术在淡水养殖中病原检测方面的有效性[48],研究数据表明eDNA宏条形码技术有望成为监测水产养殖生物威胁的有用工具[49]。宏条形码技术可保证已分离微生物种类多样性及信息测定的准确性,可对不同环境下的微生物进行高效率、高层次的生物测序。但是也存在进一步完善之处,例如利用扩增盒无法实现新菌株的培养[50]。
eDNA技术研究领域重心从微生物的研究向更广泛的生态监测、河流管理方面转变(图5(c))。在eDNA技术发展早期,微生物学家利用eDNA技术研究难以存活的微生物样本,但是在之后的20年中随着该技术的不断完善与发展已经逐步从微生物的应用转向高等水生生物的鉴定。随着人们对河流生态系统不断深入的研究,河流中所存在的环境问题不断浮现,仅仅对微生物的研究及传统的生态监测方法已经无法满足河流生态保护的需求,特别是在河流物种保护方面eDNA技术的价值已经逐渐凸显[51-52]。研究表明,对于稀有物种及难以捕捉的物种eDNA技术在其监测方面较传统方法更具灵敏性[27, 29],对于保护珍稀物种、发现早期入侵物种具有重要意义。同时,eDNA可以划定水生系统的物种范围,进一步分析环境因素对生物分布的影响[53-54]。另一方面,虽然eDNA技术在生物定量方面目前还存在一定的困难,但是却能够实现物种的半定量检测(物种的相对占比)[55]。因此,管理者可以通过获取有效的信息对河流采取保护措施,科学家也应该更进一步完善eDNA技术。
2.3 国际合作
河流eDNA的研究无论是数量还是研究方向上都具有明显的地理差异(表2,图6(a))。根据文章的通讯作者所在国家分布表明,共有111个国家及地区进行了河流eDNA的研究,其中美国在河流eDNA方面的研究数量上在世界上占主导地位,英国、法国和德国等发表的文献数量紧随其后。20世纪80年代,欧洲和北美开始了河流保护行动,因此这些国家对于该领域的研究远远早于并多于其他国家,参考文献主要来源国是美国、德国和英国(表2)。各个国家根据国内情况发展具有本国特点的研究方向,例如:欧洲国家(英国、法国和德国)更注重微生物及进化生物学的研究、亚洲国家(日本、中国)在微生物应用及微生物学、海洋淡水及淡水生态学上有较多的研究。
排名前三十的国际科学合作共现网络图的连线关系揭示了国家之间不同程度的合作关系(图6(a))。在国际合作关系图中,以美国、英国、法国、德国、澳大利亚和加拿大为中心,是河流eDNA研究领域最重要的合作关系体。在河流eDNA研究领域合作较密切的国家主要有美国与英国、美国与加拿大、美国与澳大利亚、美国与德国、英国与法国等。美国作为出版文献最多的国家,其在多国合作的研究同样也居于领先地位,与世界上76个国家有关于河流eDNA的国际合作,但是其单国发文量高于与其他国家合作数量。英国在河流eDNA领域的发表文献数量远远少于美国,但是英国的国际合作国家数量却包含74个,其中与之合作关系较密切的国家有美国、法国、丹麦、加拿大和德国等。在发表文献数量前十的国家中,瑞士、法国和西班牙是进行国际合作最积极的国家,其与他国合作文章数量占国家总发文量均高于75%。为深入了解国家间研究差异,选取发达国家代表美国,以及发展中国家代表中国进行深入分析(图6(b)和图6(c))。
在eDNA技术发展的几十年间,美国在技术发展与应用上都处于研究前沿(图6(b))。美国科学家在eDNA技术的发展上做出了重要的贡献,在eDNA技术发展的几十年间,美国在该技术的应用方向上也发生了改变。作为最早期开展河流研究的国家之一的美国,也是最具有创造力的国家,美国在河流eDNA的研究中含有53个研究方向,主要涉及环境科学生态学、科学技术及其他主题、生物化学分子生物学等研究。根据关键词平均发表年份图(图6(b)),在该技术发展的初期美国在河流环境中的研究热点集中于河流eDNA生物技术的发展及其在微生物中的研究。但是,近年来美国关于河流eDNA的研究更关注于河流生物监测[56]、生物多样性保护等方面。
中国在河流eDNA技术上的应用时间上远远落后于国际,近几年在研究方向占比上也发生了变化(图6(c))。虽然国际上早期就有了河流eDNA应用的例子,但是根据本次检索结果,中国2001在WOS中才有关于河流eDNA的发表记录(图2)。并且在之后的十几年中,中国的发文量在世界发文量中占比依旧是少数。虽然中国的发文量在排名前十的位置,但是中国的篇均被引频次数却不高(表2)。中国早期主要是在发展河流eDNA生物技术及其在微生物中的应用,直到近几年,才将研究重心转向环境科学、生态学及河流中生物多样性的保护。特别是中国在2015年为解决中国面临的水生态环境问题颁布了《水污染防治计划》,国内水环境研究也从支撑水环境管理的应用研究转向促进流域生态环境改善为前提的实验研究[10]。
表2 河流eDNA发文量前十的国家
Table 2 Top 10 countries with published literature on river eDNA
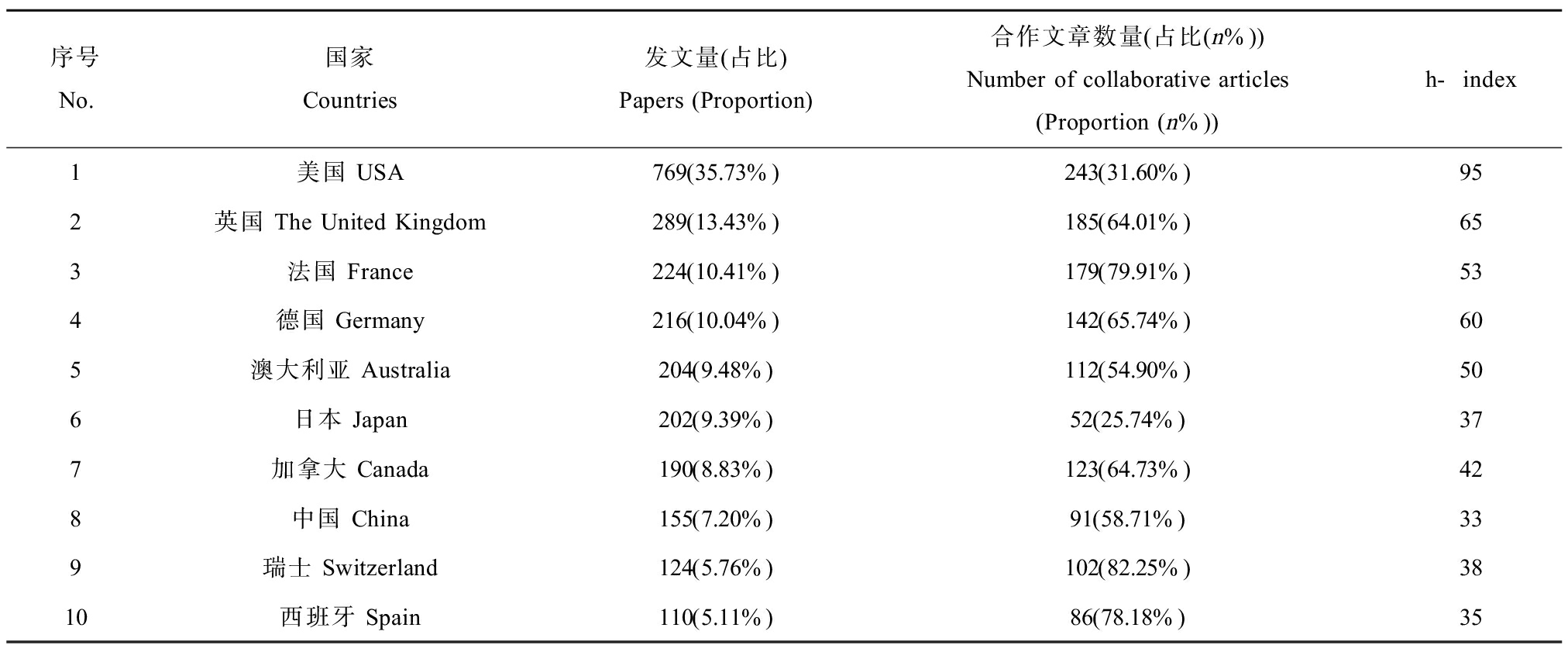
序号No.国家Countries发文量(占比)Papers (Proportion)合作文章数量(占比(n%))Number of collaborative articles (Proportion (n%))h-index1美国 USA769(35.73%)243(31.60%)952英国 The United Kingdom289(13.43%)185(64.01%)653法国 France224(10.41%)179(79.91%)534德国 Germany216(10.04%)142(65.74%)605澳大利亚 Australia204(9.48%)112(54.90%)506日本 Japan202(9.39%)52(25.74%)377加拿大 Canada190(8.83%)123(64.73%)428中国 China155(7.20%)91(58.71%)339瑞士 Switzerland124(5.76%)102(82.25%)3810西班牙 Spain110(5.11%)86(78.18%)35
注:n%,国家合作文章数量占该国文章总数量的比例。
Note: n%, the ratio of the number of national cooperative articles to the total number of articles in the country.

图6 国家合作共现网络
注:(a)河流eDNA排名前三十的国家及其国家间的合作;(b)美国河流eDNA文献关键词平均出现年份;(c)中国河流eDNA文献关键词平均出现年份。
Fig. 6 The co-occurrence network of country cooperation
Note: (a) Top 30 countries for river eDNA research and their collaboration; (b) The average year of occurrence of keywords in the river eDNA literature in USA; (c) The average year of occurrence of keywords in the river eDNA literature in China.
3 总结与展望(Summary and prospect)
eDNA技术在河流生态系统中多方面的应用使其在近几年的研究的方向、数量上都有了很大的提升,说明eDNA正在以迅猛之势发展。目前,该技术在生态环境科学、微生物研究等方面占较大的比例,但是近几年来生物多样性保护、渔业方面的研究占比有所提升。决策者们也可以通过该技术快速有效的收集河流信息,对河流生态环境进行监测。近年来,生态学、生物多样性保护和环境科学等学科的比例有所提升,而微生物学、生物技术微生物学的学科占比却呈下降趋势。对于早先进行河流保护行动的美国及其一些欧洲国家在河流eDNA的研究远远多于一些非洲及亚洲国家,研究方向也存在较大的差异。在国际合作方面,美国虽然是与其他国家的合作数量最多的,与加拿大的合作关系最密切,但是瑞士、法国和西班牙的合作文章数量占比本国文章总数量却居于前列。随着时间推移,各国在河流eDNA中的研究重心都发生了转移,美国逐渐从河流微生物的研究转向河流生物监测及生物多样性保护,中国的研究重心主要促进流域生态环境改善的方向转移。
尽管eDNA技术克服了传统技术的一些缺陷与不足,并在一定程度上对河流生态环境保护做出贡献,但是该技术在河流中的应用仍存在许多挑战与限制。首先,DNA存在降解及其样品被污染的问题。DNA在水中的降解速率会受到温度、光照和天气等自然要素的影响[57-58],限制eDNA的研究范围,影响监测精度[58]。DNA在样品采集到实验的过程中都有被污染的可能,从而产生假阳性及假阴性的结果[59-60]。其次是eDNA技术在生物定量上仍然存在挑战[61]。目前已经证实了DNA浓度与物种密度呈正相关,但是其精确度仍需要进一步探究[62]。全球公共数据库的缺少也是目前eDNA技术鉴定生物物种的困难之一,大多数生物体参考序列数据库的不完整性是使用eDNA进行生物多样性研究的一个重要限制[63]。
针对以上不足,未来eDNA技术还需在以下几个方面进一步扩展。在样品污染方面,为eDNA技术的采样和检测制定一套统一的标准,以消除方法错误。严格控制样品污染、实施标准化灭菌,避免由于操作产生的实验误差。对于DNA降解问题,结合非生物降解效应[64]与跨河网的eDNA建模[65]是有望阐明eDNA生态动力学一种途径。另一方面,完善的生物体序列数据库是进行可靠的eDNA检测的关键因素,数据库的扩充对eDNA研究具有重要意义[66]。此外,eDNA技术不能完全替代传统调查。DNA在水环境中存在许多不确定性导致其检出率低是限制eDNA技术在河流中应用的一大原因[15]。在生态保护方面,传统调查方法可以对eDNA收集的信息进行补充,同时eDNA也可以指引传统调查向正确的方向发展[67-68]。
eDNA技术为河流环境保护信息收集方面提供了强有力的技术手段,未来该技术在河流环境中应用上可进一步扩展,解决当前eDNA技术面临的风险与挑战。在全球河流环境日益严峻的情况下,eDNA技术在河流中的应用着重于生态环境的保护,发挥该技术在水质和水污染评价、河流生态系统健康评价等方面的作用。同时,为进一步发挥eDNA技术对河流生物的保护,应继续推动该技术的发展与成熟,加强国际合作推动河流eDNA的技术发展及信息交流。
[1] 唐涛, 蔡庆华, 刘建康. 河流生态系统健康及其评价[J]. 应用生态学报, 2002, 13(9): 1191-1194
Tang T, Cai Q H, Liu J K. River ecosystem health and its assessment [J]. Chinese Journal of Applied Ecology, 2002, 13(9): 1191-1194 (in Chinese)
[2] Dudgeon D, Arthington A H, Gessner M O, et al. Freshwater biodiversity: Importance, threats, status and conservation challenges [J]. Biological Reviews of the Cambridge Philosophical Society, 2006, 81(2): 163-182
[3] Freeman M C, Pringle C M, Jackson C R. Hydrologic connectivity and the contribution of stream headwaters to ecological integrity at regional Scales1 [J]. Journal of the American Water Resources Association, 2007, 43(1): 5-14
[4] Crookes S, Heer T, Casta eda R A, et al. Monitoring the silver carp invasion in Africa: A case study using environmental DNA (eDNA) in dangerous watersheds [J]. NeoBiota, 2020, 56(56): 31-47
eda R A, et al. Monitoring the silver carp invasion in Africa: A case study using environmental DNA (eDNA) in dangerous watersheds [J]. NeoBiota, 2020, 56(56): 31-47
[5] Barnes M A, Turner C R. The ecology of environmental DNA and implications for conservation genetics [J]. Conservation Genetics, 2016, 17(1): 1-17
[6] Ficetola G F, Miaud C, Pompanon F, et al. Species detection using environmental DNA from water samples [J]. Biology Letters, 2008, 4(4): 423-425
[7] Ananiadou S, Kell D B, Tsujii J I. Text mining and its potential applications in systems biology [J]. Trends in Biotechnology, 2006, 24(12): 571-579
[8] Chen C M. Searching for intellectual turning points: Progressive knowledge domain visualization [J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(Suppl 1): 5303-5310
[9] 陈俊梅, 张文翔, 刘甜甜, 等. 基于CiteSpace和知网数据库的《湖泊科学》创刊30年(1989—2018年)发表论文的文献计量学分析[J]. 湖泊科学, 2019, 31(4): 891-906
Chen J M, Zhang W X, Liu T T, et al. Scientometric analysis of papers published in 1989-2018 of Journal of Lake Sciences based on CiteSpace and CNKI database [J]. Journal of Lake Sciences, 2019, 31(4): 891-906 (in Chinese)
[10] 毛文山, 赵红莉, 蒋云钟, 等. 基于文献计量学的国内水生态环境研究知识图谱构建与应用[J]. 水利学报, 2019, 50(11): 1400-1416
Mao W S, Zhao H L, Jiang Y Z, et al. Construction and application of knowledge graph of domestic water eco-environment based on bibliometrics [J]. Journal of Hydraulic Engineering, 2019, 50(11): 1400-1416 (in Chinese)
[11] Giovannoni S J, Britschgi T B, Moyer C L, et al. Genetic diversity in Sargasso Sea bacterioplankton [J]. Nature, 1990, 345(6270): 60-63
[12] van Eck N J, Waltman L. Software survey: VOSviewer, a computer program for bibliometric mapping [J]. Scientometrics, 2010, 84(2): 523-538
[13] Handelsman J. Metagenomics: Application of genomics to uncultured microorganisms [J]. Microbiology and Molecular Biology Reviews, 2004, 68(4): 669-685
[14] Baker G C, Smith J J, Cowan D A. Review and re-analysis of domain-specific 16S primers [J]. Journal of Microbiological Methods, 2003, 55(3): 541-555
[15] Thomsen P F, Kielgast J, Iversen L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA [J]. Molecular Ecology, 2012, 21(11): 2565-2573
[16] Butchart S H, Walpole M, Collen B, et al. Global biodiversity: Indicators of recent declines [J]. Science, 2010, 328(5982): 1164-1168
[17] Maxam A M, Gilbert W. A new method for sequencing DNA [J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(2): 560-564
[18] Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceedings Biological Sciences, 2003, 270(1512): 313-321
[19] Pompanon F, Coissac É, Taberlet P. Metabarcoding, a new way of analysing biodiversity [J]. Biofutur, 2011, 319(319): 30-32
[20] Grad Y H, Lipsitch M, Feldgarden M, et al. Genomic epidemiology of the Escherichia coli O104: H4 outbreaks in Europe, 2011 [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(8): 3065-3070
[21] Goren A, Ozsolak F, Shoresh N, et al. Chromatin profiling by directly sequencing small quantities of immunoprecipitated DNA [J]. Nature Methods, 2010, 7(1): 47-49
[22] Handelsman J, Rondon M R, Brady S F, et al. Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products [J]. Chemistry & Biology, 1998, 5(10): R245-R249
[23] Ji Y Q, Ashton L, Pedley S M, et al. Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding [J]. Ecology Letters, 2013, 16(10): 1245-1257
[24] Dafforn K, Baird D, Chariton A, et al. Faster, higher and stronger? The pros and cons of molecular faunal data for assessing ecosystem condition [J]. Advances in Ecological Research, 2014, 51: 1-40
[25] Guillerault N, Bouletreau S, Iribar A, et al. Application of DNA metabarcoding on faeces to identify European catfish Silurus glanis diet [J]. Journal of Fish Biology, 2017, 90(5): 2214-2219
[26] 马睿, 陈建徽, 刘建宝, 等. 湖泊沉积物DNA在气候环境变化和生态系统响应研究中的应用[J]. 湖泊科学, 2021, 33(3): 653-666
Ma R, Chen J H, Liu J B, et al. Progress in the application of lake sediment DNA in climate and environmental change and ecosystem response [J]. Journal of Lake Sciences, 2021, 33(3): 653-666 (in Chinese)
[27] Dejean T, Valentini A, Miquel C, et al. Improved detection of an alien invasive species through environmental DNA barcoding: The example of the American bullfrog Lithobates catesbeianus [J]. Journal of Applied Ecology, 2012, 49(4): 953-959
[28] Takahara T, Minamoto T, Doi H. Using environmental DNA to estimate the distribution of an invasive fish species in ponds [J]. PLoS One, 2013, 8(2): e56584
[29] PilliodDavid S, GoldbergCaren S, ArkleRobert S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples [J]. Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70(8): 1123-1130
[30] Dougherty M M, Larson E R, Renshaw M A, et al. Environmental DNA (eDNA) detects the invasive rusty crayfish Orconectes rusticus at low abundances [J]. The Journal of Applied Ecology, 2016, 53(3): 722-732
[31] Vörös J, Márton O, Schmidt B R, et al. Surveying Europe’s only cave-dwelling chordate species (Proteus anguinus) using environmental DNA [J]. PLoS One, 2017, 12(1): e0170945
[32] Xu Y Y, Boeing W J. Mapping biofuel field: A bibliometric evaluation of research output [J]. Renewable and Sustainable Energy Reviews, 2013, 28: 82-91
[33] Carraro L, Mächler E, Wüthrich R, et al. Environmental DNA allows upscaling spatial patterns of biodiversity in freshwater ecosystems [J]. Nature Communications, 2020, 11(1): 3585
[34] Cardinale B J, Duffy J E, Gonzalez A, et al. Biodiversity loss and its impact on humanity [J]. Nature, 2012, 486(7401): 59-67
[35] Aylagas E, Borja  , Muxika I, et al. Adapting metabarcoding-based benthic biomonitoring into routine marine ecological status assessment networks [J]. Ecological Indicators, 2018, 95: 194-202
, Muxika I, et al. Adapting metabarcoding-based benthic biomonitoring into routine marine ecological status assessment networks [J]. Ecological Indicators, 2018, 95: 194-202
[36] Ogram A, Sayler G S, Barkay T. The extraction and purification of microbial DNA from sediments [J]. Journal of Microbiological Methods, 1987, 7(2-3): 57-66
[37] Sogin M L, Morrison H G, Huber J A, et al. Microbial diversity in the deep sea and the underexplored “rare biosphere” [J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(32): 12115-12120
[38] Andersson A F, Lindberg M, Jakobsson H, et al. Comparative analysis of human gut microbiota by barcoded pyrosequencing [J]. PLoS One, 2008, 3(7): e2836
[39] Hajibabaei M, Shokralla S, Zhou X, et al. Environmental barcoding: A next-generation sequencing approach for biomonitoring applications using river benthos [J]. PLoS One, 2011, 6(4): e17497
[40] Deagle B E, Roger K, Jarman S N. Analysis of Australian fur seal diet by pyrosequencing prey DNA in faeces [J]. Molecular Ecology, 2009, 18(9): 2022-2038
[41] Shu L, Ludwig A, Peng Z G. Standards for methods utilizing environmental DNA for detection of fish species [J]. Genes, 2020, 11(3): 296
[42] Tillotson M D, Kelly R P, Duda J J, et al. Concentrations of environmental DNA (eDNA) reflect spawning salmon abundance at fine spatial and temporal scales [J]. Biological Conservation, 2018, 220: 1-11
[43] Pan W B, Huang H, Yao P C, et al. Assessment methods of small watershed ecosystem health [J]. Polish Journal of Environmental Studies, 2020, 30(2): 1749-1769
[44] Peterson A, Soberón J. Essential biodiversity variables are not global [J]. Biodiversity and Conservation, 2018, 27: 1277-1288
[45] 王浩. 水生态文明建设的理论基础及若干关键问题[J]. 中国水利, 2016(19): 5-7
Wang H. Theoretical basis and key problems of water ecological civilization construction [J]. China Water Resources, 2016(19): 5-7 (in Chinese)
[46] Wang P Y, Yan Z G, Yang S W, et al. Environmental DNA: An emerging tool in ecological assessment [J]. Bulletin of Environmental Contamination and Toxicology, 2019, 103(5): 651-656
[47] Yang Y Z, Gao Y C, Huang X N, et al. Adaptive shifts of bacterioplankton communities in response to nitrogen enrichment in a highly polluted river [J]. Environmental Pollution, 2019, 245: 290-299
[48] Bastos Gomes G, Hutson K S, Domingos J A, et al. Use of environmental DNA (eDNA) and water quality data to predict protozoan parasites outbreaks in fish farms [J]. Aquaculture, 2017, 479: 467-473
[49] Peters L, Spatharis S, Dario M A, et al. Environmental DNA: A new low-cost monitoring tool for pathogens in salmonid aquaculture [J]. Frontiers in Microbiology, 2018, 9: 3009
[50] 刘莹, 韩锰, 王文磊, 等. 高通量技术在微生物培养中的应用进展及分子测序对比分析[J]. 安徽农业科学, 2020, 48(15): 16-19
Liu Y, Han M, Wang W L, et al. Application progress of high-throughput technology in microbial culture and comparative analysis of molecular sequencing [J]. Journal of Anhui Agricultural Sciences, 2020, 48(15): 16-19 (in Chinese)
[51] Rojahn J, Pearce L, Gleeson D M, et al. The value of quantitative environmental DNA analyses for the management of invasive and endangered native fish [J]. Freshwater Biology, 2021, 66(8): 1619-1629
[52] Rees H, Maddison B, Middleditch D J, et al. The detection of aquatic animal species using environmental DNA-A review of eDNA as a survey tool in ecology [J]. Journal of Applied Ecology, 2014, 51: 1450-1459
[53] Cantera I, Decotte J B, Dejean T, et al. Characterizing the spatial signal of environmental DNA in river systems using a community ecology approach [J]. Molecular Ecology Resources, 2022, 22(4): 1274-1283
[54] Itakura H, Wakiya R, Yamamoto S, et al. Environmental DNA analysis reveals the spatial distribution, abundance, and biomass of Japanese eels at the river-basin scale [J]. Aquatic Conservation Marine and Freshwater Ecosystems, 2019, 29(3): 313-361
[55] 孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究[J]. 生态毒理学报, 2018, 13(5): 76-86
Sun J Y, Yang J H, Zhang X W. Identification and biomass monitoring of zooplankton Cladocera species with eDNA metabarcoding technology [J]. Asian Journal of Ecotoxicology, 2018, 13(5): 76-86 (in Chinese)
[56] Sepulveda A, Schmidt C, Amberg J, et al. Adding invasive species biosurveillance to the US Geological Survey streamgage network [J]. Ecosphere, 2019, 10(8): e02843
[57] Stewart K A. Understanding the effects of biotic and abiotic factors on sources of aquatic environmental DNA [J]. Biodiversity and Conservation, 2019, 28(5): 983-1001
[58] Lacoursière-Roussel A, Rosabal M, Bernatchez L. Estimating fish abundance and biomass from eDNA concentrations: Variability among capture methods and environmental conditions [J]. Molecular Ecology Resources, 2016, 16(6): 1401-1414
[59] Francesco F G, Pierre T, Eric C. How to limit false positives in environmental DNA and metabarcoding? [J]. Molecular Ecology Resources, 2016, 16(3): 604-607
[60] Doi H, Fukaya K, Oka S I, et al. Evaluation of detection probabilities at the water-filtering and initial PCR steps in environmental DNA metabarcoding using a multispecies site occupancy model [J]. Scientific Reports, 2019, 9(1): 3581
[61] Tréguier A, Paillisson J M, Dejean T, et al. Environmental DNA surveillance for invertebrate species: Advantages and technical limitations to detect invasive crayfish “Procambarus clarkii” in freshwater ponds [J]. Journal of Applied Ecology, 2014, 51(4): 871-879
[62] Currier C A, Morris T J, Wilson C C, et al. Validation of environmental DNA (EDNA) as a Detection tool for at-risk freshwater pearly mussel species (Bivalvia: Unionidae) [J]. Aquatic Conservation: Marine and Freshwater Ecosystems, 2018, 28(3): 545-558
[63] Thomsen P F, Sigsgaard E E. Environmental DNA metabarcoding of wild flowers reveals diverse communities of terrestrial arthropods [J]. Ecology and Evolution, 2019, 9(4): 1665-1679
[64] Shogren A J, Tank J L, Andruszkiewicz E, et al. Controls on eDNA movement in streams: Transport, retention, and resuspension [J]. Scientific Reports, 2017, 7(1): 5065
[65] Carraro L, Hartikainen H, Jokela J, et al. Estimating species distribution and abundance in river networks using environmental DNA [J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(46): 11724-11729
[66] Beng K C, Corlett R T. Applications of environmental DNA (eDNA) in ecology and conservation: Opportunities, challenges and prospects [J]. Biodiversity and Conservation, 2020, 29(7): 2089-2121
[67] Rose J P, Wademan C, Weir S, et al. Traditional trapping methods outperform eDNA sampling for introduced semi-aquatic snakes [J]. PLoS One, 2019, 14(7): e0219244
[68] Sales N G, Wangensteen O S, Carvalho D C, et al. Space-time dynamics in monitoring neotropical fish communities using eDNA metabarcoding [J]. The Science of the Total Environment, 2021, 754: 142096