蜜蜂(Apis)是重要的授粉昆虫,对农业生产和生态环境具有不可忽视的意义。据报道,全球75%的作物和88%的开花植物都依赖蜜蜂等昆虫授粉达到繁殖目的[1-3]。随着世界各地频繁出现的蜜蜂中毒和数量急剧下降现象,人们对各种可能引发蜂群失衡甚至崩溃的因素给予了极大关注,其中农药对蜜蜂健康与生存的威胁一直是研究的热点问题。
乙虫腈(ethiprole)是一种新型苯基吡唑杀虫剂,与氟虫腈结构相似[4]。乙虫腈对多种刺吸式和咀嚼式害虫具有很好的防效,中国、巴西和越南等多个国家在水稻、玉米和大豆等粮食作物的生产和储存上都有广泛使用[5-6]。据环洋市场咨询(Global Info Research, GIR)调研,2005年,全球乙虫腈的销量已达6 600万美元,2021年销量达近1亿美元[7]。
除良好的生物活性外,乙虫腈在各种环境中稳定,不易降解,环境风险亦不容忽视[8]。据报道,乙虫腈可诱导多种非靶标生物的发育缺陷、免疫作用紊乱以及繁殖异常[9]。亚致死浓度的乙虫腈可显著抑制蜜蜂幼虫的化蛹和羽化率,导致发育失败[10];还显著影响意大利蜜蜂成年工蜂多种免疫基因的表达[11]。
DNA甲基化是研究较深入的表观遗传修饰。5-甲基胞嘧啶(5mC)是动植物等真核生物DNA甲基化的主要形式[12]。5mC甲基化是通过DNA甲基转移酶(DNA methyltransferase, Dnmt)进行的,包括Dnmt1、Dnmt2和Dnmt3。其中Dnmt1可变剪接产生的Dnmt1a和Dnmt1b参与DNA甲基化的维持[13-14];Dnmt2是真核生物中最为保守的甲基转移酶[15];Dnmt3主要参与从头甲基化[16]。蜜蜂具有完整的DNA甲基转移酶系统,在调控蜜蜂生长发育过程中发挥重要作用[17-18]。此外,6-甲基腺嘌呤(6mA)也是近年来新兴研究的DNA修饰,果蝇中的DNA 6mA去甲基化酶DMAD能调控胚胎发生[19],蜜蜂中6mA研究尚属空白。
与DNA相似,RNA上亦存在化学修饰,迄今已发现了100多种形式[20],在真核生物的基因表达调控中发挥着重要作用。N6-甲基腺嘌呤(m6A)甲基化是真核生物mRNA中最丰富的修饰之一,是当今RNA甲基化研究的热点[21]。RNA的m6A位点由甲基化阅读蛋白(YTHDC、YTHDF等)识别,经甲基转移酶复合体(METTL3、METTL14和WTAP等)和去甲基化酶(ALKBH5等)动态调控[22-24],从而实现对生物体生长发育的控制。然而,在蜜蜂中RNA m6A甲基化相关基因尚未进行完整的功能验证,通过与果蝇序列的同源比对可知,蜜蜂具有全部的m6A甲基化酶和阅读蛋白基因,但相关功能尚未完整鉴定。除m6A外,2’-O-甲基腺苷(Am)[25]、5-甲基胞嘧啶(m5C)[26]和3-甲基胞苷(m3C)[27]等也是较为常见的RNA甲基化修饰。报道显示其具有响应外界环境刺激[28]和调控胚胎发育[29]等功能。此外,通过与果蝇序列的同源比对发现蜜蜂具有m5C甲基转移酶(NOP2、NSUN2等)的同源基因[30],但其功能尚未鉴定。
农药暴露会影响生物的核酸甲基化水平[31],因此核酸甲基化可以作为表征农药危害的标志物。虽然目前关于不同农药对蜜蜂的毒性研究报道较多,但在核酸甲基化水平上对农药评估的研究还很少,核酸甲基化标志物和标签基因的开发尚属空白。因此,本研究以乙虫腈为研究对象,从核酸甲基化水平研究其对意大利蜜蜂(Apis mellifera ligustica,简称意蜂)工蜂幼虫的暴露风险,一方面完善乙虫腈对蜜蜂的环境风险评价,一方面探索核酸甲基化在未来农药蜜蜂风险评估工作中的应用潜力。
1 材料与方法(Materials and methods)
1.1 试验生物与材料
意蜂幼虫由中国农业科学院蜜蜂研究所提供。乙虫腈(纯度≥97%)购自德国拜耳公司,葡萄糖和果糖购自中国上海玉博生物科技有限公司,酵母提取物购自英国OXOID公司;DEPC水购自中国上海生工生物工程股份有限公司,丙酮(分析纯)和PBS缓冲液购自中国北京化学瑞金公司;氯仿、异丙醇和乙醇(纯度≥99.7%)购自中国上海麦克林生化科技有限公司;DNase Ⅰ、Nuclease P1和虾碱性磷酸酶购自中国NEB(北京)有限公司;昆虫DNA提取试剂盒购自美国Omega Bio-tek,Trizol试剂购自中国广州美基生物科技有限公司;cDNA合成试剂盒购自中国北京全式金生物技术有限公司;Dynabeads mRNA Purification Kit购自购自美国Invitrogen公司,PowerUp SYBR Green Master Mix、Qubit®RNA HS Assay Kits购自赛默飞世尔科技(中国)有限公司;甲基化标准品购自美国Sigma公司和MedChemExpress公司;引物由中国华大基因生物有限公司合成。
1.2 试验仪器与设备
液相色谱-三重四级杆串联质谱联用仪(LC-QqQ-MS/MS)(Agilent 6495,Agilent Technologies,美国)、PCR仪(Biometra Tone 96G,Eppendorf,德国)、荧光实时定量PCR仪(A40425,Thermo Fisher,美国)、冷冻离心机(Centrifuge 5424R,Eppendorf,德国)、超低温冰箱(DW-86L626,青岛海尔生物医疗股份有限公司,中国)、NanoDrop核酸含量检测分光光度计(NanoDrop 2000,Thermo Fisher,美国)、Qubit 4微量核酸检测分光光度计(Q33226,Thermo Fisher,美国)、研磨器(OSE-Y50,北京天根生化科技有限公司,中国)、恒温恒湿培养箱(PQX-350H,中仪国科科技有限公司,中国)。
1.3 乙虫腈对工蜂幼虫的暴露
文献报道显示,乙虫腈对意蜂的急性接触半数致死剂量(48 h-LD50)为0.0187 μg·蜂-1[32]。以此为参考,结合课题组前期研究结果[10],设置工蜂幼虫的暴露浓度为10-5、10-4、10-3和10-2mg·L-1。
工蜂幼虫饲养和暴露处理方法参照经济合作与发展组织(OECD)颁布的“化学品测试准则NO.237”和本实验室已发表的方法[10, 33]进行。实验开始前,将一张空巢脾放入蜂群,使用限王产卵框,限制蜂王在巢脾上产卵。3 d后,挑取脾中孵化的1日龄幼虫,转移到有20 μL饲料的48孔组织培养板中,2日龄的幼虫不饲喂饲料,3日龄和4日龄分别饲喂20 μL和30 μL含乙虫腈的饲料,5日龄和6日龄分别饲喂40 μL和50 μL含乙虫腈饲料。培养板放入(34.5±0.5)℃、(95±5)% RH的恒温恒湿培养箱中培养。每天补充新鲜饲料,若发现无食物消耗、没有移动或触后无反应的幼虫,则判定为死亡,记录死亡数后清除。
暴露结束时,收集7日龄幼虫,经PBS(pH 7.4)溶液冲洗,无菌滤纸吸干表面水分后,立即液氮速冻,-80 ℃冷冻保存待分析。
1.4 利用LC-MS/MS检测核酸甲基化
将收集的工蜂幼虫样品加液氮研磨粉碎。一部分研磨粉碎的样品利用Trizol法提取总RNA。保留一部分总RNA,另一部分总RNA使用Dynabeads mRNA Purification Kit分离mRNA。另一部分研磨粉碎的样品使用Insect DNA Kit提取DNA。用Nuclease P1和虾碱性磷酸酶酶解总RNA和mRNA,使用DNase Ⅰ和虾碱性磷酸酶酶解DNA。酶解液离心后转移上清至进样瓶,使用实验室建立的LC-MS/MS方法进行检测[34]。计算样品RNA、mRNA上m6A(m6A/A)、Am(Am/A)、m5C(m5C/C)、m3C(m3C/C)甲基化总量和样品DNA上6mA(6mA/dA)、5mC(5mC/dC)甲基化总量。
1.5 实时荧光定量PCR检测核酸甲基化相关基因表达
为验证乙虫腈对蜜蜂幼虫核酸甲基化的影响,采用RT-qPCR技术测定了DNA和RNA甲基化酶基因(基因引物序列见表1)在不同浓度乙虫腈暴露后的表达情况。用Trizol试剂提取的总RNA,按照cDNA合成试剂盒步骤合成cDNA,使用PowerUp SYBR Green Master Mix配制反应体系。在QuantStudio 1系统上对目标基因进行RT-qPCR。选取AmActin为内参基因,利用ΔΔCT法计算各目标基因的相对表达量。
表1 RT-qPCR引物
Table 1 RT-qPCR primers

引物名称Primer name碱基序列(5’~3’)Primer sequence (5’~3’)AmActin-FGTTTTCCCATCTATCGTCGGAmActin-RTTTTCTCCATATCATCCCAGAmDnmt1a-FACTCGAGTAGTAAGCGTGCGAmDnmt1a-RACTCCAAGTGGTGGAGGAACAmDnmt1b-FGCGAGCATGTCAACTAAGCGAmDnmt1b-RATACGGTGCTGATTCCGTCCAmDnmt2-FATGAGTCCTCCATGTCAACCTTAmDnmt2-RTGCCAAATTGACAAGGGCTTAGAmDnmt3-FACTCGAATGTGGAACACCTGGAmDnmt3-RGTCTTGGTCTATCTCGCTCGCAmMETTL3-FGACGAATCCTTGGGTGACTGTAmMETTL3-RTCCACTTCATAATGTACATACTTGCAmMETTL14-FACTTGCTCAGACGCTAGGTGAmMETTL14-RTATCCGCCAAACCCACATCCAmWTAP-FGGACAGTGCGTGTGTGTTTGAmWTAP-RCAGGGATGGTTTGTTTCACCAAmYTHDC1-FTGAAGAACGAGTGTGCACAAAAAmYTHDC1-RTTCGCCGCAGCTCAGATTTAAmYTHDF-FGGATGCACAGGAAGTCCCTCAmYTHDF-RCCACCATCTCGATCTTTGCGAmALKBH1-FCATACCGACCATTCCGAACCTAmALKBH1-RTCCTCTTGCATAAGTCCCCCAAmALKBH4-FTGATTGCTGGATTTGGGGTGAAmALKBH4-RTCCTTCTGGCATTGGGAGTCAmALKBH6-FGTGTTCCTTTGCCAAAATGGACTAmALKBH6-RGGATGTGGAATACCACCCCAAAmALKBH8-FAGCTCGTTATGCATGGTCACAmALKBH8-RACAACAACAATCACCCCTGTGAmNOP2-FCGCGTCGTCGTGATTTAGCAmNOP2-RCACCCATGGGAACTTGCGATAmNSUN2-FCCACCAAAACCTGAGGATGCAmNSUN2-RTGATGCACGTTCCCAAGGTAAmNSUN4-FTTCAATCTGAGATTTTAGCGAAmNSUN4-RGCTACTTGTACAACACCATCAmNSUN5-FATGTGTGCAGCTCCTGGAATAmNSUN5-RAGTTTCGACACAAGAAGCGTT
1.6 数据处理
核酸甲基化含量(%)数据先进行反正弦平方根转换后,再用Graphpad prism 8.0软件的单因素方差分析(One-way ANOVA)中Tukey’s HSD检验比较处理组与对照组之间的差异,用相对定量法比较目标基因的表达差异。文中*表示P<0.05,差异显著;**表示P<0.01,差异显著。每个处理设置3个生物学重复,每个生物学重复进行3次技术重复。
2 结果(Results)
2.1 乙虫腈暴露对工蜂幼虫DNA甲基化及其相关基因表达的影响
首先采用LC-MS/MS对乙虫腈暴露的工蜂幼虫中2个主要的DNA甲基化6mA和5mC进行了分析。结果发现,在设置的4个亚致死剂量下,乙虫腈对工蜂幼虫DNA 6mA的含量没有显著影响,各处理组间DNA 6mA甲基化水平差异不显著(图1(a))。与6mA变化不同,乙虫腈导致各处理组中幼虫DNA 5mC的含量呈现下降趋势,且10-3mg·L-1处理组下降显著(P<0.05)(图1(b))。这表明亚致死剂量乙虫腈暴露可引起DNA 5mC甲基化的响应变化。
为进一步探明乙虫腈暴露引发的DNA甲基化变化,对4种主要DNA 5mC甲基化酶基因的表达进行了检测。结果显示(图1(c)),工蜂幼虫AmDnmt1a和AmDnmt1b的基因表达趋势相同,与对照组相比,AmDnmt1a和AmDnmt1b表达在最高浓度(10-2mg·L-1)乙虫腈暴露下显著上调(P<0.01)。AmDnmt2基因的表达水平仅在10-3mg·L-1处理组中显著降低;AmDnmt1a、AmDnmt1b和AmDnmt2基因的表达变化与5mC甲基化含量变化一致。值得注意的是,在所有暴露浓度中,AmDnmt3基因表达比对照上调了1.46倍~5.41倍,差异显著(P<0.01),表现出其对乙虫腈暴露的灵敏响应。
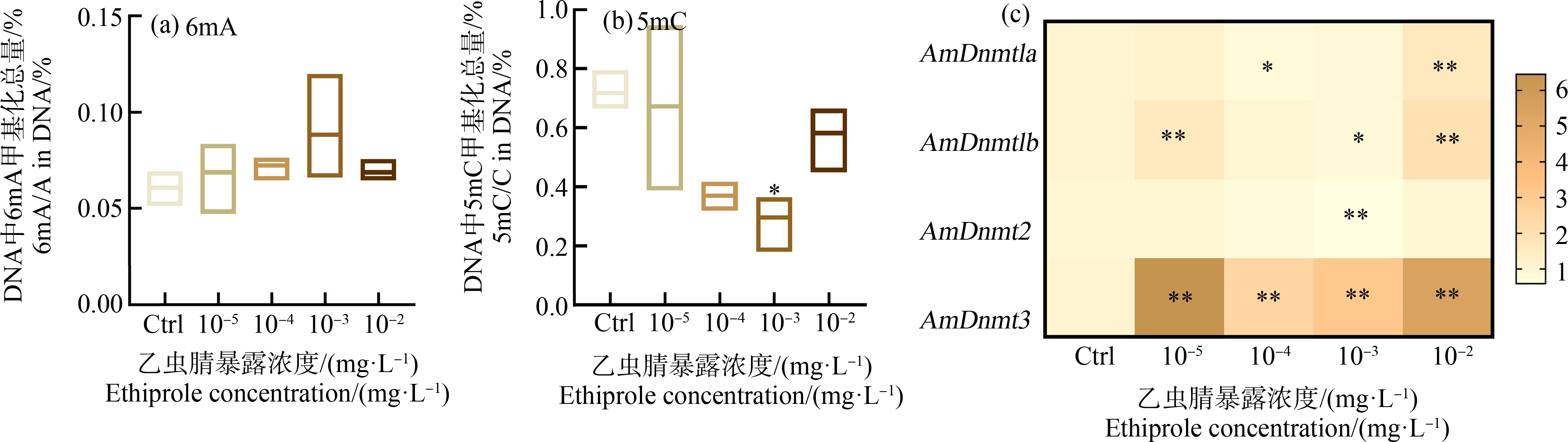
图1 乙虫腈对意蜂工蜂幼虫DNA甲基化水平及相关基因表达的影响
注:(a)DNA 6mA甲基化水平,(b)DNA 5mC甲基化水平,(c)DNA 5mC甲基转移酶相关基因的表达;(c)图右侧标尺表示相对表达量数值;*,P<0.05,代表对照组与处理组之间差异显著;**,P<0.01,代表对照组与处理组之间差异显著;下同。
Fig.1 Effects of ethiprole on DNA methylation and gene expression of Apis mellifera ligustica worker larvae
Note:(a)the methylation levels of DNA 6mA,(b)the methylation levels of 5mC,(c)the gene expression of DNA 5mC methyltransferase; the scale on right panel shows relative expression level; *, P<0.05, indicating significant difference between the control and the treatment group; **, P<0.01, indicating extremely significant difference between the control and the treatment group; the same below.
2.2 乙虫腈暴露对工蜂幼虫总RNA甲基化及其相关基因表达的影响
对蜜蜂幼虫总RNA的m6A、Am、m5C和m3C甲基化进行了LC-MS/MS测定,结果表明,在所有乙虫腈处理组中,腺嘌呤m6A和Am甲基化的含量均低于对照组,且在高浓度10-2mg·L-1处理组中显著降低(P<0.05)(图2(a)和2(b))。与之相反,在所有乙虫腈处理组中,胞嘧啶m5C和m3C甲基化的含量都显著增高(P<0.05)(图2(c)和2(d))。以上结果表明,与DNA甲基化相比,RNA甲基化响应乙虫腈暴露风险更灵敏。
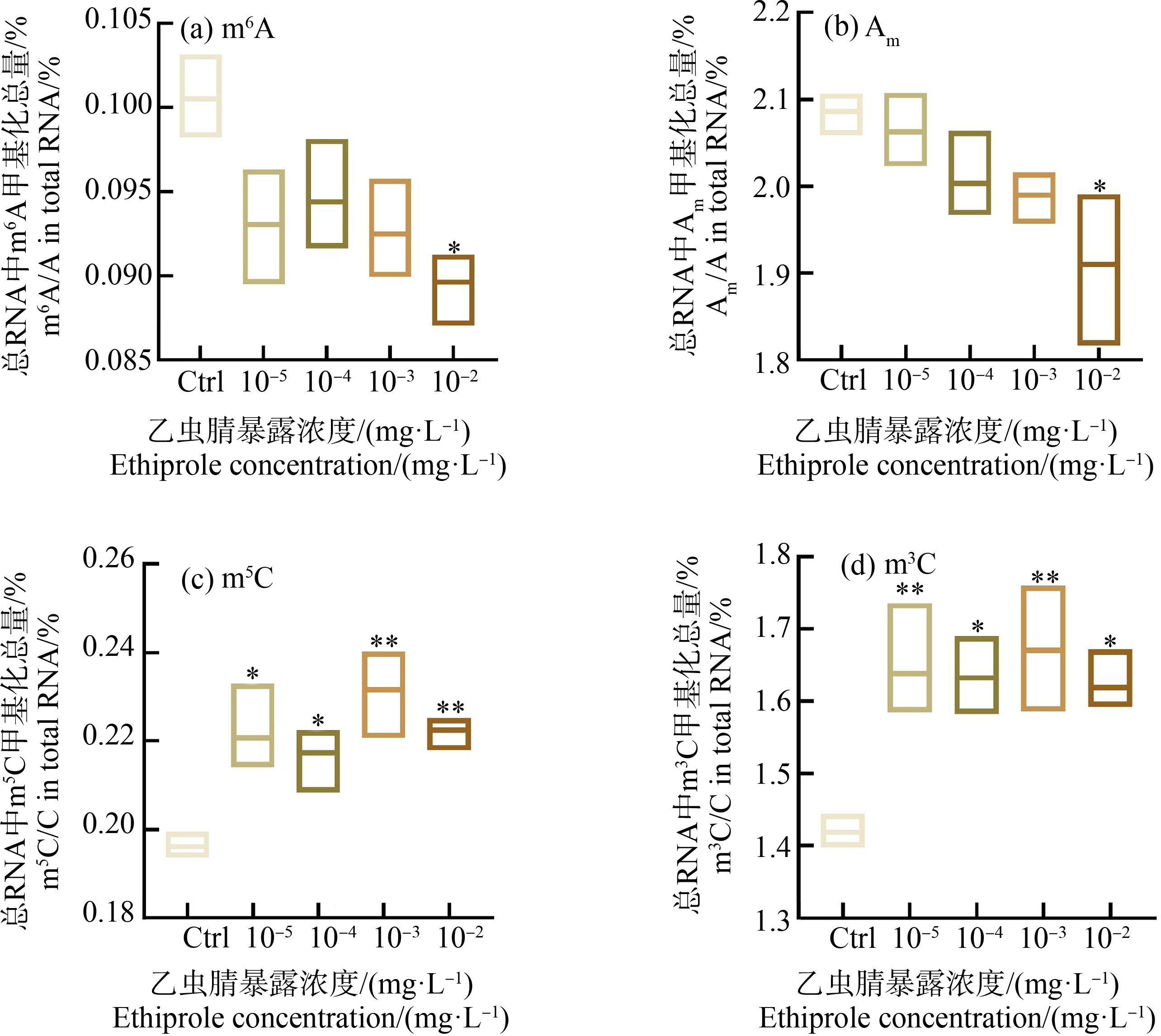
图2 乙虫腈对意蜂工蜂幼虫总RNA上的m6A(a)、Am(b)、m5C(c)和m3C(d)甲基化水平的影响
Fig. 2 Effects of ethiprole on the methylation level of m6A(a)、Am(b)、m5C(c)and m3C(d)in total RNA in Apis mellifera ligusticaworker larvae
2.3 乙虫腈暴露对工蜂幼虫mRNA甲基化水平及其相关基因表达的影响
为了进一步验证RNA甲基化对乙虫腈暴露的响应,对mRNA上甲基化修饰的变化进行了检测。结果发现,在所有浓度乙虫腈处理中,腺嘌呤上的甲基化m6A和Am含量均显著增高(图3(a)和3(b)),其中m6A甲基化水平显著增高1.1倍~1.8倍(P<0.01)。这说明mRNA m6A和Am甲基化对乙虫腈暴露风险响应灵敏。
进一步对m6A甲基化相关基因,包括3个主要的甲基化酶基因AmMETTL3、AmMETTL14和AmWTAP,2个甲基化阅读蛋白基因AmYTHDC1和AmYTHDF,以及4个去甲基化酶基因AmALKBH1、AmALKBH4、AmALKBH6和AmALKBH8的表达情况进行了检测。结果显示,AmMETTL3、AmMETTL14、AmYTHDC1和AmYTHDF基因都在10-5mg·L-1和10-2mg·L-1处理组中显著上调;除AmMETTL14表达在10-4mg·L-1浓度处理组显著下调外,其他3个基因在10-3mg·L-1和10-4mg·L-1处理组中的表达水平差异均不显著。AmWTAP基因在乙虫腈各处理组中的表达均低于对照组,且在2个较低浓度(10-5mg·L-1和10-4mg·L-1)组中下调显著(P<0.01,图3(c))。对于4个ALKBH家族的甲基化酶基因,AmALKBH4、AmALKBH6和AmALKBH8的表达变化类似,都在10-5mg·L-1处理组中显著上调,在10-4mg·L-1处理组中显著下调,而在较高的2个浓度(10-3mg·L-1和10-2mg·L-1)中表达略有波动,但与对照组相比差异不显著。值得注意的是,在所有乙虫腈处理浓度中,AmALKBH1基因均显著下调(P<0.01),在高浓度(10-3mg·L-1和10-2mg·L-1)中未能检测到表达量(图3(d)),说明该甲基化酶基因对乙虫腈非常敏感。
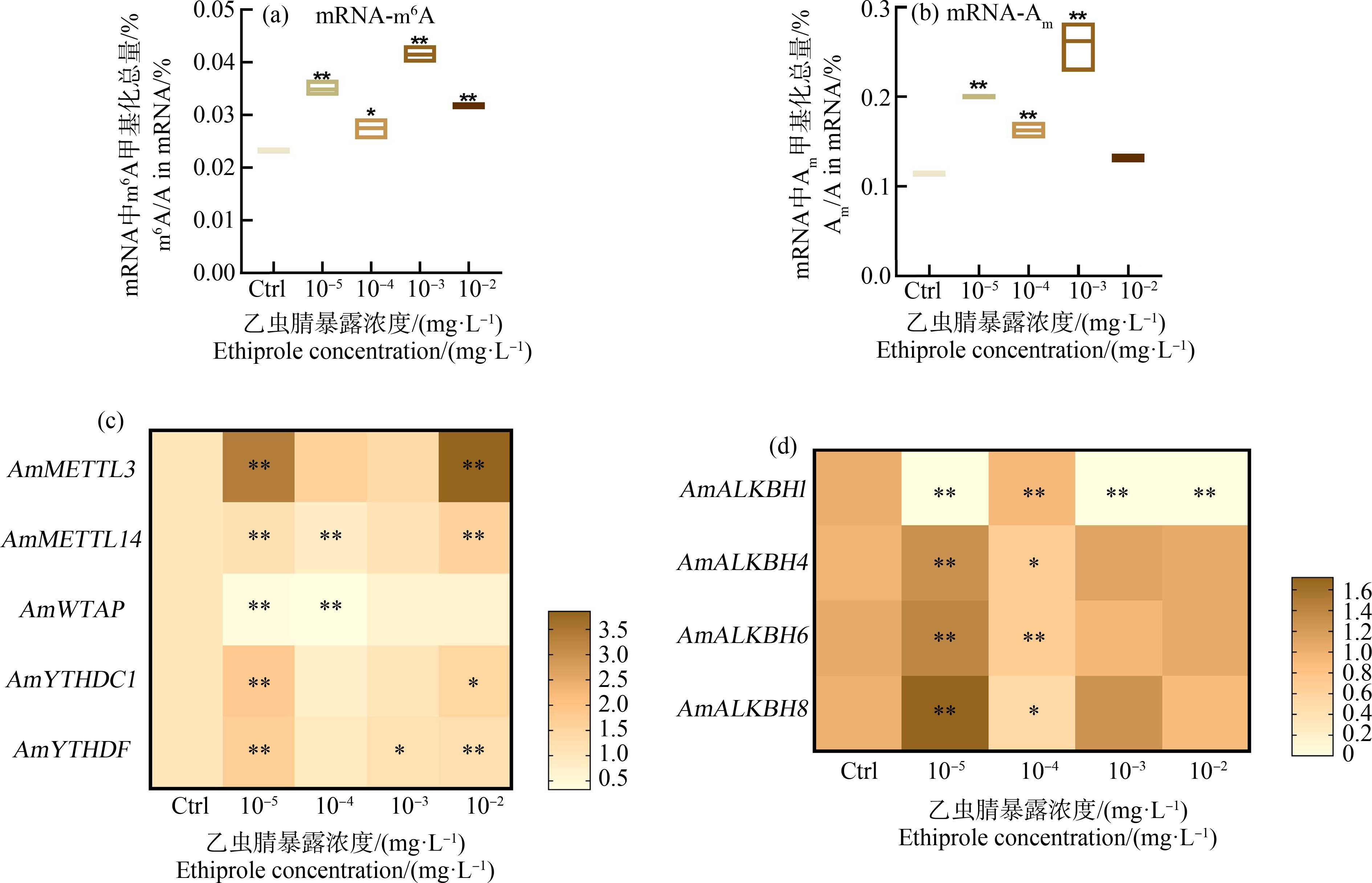
图3 乙虫腈对mRNA m6A和Am甲基化水平及m6A甲基化及相关基因表达的影响
注:(a)m6A甲基化水平,(b)Am甲基化水平,(c)m6A甲基转移酶基因和m6A阅读蛋白基因,(d)m6A去甲基化酶基因。
Fig.3 Effects of ethiprole on the mRNA m6A and Ammethylation and the related gene expression
Note:(a)m6A methylation,(b)Ammethylation,(c)gene expression of m6A methyltransferase, m6A binding proteins,(d)gene expression of m6A demethylase.
对于mRNA胞嘧啶上的甲基化m3C和m5C,我们发现m3C含量在所有乙虫腈处理组中均显著高于对照组(图4(b)),约增高了1.95倍~13.3倍。而m5C甲基化水平在所有处理组中差异不显著(图4(a))。此外,对4种主要的mRNA m5C甲基化酶基因的表达检测发现,在所有乙虫腈处理组中,NSUN4的表达量均显著上调(P<0.01);NSUN2在10-5mg·L-1和10-4mg·L-1处理组中的表达显著上调(P<0.05),在10-2mg·L-1处理组中显著上调(P<0.01);NSUN4表达在所有乙虫腈处理组中的表达均显著上调(P<0.01),且与对照相比上调了1.7倍~3.4倍;NSUN5的表达在10-5mg·L-1处理组中显著上调(P<0.05),在10-3mg·L-1和10-4mg·L-1处理组中上调显著(P<0.01)(图4(c));NOP2在10-5mg·L-1和10-2mg·L-1处理组中表达显著上调(P<0.05)。
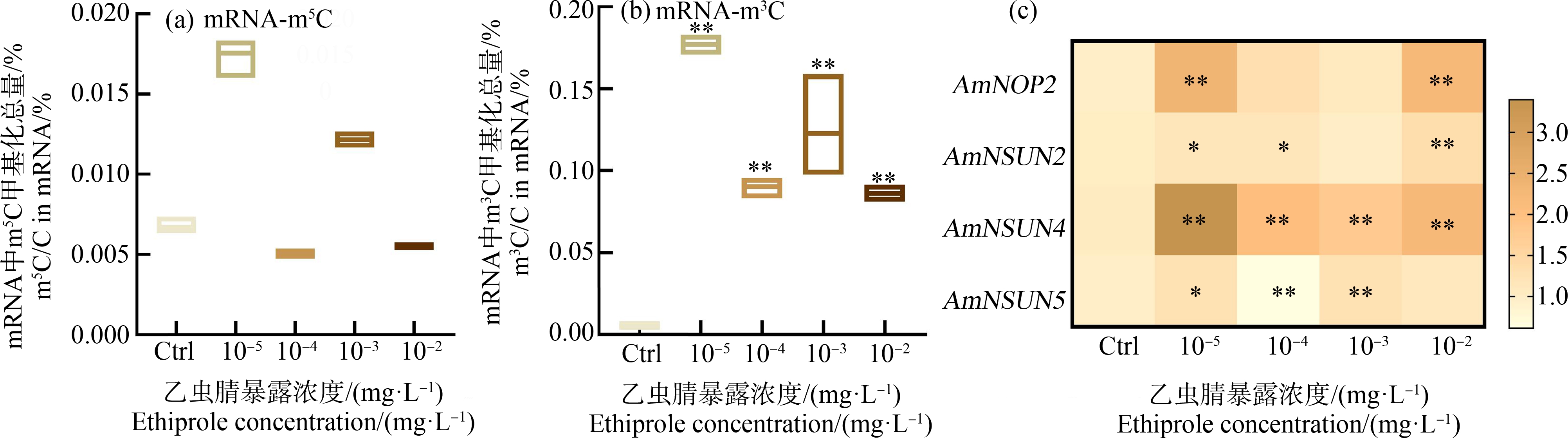
图4 乙虫腈对mRNA m5C和m3C甲基化水平及相关基因表达的影响
注:(a)m5C甲基化水平,(b)m3C甲基化水平,(c)m5C甲基转移酶基因。
Fig.4 Effects of ethiprole on the mRNA m5C and m3C methylation and the related gene expression
Note:(a)m5C methylation,(b)m3C methylation,(c)gene expression of m5C methyltransferase.
3 讨论(Discussion)
鉴于蜜蜂的生物学特性,蜜蜂以及蜂群整个暴露在环境中,可直接或间接接触环境中的残留农药。现阶段农药对蜜蜂的风险评估研究大多集中在成年蜜蜂上,对蜜蜂幼虫的关注还较少。蜜蜂幼虫是蜂群生存繁衍繁盛的基础,依赖于蜂群中成年工蜂的饲喂实现生长发育,而当幼虫长期接触或摄入含有残留农药的环境或食物时,残留农药可能在幼虫体内蓄积,进而造成幼虫的畸形甚至死亡,严重的可诱发蜂群崩溃失调。蜜蜂幼虫对农药胁迫响应灵敏,如Zhu等[35]研究发现蜜蜂幼虫对百菌清的敏感性高于成虫,蜜蜂幼虫暴露于1.44 ng·μL-1的噻虫嗪可导致突触蛋白水平下降,且成蜂可产生显著的神经生理障碍[36]。我们前期研究也发现亚致死剂量的氟虫腈可显著降低幼虫的化蛹率和羽化率,导致幼虫发育失败[37],而亚致死剂量的乙虫腈则可诱导蜜蜂幼虫发生氧化应激和免疫反应[10]。这些都说明蜜蜂幼虫对农药胁迫敏感,且早期的暴露风险可导致后期发育和身体机能的损害,蜜蜂幼虫可作为评估农药环境风险的敏感昆虫。
DNA甲基化已开发为一种生物标志物,用于判别各种风险因子对生物及人类健康的影响[38]。在本研究中发现乙虫腈暴露可显著诱导蜜蜂幼虫的DNA甲基化响应,DNA水平上的5mC和6mA甲基化发生了变化(图5)。进一步研究发现RNA甲基化对乙虫腈的响应比DNA更灵敏。DNA甲基化对转录过程进行调控,而RNA甲基化主要发挥转录后调控作用,因此推测乙虫腈对转录后过程影响更大。

图5 DNA和RNA甲基化及相关基因响应乙虫腈暴露模型
Fig. 5 Model of epigenetic marks and related genes responding to ethiprole exposure
然而,RNA甲基化作为环境危害因子指示物的研究刚刚起步,目前只有报道显示RNA修饰能够响应外界环境因子的刺激[39-40],在蜜蜂上的研究应用还很少。我们的研究结果首次表明各亚致死浓度乙虫腈暴露均可显著提高意蜂工蜂幼虫总RNA的m5C、m3C甲基化含量,以及mRNA m6A和m3C甲基化水平(图5),这些结果一方面表明了乙虫腈对蜜蜂幼虫的暴露胁迫,另一方面证明了RNA甲基化对外界胁迫的灵敏响应,可作为生物指标指示乙虫腈等农药对蜜蜂的胁迫风险。
本研究中,乙虫腈对蜜蜂幼虫核酸甲基化含量的影响与对相关基因表达的影响并不完全一致,这是因为甲基化水平调控是多种基因协同作用的结果。文中检测的多个靶标基因均对乙虫腈暴露有不同程度的响应(图5),其中Dnmt3和NSUN4在设置的4个乙虫腈处理组中均有强烈上调,ALKBH1则下调,此3个基因未来可开发为乙虫腈风险评估的标签基因。值得注意的是,ALKBH1对乙虫腈暴露的敏感性显著高于其他目标基因,在10-3mg·L-1和10-2mg·L-1浓度下则几乎完全难以检测到表达,前人研究表明ALKBH1主要参与外界环境因子对机体的损伤修复[41],而在昆虫上的功能研究尚属空白。未来可对ALKBH1基因的功能和对农药响应的风险水平及特异性进行挖掘。
综合上述研究结果可知,乙虫腈虽是对各种害虫具有良好防效的苯基咪唑类杀虫剂,但亚致死剂量的乙虫腈依然对蜜蜂具有不同程度的风险,且可显著诱导蜜蜂幼虫的核酸甲基化反应,因此仍需对其环境风险性进行细致的评估;此外Dnmt3、ALKBH1和NSUN4基因对乙虫腈暴露响应灵敏,可作为杀虫剂暴露风险的标志基因。本研究填补了乙虫腈对蜜蜂幼虫核酸甲基化影响研究的空白,有望开发全新的风险评估标志物和技术手段,为乙虫腈等农药的风险评估和科学使用提供理论基础和技术支撑。
[1] Potts S G, Imperatriz-Fonseca V, Ngo H T, et al.Safeguarding pollinators and their values to human well-being[J].Nature, 2016, 540(7632): 220-229
[2] Ollerton J, Winfree R, Tarrant S.How many flowering plants are pollinated by animals?[J].Oikos, 2011, 120(3): 321-326
[3] Potts S G, Biesmeijer J C, Kremen C, et al.Global pollinator declines: Trends, impacts and drivers[J].Trends in Ecology &Evolution, 2010, 25(6): 345-353
[4] 李定银, 郅军锐, 张涛, 等.乙基多杀菌素和乙虫腈对西花蓟马解毒酶和乙酰胆碱酯酶活性的影响[J].应用昆虫学报, 2020, 57(6): 1385-1393
Li D Y, Zhi J R, Zhang T, et al.Effects of spinetoram and ethiprole on detoxification enzyme and acetylcholin esterase activity in Frankliniella occidentalis(Pergande)[J].Chinese Journal of Applied Entomology, 2020, 57(6): 1385-1393(in Chinese)
[5] Punyawattoe P, Han Z J, Sriratanasak W, et al.Ethiprole resistance in Nilaparvata lugens(Hemiptera: Delphacidae): Possible mechanisms and cross-resistance[J].Applied Entomology and Zoology, 2013, 48(2): 205-211
[6] Elzaki M A, Zhang W, Han Z.Cytochrome P450 CYP4DE1 and CYP6CW3v2 contribute to ethiprole resistance in Laodelphax striatellus(Fallén)[J].Insect Molecular Biology, 2015, 24(3): 368-376
[7] Global Info Research.Global ethiprole market 2022 by manufacturers, regions, type and application, forecast to 2022[R].Hongkong: Global Info Research, 2022
[8] Liu X G, Dong F S, Xu J, et al.Dissipation and adsorption behavior of the insecticide ethiprole on various cultivated soils in China[J].Journal of Integrative Agriculture, 2014, 13(11): 2471-2478
[9] Tanaka T, Suzuki T, Inomata A.Reproductive and neurobehavioral effects of maternal exposure to ethiprole in F1-generation mice[J].Birth Defects Research, 2018, 110(3): 259-275
[10] Liu Y Y, Wang C, Qi S Z, et al.The sublethal effects of ethiprole on the development, defense mechanisms, and immune pathways of honeybees(Apis melliferaL.)[J].Environmental Geochemistry and Health, 2021, 43(1): 461-473
[11] 牛新月, 齐素贞, 吴黎明, 等.乙虫腈悬浮剂对新出房意大利蜜蜂的毒性研究[J].生态毒理学报, 2019, 14(3): 203-213
Niu X Y, Qi S Z, Wu L M, et al.Toxicity studies of ethiprole suspension concentrate to newly emerged honey bees(Apis melliferaL.)[J].Asian Journal of Ecotoxicology, 2019, 14(3): 203-213(in Chinese)
[12] Bohnsack K E, Höbartner C, Bohnsack M T.Eukaryotic 5-methylcytosine(m5C)RNA methyltransferases: Mechanisms, cellular functions, and links to disease[J].Genes, 2019, 10(2): 102
[13] Bonfils C, Beaulieu N, Chan E, et al.Characterization of the human DNA methyltransferase splice variant Dnmt1b[J].Journal of Biological Chemistry, 2000, 275(15): 10754-10760
[14] Castillo P, Ibá ez F, Guajardo A, et al.Impact of cadmium exposure during pregnancy on hepatic glucocorticoid receptor methylation and expression in rat fetus[J].PLoS One, 2012, 7(9): e44139
ez F, Guajardo A, et al.Impact of cadmium exposure during pregnancy on hepatic glucocorticoid receptor methylation and expression in rat fetus[J].PLoS One, 2012, 7(9): e44139
[15] Ashapkin V V, Kutueva L I, Vanyushin B F.Dnmt2 is the most evolutionary conserved and enigmatic cytosine DNA methyltransferase in eukaryotes[J].Russian Journal of Genetics, 2016, 52(3): 237-248
[16] Chen Z Y, Zhang Y.Role of mammalian DNA methyltransferases in development[J].Annual Review of Biochemistry, 2020, 89: 135-158
[17] Cardoso-Júnior Jr, Guidugli-Lazzarini K R, Hartfelder K.DNA methylation affects the lifespan of honey bee(Apis melliferaL.)workers—Evidence for a regulatory module that involves vitellogenin expression but is independent of juvenile hormone function[J].Insect Biochemistry and Molecular Biology, 2018, 92: 21-29
[18] Wang Y, Jorda M, Jones P L, et al.Functional CpG methylation system in a social insect[J].Science, 2006, 314(5799): 645-647
[19] Zhang G Q, Huang H, Liu D, et al.N6-methyladenine DNA modification in Drosophila[J].Cell, 2015, 161(4): 893-906
[20] Nachtergaele S, He C.The emerging biology of RNA post-transcriptional modifications[J].RNA Biology, 2017, 14(2): 156-163
[21] Motorin Y, Helm M.RNA nucleotide methylation[J].Wiley Interdisciplinary Reviews RNA, 2011, 2(5): 611-631
[22] Meyer K D, Jaffrey S R.Rethinking m6A readers, writers, and erasers[J].Annual Review of Cell and Developmental Biology, 2017, 33: 319-342
[23] Yang Y, Hsu P J, Chen Y S, et al.Dynamic transcriptomic m6A decoration: Writers, erasers, readers and functions in RNA metabolism[J].Cell Research, 2018, 28(6): 616-624
[24] Shi H L, Wei J B, He C.Where, when, and how: Context-dependent functions of RNA methylation writers, readers, and erasers[J].Molecular Cell, 2019, 74(4): 640-650
[25] Mauer J, Luo X B, Blanjoie A, et al.Reversible methylation of m6Am in the 5’ cap controls mRNA stability[J].Nature, 2017, 541(7637): 371-375
[26] Delatte B, Wang F, Ngoc L V, et al.RNA biochemistry.Transcriptome-wide distribution and function of RNA hydroxymethylcytosine[J].Science, 2016, 351(6270): 282-285
[27] Li S B, Zhou H L, Liao S H, et al.Structural basis for METTL6-mediated m3C RNA methylation[J].Biochemical and Biophysical Research Communications, 2022, 589: 159-164
[28] Wang Y M, Li D Q, Gao J B, et al.The 2’-O-methyladenosine nucleoside modification gene OsTRM13 positively regulates salt stress tolerance in rice[J].Journal of Experimental Botany, 2017, 68(7): 1479-1491
[29] Cui W, Pizzollo J, Han Z B, et al.Nop2 is required for mammalian preimplantation development[J].Molecular Reproduction and Development, 2016, 83(2): 124-131
[30] Bataglia L, Simões Z P, Nunes F F.Active genic machinery for epigenetic RNA modifications in bees[J].Insect Molecular Biology, 2021, 30(6): 566-579
[31] Yang X, Wei X G, Yang J, et al.Epitranscriptomic regulation of insecticide resistance[J].Science Advances, 2021, 7(19): eabe5903
[32] 苍涛, 章虎, 王新全, 等.手性乙虫腈对3种非靶标生物的急性毒性及初步风险评价[J].农药学学报, 2016, 18(1): 65-70
Cang T, Zhang H, Wang X Q, et al.Acute toxicities and preliminary risk evaluation of chiral ethiprole to three non-target organisms[J].Chinese Journal of Pesticide Science, 2016, 18(1): 65-70(in Chinese)
[33] Organizationfor Economic Co-operation and Development(OECD).No.237, OECD guidelines for the testing of chemicals: Honey bee(Apis mellifera)larval toxicity test, single exposure[S].Paris: OECD, 2013
[34] Wang M, Xiao Y, Li Y, et al.RNA m6A modification functions in larval development and caste differentiation in honeybee(Apis mellifera)[J].Cell Reports, 2021, 34(1): 108580
[35] Zhu W Y, Schmehl D R, Mullin C A, et al.Four common pesticides, their mixtures and a formulation solvent in the hive environment have high oral toxicity to honey bee larvae[J].PLoS One, 2014, 9(1): e77547
[36] Tavares D A, Roat T C, Silva-Zacarin E C M, et al.Exposure to thiamethoxam during the larval phase affects synapsin levels in the brain of the honey bee[J].Ecotoxicology and Environmental Safety, 2019, 169: 523-528
[37] 李佳欢, 齐素贞, 吴黎明, 等.氟虫腈对意大利蜜蜂工蜂幼虫及幼龄工蜂的亚致死效应[J].生态毒理学报, 2021, 16(5): 314-325
Li J H, Qi S Z, Wu L M, et al.Sublethal effects of fipronil on larvae and young worker honey bees(Apis mellifera ligustica)[J].Asian Journal of Ecotoxicology, 2021, 16(5): 314-325(in Chinese)
[38] Peters A, Nawrot T S, Baccarelli A A.Hallmarks of environmental insults[J].Cell, 2021, 184(6): 1455-1468
[39] Xiang Y, Laurent B, Hsu C H, et al.RNA m6A methylation regulates the ultraviolet-induced DNA damage response[J].Nature, 2017, 543(7646): 573-576
[40] Heng J H, Tian M, Zhang W F, et al.Maternal heat stress regulates the early fat deposition partly through modification of m6A RNA methylation in neonatal piglets[J].Cell Stress &Chaperones, 2019, 24(3): 635-645
[41] Wakisaka K T, Muraoka Y, Shimizu J, et al.Drosophilaalpha-ketoglutarate-dependent dioxygenase AlkB is involved in repair from neuronal disorders induced by ultraviolet damage[J].Neuroreport, 2019, 30(15): 1039-1047