随着人民生活水平的提高,我国的畜禽及水产养殖业日益规模化,为了防治疾病,提高动物生产性能,越来越多的抗生素被用于日常饲喂中[1-2]。据统计,我国每年抗生素原料生产量约为21万t,其中9.7万t用于畜牧养殖业,占比为46.2%[3]。抗生素的使用虽然极大地促进了养殖业的发展,但由于抗生素在动物体内不能被完全由吸收途径降解,60%~90%以原药形式排出体外[4]。据统计,畜禽粪便的排放量从2016年的31.6亿t增加到了2021年的38亿t,并且仍在逐年增加[5],因此造成了生态环境中抗生素残留的点源及面源污染[6]。抗生素的长期大量使用诱导了耐药菌的产生,进而传播了一种新型环境污染物——抗生素抗性基因(antibiotics resistance genes, ARGs)。
目前研究发现,ARGs广泛存在于畜禽粪污[7]、养殖场周围[8-9]及长期畜禽粪便还田的土壤[10-12]、城市农贸市场的空气[13]、甚至饮用水系统[14]及蔬菜中[15-16]。其中,含量较多的为四环素类抗性基因、β-内酰胺类抗性基因、磺胺类抗性基因和喹诺酮类抗性基因等。携带ARGs的微生物菌株死亡后,携带ARGs的基因片段在脱氧核糖核酸酶的保护下依然能够持久存在于环境中,并通过水平基因转移(horizontal gene transfer, HGT),在不同微生物之间进行传递[17-19]。当环境中的ARGs位于基因转移单位时,可整合到诸如插入序列、质粒、转座子、整合子、基因岛和噬菌体等可移动遗传元件(mobile genetic elements, MGEs)上,然后通过转化、转导和接合等方式,在不同微生物物种间传播扩散[20-21],进而出现了“超级细菌”,对生态环境造成严重的威胁[22]。
研究表明,抗生素目前已经克隆的抗生素抗性相关基因有384个,高通量实时荧光定量聚合酶链式反应(high-throughput real-time quantitative polymerase chain reaction, HT-qPCR)弥补了传统qPCR的局限性,检测的通量更大,效率更高,可以同时准确定量384个抗生素抗性相关基因,是目前基因定量检测最为先进的技术。Zhu等[23]应用HT-qPCR技术在养猪场粪便、堆肥及周围施用粪肥农田土壤中共检到149种ARGs,基本涵盖目前的主要ARGs亚型,可以较为客观全面地反映出ARGs的多样性。Li等[19]应用HT-qPCR技术对东北黑土农田的ARGs进行了研究,发现畜禽粪便有机肥显著增加了土壤中ARGs和MGEs的数量和丰度。
农田中抗生素的主要来源为未经充分腐熟的畜禽粪污还田[24-26],因此,农田土壤中ARGs污染也成为了一种潜在风险,张俊华等[27]研究发现宁夏养牛场粪污中主要抗生素为四环素类、喹诺酮类和磺胺类,主要的ARGs为氨基糖胺类;李金阳[28]发现施加牛粪/堆肥显著增加了生菜根际土壤中ARGs的多样性。褐土占山西省总耕地面积的54.9%,是山西省粮食生产的重要基地,由于当地少雨缺水,粪肥还田显著提高了水分的利用率,而长期施用牛粪对褐土ARGs种类及丰度的影响还缺乏系统的研究,这在一定程度上阻碍的人们对农田土壤ARGs的全面客观的认识。本研究基于山西寿阳褐土长达30年的长期牛粪还田定位试验,研究不同牛粪用量对褐土中ARGs多样性的影响,本研究的结果将为农田系统中ARGs的风险评估提供一定的理论依据。
1 材料与方法(Materials and methods)
1.1 研究区概况
本长期定位试验位于山西省晋中市寿阳县宗艾镇宗艾村“国家农业环境寿阳观测实验站”(北纬37°58′23.0″、东经113°06′38″),海拔为1 130 m,试验地块基本平坦,土壤类型为褐土,试验地初始pH值为8.4,有机质为23.8 g·kg-1、碱解氮为117.69 mg·kg-1、有效磷为4.84 mg·kg-1、有效钾为100 mg·kg-1,其余条件见文献[29]。
本长期定位试验从1992年春开始种植春玉米,采用大田裂区方式分布,至2021年已历时29年。设置5个处理:CK(不施肥)、N2P1M1(N:120 kg·hm-2;P:37.5 kg·hm-2;牛粪:22 500 kg·hm-2)、N4P2M2(N:240 kg·hm-2;P:75 kg·hm-2;牛粪:45 000kg·hm-2)、N3P2M3(N:180 kg·hm-2;P:75 kg·hm-2;牛粪:67 500 kg·hm-2)和N0P0M6(牛粪:135 000kg·hm-2),氮肥种类为尿素,磷肥的种类为过磷酸钙,牛粪施用前预先自然堆腐(全磷1.37~1.46g·kg-1、有机质90.5~127.3 g·kg-1、全钾14.1~34.3 g·kg-1和全氮3.93~4.97 g·kg-1)。每个处理3个生物学重复,共15个小区,每个小区面积为66.7 m2,玉米品种为大丰30,播种密度为66 000株·hm-2,玉米播种前进行旋地和施肥,方式为全部播前基施。
1.2 样品采集
2021年6月,于各小区采用5点“S”法,用土钻采集0~20 cm的土壤样品,将同一小区的5个样混合为一个样品,去掉明显的石砾、残枝败叶等杂物,一部分风干用于土壤理化性质的检测,另一部分用无菌袋装好,将其置于冰盒带回实验室,进行土壤DNA的提取及后续HT-qPCR分析。
1.3 测定方法
1.3.1 土壤理化性质测定方法
土壤理化性质的测定方法均采用鲍士旦编著的《土壤农业化学分析方法》进行[30]。其中,pH采用酸度计检测,水土比为2.5∶1;碱解氮(alkali-hydrolysable nitrogen, AN)采用比色法检测;有效钾(available K, AK)采用1 mol·L-1乙酸铵火焰光度计检测;有效磷(available P, AP)采用NaHCO3(pH 8.5、0.05 mol·L-1)浸提法测定;有机质(organic matter, OM)采用重铬酸钾容量法检测;全氮(total N, TN)采用凯氏定氮仪检测;全磷(total K, TP)采用钼锑抗比色法检测;全钾(total K, TK)采用火焰光度法检测。
1.3.2 土壤HT-qPCR分析
准确称取土壤鲜样0.5 g,采用FastDNA®SPIN Kit for soil提取试剂盒(MP Biomedicals, Santa Ana, CA, USA),根据生产商说明书的步骤提取土壤基因组DNA,通过琼脂糖凝胶电泳,鉴定条带的完整性,用微量紫外分光光度计NanoDrop 2000(Thermo Fisher Scientific, Waltham, MA, USA)测定DNA的浓度。
参考ARGs相关基因的384对引物[31],采用SmartChip Real-time PCR(Warfergen Inc., USA)高通量荧光定量反应平台对不同处理下褐土ARGs相关基因进行定量检测[6],其中包括319个已知的ARGs、57个MGEs及包括16S rRNA基因在内的8个分类学基因(分类学基因与ARGs的存在及移动密切相关),基本涵盖了目前常见的与ARGs相关的基因类型。方法采用染料法,反应体系为100 nL,包括LightCycler 480 SYBR Green Ⅰ Master Mix 1×,牛血清白蛋白1 mg·mL-1,上下游引物各0.5 μmol·L-1,DNA模板5 ng·μL-1。反应过程为:95 ℃预变性10 min;在95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸15 s。所有样本均设3个技术重复,每个反应均设置空白对照。使用Canco软件获得各基因在各样本中的检出情况和Ct值(扩增循环数),Ct阈值为31,Ct值>31时将认为没有扩增。同时构建16S rRNA基因的标准质粒,根据已知浓度的标准质粒的Ct值绘制标准曲线y=kx+b。将样本Ct值代入标准曲线公式,获得样本基因的绝对定量信息,统计各基因在各样本中的基因数目,具体方法见参考文献[19]。
1.4 数据统计
采用SPSS软件(version 19)检验土壤理化性质和ARGs的正态分布和方差同质性,并进行数据的单因素方差分析(ANOVA)。在P=0.05水平下,采用LSD(least significant difference)检验分析数据间的显著性差异。采用R语言的“VennDiagram”和“grid”程序包绘制不同处理ARGs相关基因的维恩图,采用“psych”“reshape”“pheatmap”进行ARGs与MGEs的相关性分析,并通过Gephi软件进行共现网络分析(co-occurrence network analysis)的可视化,采用“ggplot2”程序包进行不同样品间ARGs相关基因的主成分分析(principal component analysis, PCA),采用置换多元方差分析(PERMANOVA)[32]评估各处理间ARGs相关基因组成差异的显著性,通过CANOCO4.5软件进行ARGs与土壤理化因子的冗余分析(redundancy analysis, RDA),并分析各项因子对ARGs相关基因变异的解释度。
2 结果(Results)
2.1 不同施肥措施对土壤理化性质的影响
不同施肥措施对土壤的理化性质造成了明显的影响,由表1可知,与CK相比,随着牛粪施用量的增加,土壤pH逐渐下降,降幅为3.1%~9.6%,且不同处理间差异显著(P<0.05)。而土壤有机质、全氮、全磷、碱解氮和有效磷呈现相反的规律,与CK相比,随着牛粪施用量的增加,呈现逐渐显著增加趋势,增幅分别为20.5%~125.6%、61.4%~224.1%、66.0%~196.0%、159.6%~360.3%和442.5%~4532.5%。此外,施肥也增加了土壤有效钾的含量。
表1 不同施肥措施下土壤理化性质
Table 1 Soil physicochemical properties under different fertilization measures
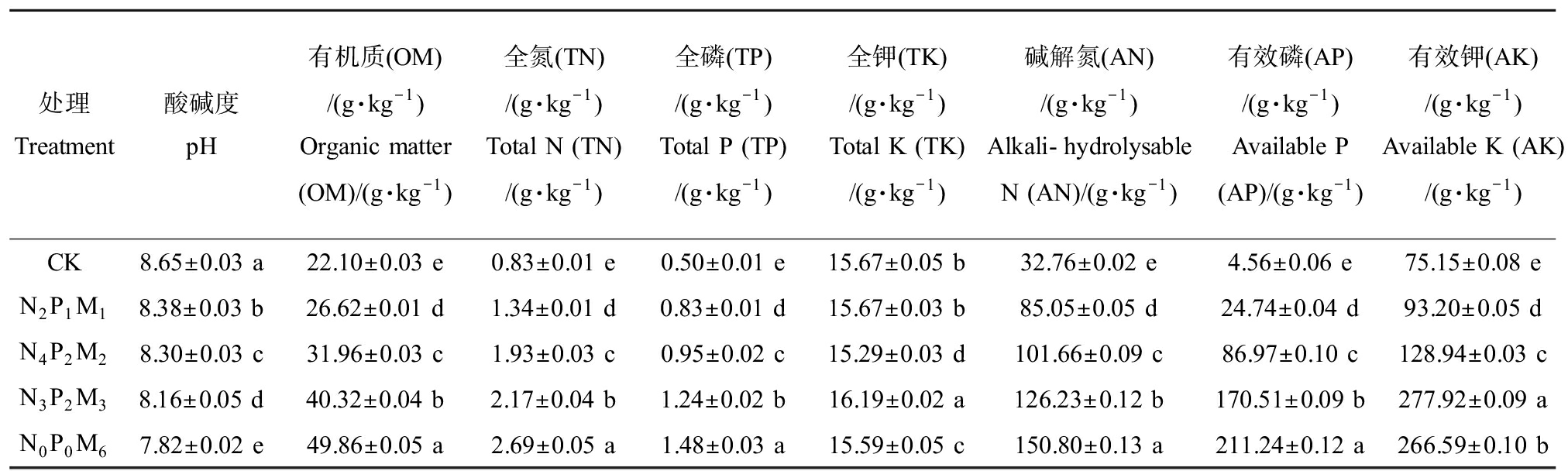
处理Treatment酸碱度pH有机质(OM)/(g·kg-1)Organic matter (OM)/(g·kg-1)全氮(TN)/(g·kg-1)Total N (TN)/(g·kg-1)全磷(TP)/(g·kg-1)Total P (TP)/(g·kg-1)全钾(TK)/(g·kg-1)Total K (TK)/(g·kg-1)碱解氮(AN)/(g·kg-1)Alkali-hydrolysable N (AN)/(g·kg-1)有效磷(AP)/(g·kg-1)Available P (AP)/(g·kg-1)有效钾(AK)/(g·kg-1)Available K (AK)/(g·kg-1)CK8.65±0.03 a22.10±0.03 e0.83±0.01 e0.50±0.01 e15.67±0.05 b32.76±0.02 e4.56±0.06 e75.15±0.08 eN2P1M18.38±0.03 b26.62±0.01 d1.34±0.01 d0.83±0.01 d15.67±0.03 b85.05±0.05 d24.74±0.04 d93.20±0.05 dN4P2M28.30±0.03 c31.96±0.03 c1.93±0.03 c0.95±0.02 c15.29±0.03 d101.66±0.09 c86.97±0.10 c128.94±0.03 cN3P2M38.16±0.05 d40.32±0.04 b2.17±0.04 b1.24±0.02 b16.19±0.02 a126.23±0.12 b170.51±0.09 b277.92±0.09 aN0P0M67.82±0.02 e49.86±0.05 a2.69±0.05 a1.48±0.03 a15.59±0.05 c150.80±0.13 a211.24±0.12 a266.59±0.10 b
注:同列数据后不同小写字母表示处理间土壤的理化性质差异达到显著水平(P<0.05)。
Note: Different lowercase letters after the same column data indicate significant difference between soil physicochemical properties of different treatments(P<0.05).
2.2 不同施肥措施对ARGs多样性的影响
5个施肥处理的土壤中,共测到186个ARGs相关基因亚型。CK、N2P1M1、N4P2M2、N3P2M3和N0P0M65个处理中检测到的ARGs相关基因亚型的个数分别为94、108、111、117和147个,可见随着牛粪施用量的增加,ARGs相关基因亚型的个数逐渐增加,即ARGs相关基因的多样性逐渐增加。维恩图(图1)展示了不同处理土样共有及特有的ARGs相关基因亚型的个数,5个处理共有的ARGs相关基因亚型有64个,CK、N2P1M1、N4P2M2、N3P2M3和N0P0M6特有的ARGs相关基因亚型个数分别为9、8、6、6和21。随着牛粪施用量的增加,特有的ARGs相关基因亚型个数呈现先减少后增加的趋势,其中,N0P0M6处理中独特ARGs相关基因亚型的个数显著高于其他的处理。
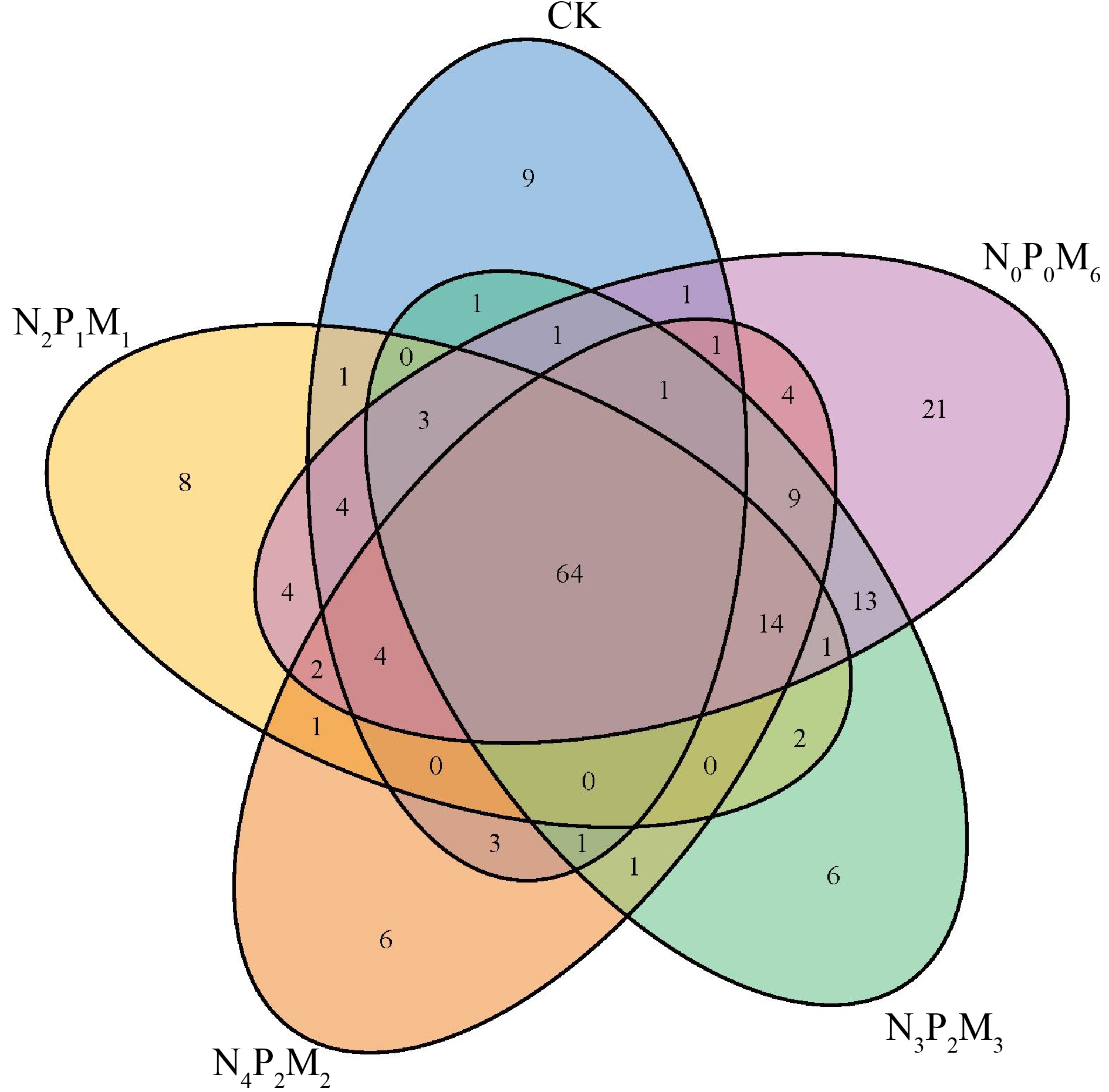
图1 不同处理间抗生素抗性基因(ARGs)相关基因维恩图
Fig. 1 Venn diagram of antibiotics resistance genes(ARGs)related genes among different treatments
2.3 不同施肥措施下ARGs相关基因主成分分析
用主成分分析(principal component analysis, PCA)对检测到的ARGs相关基因进行降维处理,以分析不同处理土样中ARGs相关基因的β-多样性。由图2可知,2个主成分共可解释40.6%的ARGs相关基因组成差异,第一轴可解释22.4%,第二轴可解释18.2%。每个处理的3个重复聚集在一起,说明平行性良好;不同处理样本间相互分离,且置换多元方差分析结果显示,R2=0.65,P=0.004,说明不同处理间ARGs相关基因的β-多样性差异显著。由图2可知,N2P1M1、N4P2M2和N3P2M33个处理的样本距离相对较近,可能是因为三者均为有机无机配施,养分含量的差异较小;而CK和N0P0M6处理的样本有偏离其他处理的倾向,这进一步说明施肥模式对土壤中ARGs相关基因影响较大。
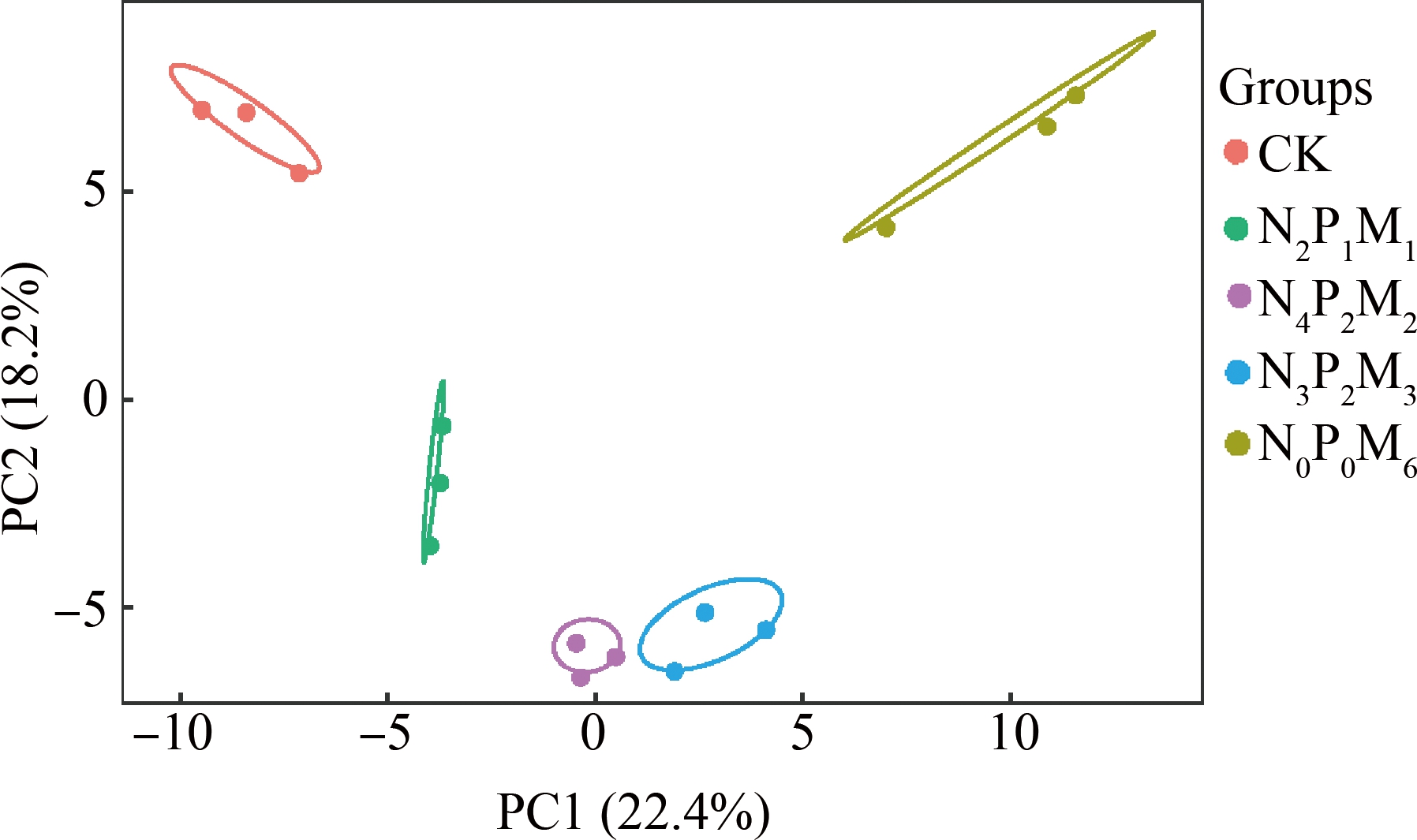
图2 不同处理间ARGs相关基因的主成分分析
Fig. 2 Principal component analysis of ARGs related genes among different treatments
2.4 不同施肥措施对ARGs相关基因绝对丰度的影响
CK、N2P1M1、N4P2M2、N3P2M3和N0P0M65个处理中除MGEs之外的ARGs的绝对丰度分别为9.03×105、7.42×105、7.16×105、6.78×105和5.89×105copies·ng-1(以单位质量的土壤DNA计);MGEs的绝对丰度分别为6.94×103、5.69×103、3.26×103、3.97×103和5.72×103copies·ng-1。可见,随着牛粪施用量的增加,除MGEs之外的ARGs的绝对丰度呈现降低趋势;而MGEs的绝对丰度呈现先降低后增加的趋势。
如图3所示,绝对丰度排名前十的ARGs相关基因占总数的98.5%~99.3%,分别为AAC(6’)-Im(46.9%~55.2%)、ANT(4’)-Ia(6.4%~19.4%)、Bacteroidetes(8.9%~20.1%)、Firmicutes(7.4%~14.3%)、aadA5(4.4%~8.2%)、AAC(3)-Via(2.4%~4.0%)、copA(0.3%~0.5%)、trb-C(0.3%~0.5%)、tetA(P)(<0.3%)和czcA(<0.3%)。其中Bacteroidetes和Firmicutes为分类学基因,trb-C为MGE,均与ARGs的存在及移动密切相关。
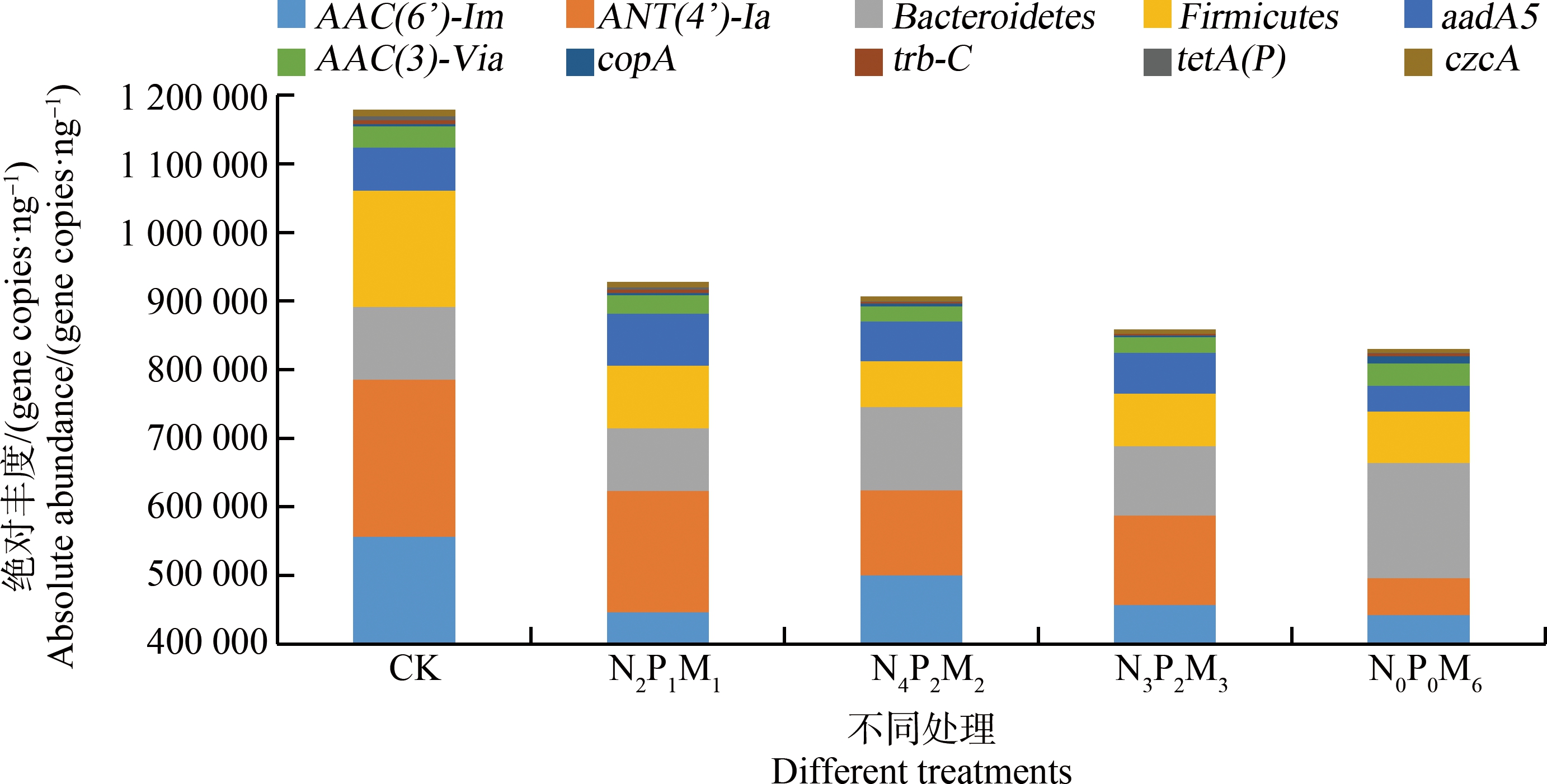
图3 不同处理ARGs相关基因亚型绝对丰度
Fig. 3 Absolute abundance of ARGs related genes subtypes in different treatments
与CK相比,施肥显著增加了AAC(6’)-Im、Bacteroidetes和copA基因的绝对丰度,增加范围分别为2.2%~17.5%、10.4%~126.1%和23.7%~384.5%,Bacteroidetes和copA基因均在N0P0M6处理中达到最高;相反,施肥显著降低了ANT(4’)-Ia、Firmicutes、tetA(P)和czcA基因的绝对丰度,降低范围分别为1.9%~67.0%、31.4%~48.7%、31.4%~95.6%和12.8%~84.1%,随着施肥量的增加,tetA(P)和czcA的含量呈现先降低后升高的趋势,且在N3P2M3处理中达到最低。
2.5 不同施肥措施下ARGs基因家族分类
图4展示了排名前十五的ARGs基因家族分类,可见,主要ARGs的类型分别为氨基糖苷类(aminoglycosides antibiotic)(68.0%~78.2%)、多药类(multidrug)(0.4%~1.5%)、四环素类(tetracycline antibiotic)(0.1%~0.5%)、氟喹诺酮类(fluoroquinolone antibiotic)(0.1%~0.3%)、大环内酯-林可酞胺-链阳菌素B类(MLSB antibiotic)(0.04%~0.2%)、β-内酰胺酶类(β-lactamase)(<0.2%)、磺胺类(sulfonamide antibiotic)(<0.2%)、利胆醇类(phenicol antibiotic)(<0.2%)。主要水平遗传元件的类型为:质粒(plasmid)(0.3%~0.5%)、转座酶(transposase)(<0.05%)、插入序列(insertional)(<0.05%)、incp-1质粒(plasmid-inc1)(<0.05%)和整合酶(intergrase)(<0.05%)等。
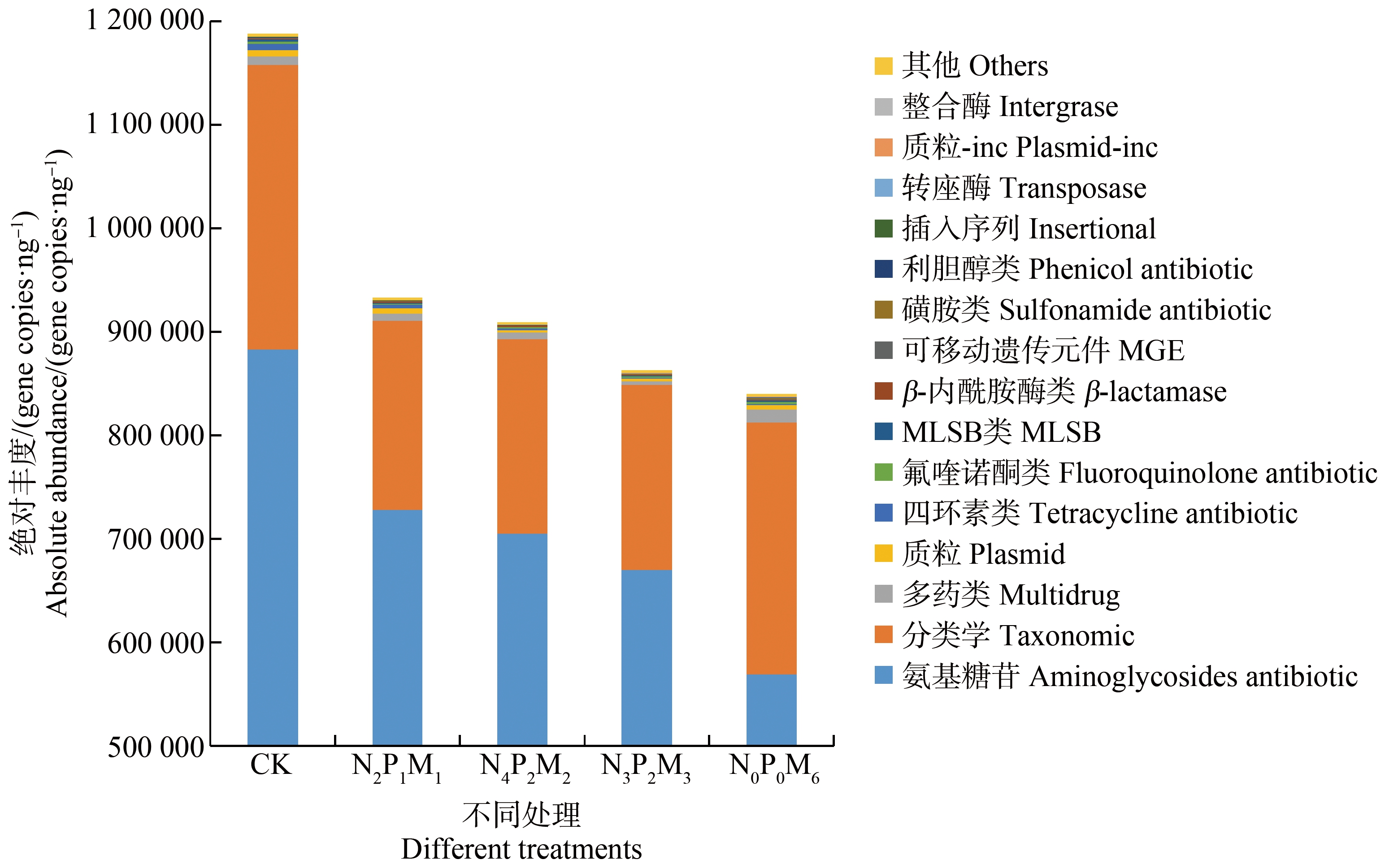
图4 不同处理主要ARGs基因家族分类
Fig. 4 Main ARGs gene family classification under different treatments
与CK相比,施用牛粪(N2P1M1、N4P2M2、N3P2M3和N0P0M6)显著降低了氨基糖苷类、四环素类、喹诺酮类ARGs基因家族的绝对丰度,降幅分别为9.9%~35.5%、18.6%~86.8%和12.6%~90.9%;与此相反,施用牛粪显著增加了磺胺类基因家族的绝对丰度,增幅为159.8%~414.6%。此外,高量施用牛粪的处理N0P0M6显著增加了多药类、MGE类、Transposase类基因家族的绝对丰度。不同处理间ARGs基因家族相似性分析结果显示,R2=0.28,P=0.023,说明不同处理间ARGs基因家族的差异显著。
2.6 ARGs与MGEs的共现网络分析
基于皮尔逊相关性分析结果,对相关系数(r)>0.6且P<0.05的ARGs和MGEs进行共现网络分析(co-occurrence network analysis),由图5可知,ARGs与MGEs共现网络包含52个节点,305条边,可以分为5个主要模块。其中的10个MGEs(IS1133、IS3、IS21-ISAs29、IncQ_oriT、tnpA-4、tnpA-6、tnpA-7、IncN_rep、Tp614和TraN)分布于模块Ⅰ、Ⅱ、Ⅳ和Ⅴ中,与不同种类的ARGs相互连接。转座子基因tnpA-7、tnpA-6是模块Ⅰ的关键网络枢纽,与不同亚型的ARGs紧密相连,进一步说明了ARGs与MGEs的相关性。

图5 ARGs与可移动遗传元件(MGEs)的共现网络分析
Fig. 5 Co-occurrence network analysis of ARGs and mobile genetic elements(MGEs)
2.7 ARGs相关基因与环境理化因子的冗余分析
通过CANOCO软件对各处理下排名前十的ARGs相关基因与土壤理化性质进行RDA分析,结果如图6所示。前2个轴总共解释了41.7%的ARGs相关基因组成变化,第一轴为36.5%,第二轴为5.2%。其中,土壤pH(F=4.579,P=0.018)对ARGs相关基因变化的贡献率最大,解释了26.0%的群落变化;其次为TN(F=4.536,P=0.018),解释了11.5%的ARGs相关基因变化,8项土壤理化因子对ARGs相关基因变化的贡献率顺序为:pH>TN>OM>TP>AP>AN>AK>TK。由图6可知,pH与aadA5、ANT(4’)-Ia、Firmicutes、tetA(P)、czcA、trb-C、AAC(6’)-Im、AAC(3)-Via的夹角均<90°,说明它们之间呈正相关;与Bacteroidetes和copA的夹角>90°,说明它们之间呈负相关。而AK、AP、TP、AN、TN和OM与Bacteroidetes和copA之间呈正相关。TK与aadA5和ANT(4’)-Ia呈正相关,与其他ARGs相关基因均呈负相关。
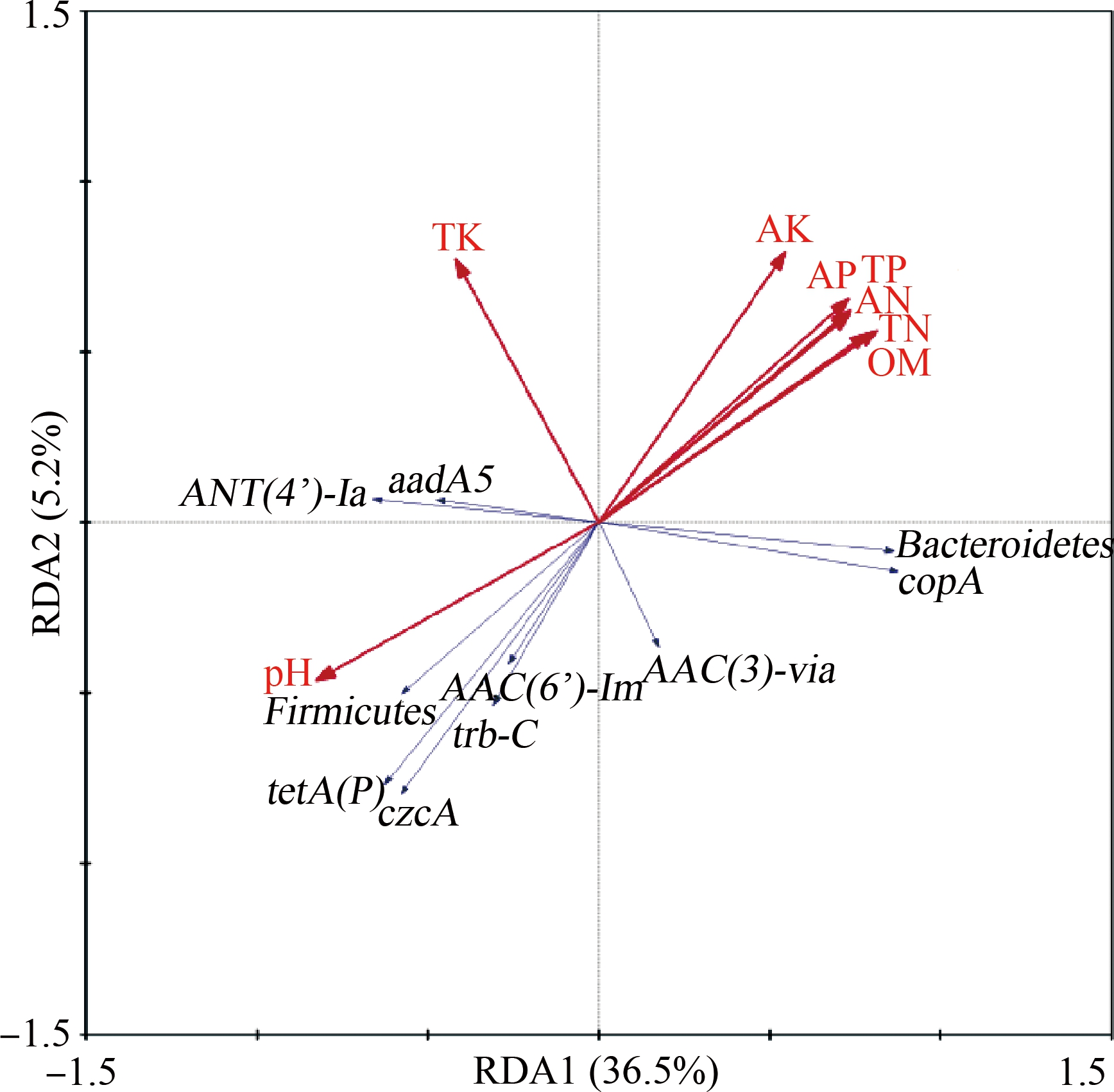
图6 ARGs相关基因与环境因子的冗余分析(RDA)
Fig. 6 Redundancy analysis(RDA)of ARGs-related genes and environmental factors
3 讨论(Discussion)
3.1 不同施肥措施对ARGs相关基因亚型及丰度的影响
Zhang等[33]研究发现施用鸡粪、牛粪和猪粪均显著提高了土壤中ARGs的多样性和丰度。Han等[24]在进行微宇宙试验中发现,施用有机肥可以引入新的ARGs,在整个培养过程中,施用有机肥的土壤中ARGs的数量均高于对照处理,在施肥4个月后,施用有机肥的土壤中ARGs的丰度高于未施有机肥的土壤,这说明有机肥是ARGs的一个主要来源。在本研究中,随着牛粪施用量的增加,ARGs相关基因亚型的数量逐渐增加,即ARGs相关基因的多样性逐渐增加。在长期不施肥的土壤中也检测到了大量的ARGs相关基因,可能是本底值和ARGs扩散传播的共同结果。土壤中的ARGs除了能通过质粒、转座子和整合子等可移动遗传元件在不同微生物之间转移,还能垂直传播,即携带ARGs的细菌在增殖过程中将ARGs传递给子代细菌,从而使得ARGs在环境中传播和扩散[34]。研究表明,高浓度的ARGs还可以形成生物气溶胶,对空气造成污染,红霉素和四环素耐药菌动力粒径研究结果显示,2种耐药菌可能主要沉降在人体的咽喉部[35]。如果ARGs转移到人类致病菌中,会形成“超级细菌”,2010年印度新德里报道的“超级细菌”对近30种抗生素产生耐药性[36],这给依赖抗生素治疗的疾病用药选择造成了极大的困难。近10年,新抗生素多黏菌素成为治疗多重耐药菌的极少数药物之一,但在2015年,中国科学家首次从人和猪体内发现了多黏菌素耐药性基因mcr-1,其一旦在全球开始传播和蔓延,人类将进入后抗生素时代,可能会出现无药可医的困境,最终会威胁人类的健康[37]。
本研究的5个处理中检测到的ARGs种类主要有氨基糖苷类、多药类、四环素类、氟喹诺酮类和MLSB类。施用牛粪(N2P1M1、N4P2M2、N3P2M3和N0P0M6)显著增加了氨基糖苷类和β-内酰胺酶类抗性基因的绝对丰度,并且高量施用牛粪的处理N0P0M6显著增加了质粒类、四环素类、MLSB类、利胆醇类、插入序列及IncP-1质粒的绝对丰度。在不同研究中检测到的ARGs类型大体一致,只是比例不尽相同。例如,Zhang等[33]在发现澳大利亚蔬菜农场土壤中主要的ARGs类型为氨基糖苷类、β-内酰胺类、FCA类、MLSB类及多药类;而Li等[19]在东北黑土农田中检测到的ARGs类型主要是MLSB类、多药类、四环素类、氨基糖苷类和β-内酰胺类等,这个差异主要取决于畜禽类型及养殖中饲喂的抗生素种类及数量,通常,猪粪和鸡粪中的ARGs比牛粪中更丰富[28],对土壤抗性造成了更大的选择压力[33]。
3.2 ARGs相关基因差异的环境驱动因子
ARGs的差异驱动因子包括微生物群落、土壤理化因子、气候变化、MGEs和植物群落等[38-39]。Hu等[40]研究发现土壤ARGs的分布主要受地上植物和地下微生物的影响;Chen等[6]研究发现,土壤微生物群落对ARGs的影响最大(解释率为39.8%),其次是环境因素(解释率为8.2%)和MGEs(解释率为4.1%);在本研究中8项理化因子对ARGs变异的解释度占41.7%。在土壤理化因子中,Li等[19]研究东北黑土农田时发现,OM与ARGs呈显著负相关。左金龙等[16]研究北京大兴不同施肥处理蔬菜地时发现pH与不施肥和单施化肥土壤中的ARGs呈显著正相关;何燕等[11]研究四川稻田土壤时发现,TN与ARGs呈显著正相关。本研究中,对不同处理ARGs差异影响最大的环境因子是pH、TN和OM,其中,pH与绝对丰度排名前十的ARGs相关基因呈显著正相关,可能是因为ARGs主要存在于土壤微生物中,而微生物的生长需要合适的pH。TN与OM是土壤环境中的核心要素,通常成显著正相关,为土壤微生物的生长提供基本的碳源和氮源,因此对ARGs差异贡献率也相对较大。
[1] Lucien M A B, Canarie M F, Kilgore P E, et al.Antibiotics and antimicrobial resistance in the COVID-19 era: Perspective from resource-limited settings[J].International Journal of Infectious Diseases, 2021, 104: 250-254
[2] Zhou X Q, Cuasquer G J P, Li Z F, et al.Occurrence of typical antibiotics, representative antibiotic-resistant bacteria, and genes in fresh and stored source-separated human urine[J].Environment International, 2021, 146: 106280
[3] 周明丽.畜牧业中滥用抗生素的现状及应对措施[J].畜禽业, 2013(8): 20-22
Zhou M L.Current situation and countermeasures of abuse of antibiotics in animal husbandry[J].Livestock and Poultry Industry, 2013(8): 20-22(in Chinese)
[4] Chee-Sanford J C, Mackie R I, Koike S, et al.Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J].Journal of Environmental Quality, 2009, 38(3): 1086-1108
[5] 刘春, 刘晨阳, 王济民, 等.我国畜禽粪便资源化利用现状与对策建议[J].中国农业资源与区划, 2021, 42(2): 35-43
Liu C, Liu C Y, Wang J M, et al.The current situation of resource utilization of livestock and poultry manure in China and the countermeasures and suggestions[J].Chinese Journal of Agricultural Resources and Regional Planning, 2021, 42(2): 35-43(in Chinese)
[6] Chen Q L, An X L, Li H, et al.Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J].Environment International, 2016, 92-93: 1-10
[7] Udikovic-Kolic N, Wichmann F, Broderick N A, et al.Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(42): 15202-15207
[8] 邹威, 金彩霞, 魏闪, 等.华北地区不同规模畜禽养殖场粪便中抗生素抗性基因污染特征[J].农业环境科学学报, 2020, 39(11): 2640-2652
Zou W, Jin C X, Wei S, et al.Occurrence of antibiotic resistance genes in livestock farms of different scales in North China[J].Journal of Agro-Environment Science, 2020, 39(11): 2640-2652(in Chinese)
[9] Han W X, Wang F H, Bai Z H, et al.Vertical distribution of antibiotic resistance genes and bacterial communities in soil of livestock manure stacking site[J].Chinese Journal of Eco-Agriculture, 2022, 30(2): 268-275
[10] 苑学霞, 梁京芸, 范丽霞, 等.粪肥施用土壤抗生素抗性基因来源、转移及影响因素[J].土壤学报, 2020, 57(1): 36-47
Yuan X X, Liang J Y, Fan L X, et al.Effects of manure application on source and transport of antibiotic resistant genes in soil and their affecting factors[J].Acta Pedologica Sinica, 2020, 57(1): 36-47(in Chinese)
[11] 何燕, 朱冬, 王东, 等.四川省稻田土壤的抗生素抗性基因多样性研究[J].农业环境科学学报, 2020, 39(6): 1249-1258
He Y, Zhu D, Wang D, et al.Diversity of antibiotic resistance genes in paddy soils in Sichuan Province, China[J].Journal of Agro-Environment Science, 2020, 39(6): 1249-1258(in Chinese)
[12] 吴楠, 乔敏.土壤环境中四环素类抗生素残留及抗性基因污染的研究进展[J].生态毒理学报, 2010, 5(5): 618-627
Wu N, Qiao M.Tetracycline residues and tetracycline resistance gene pollution in soil: A review[J].Asian Journal of Ecotoxicology, 2010, 5(5): 618-627(in Chinese)
[13] 董玉芳.城市农贸市场空气微生物群落特征及抗生素抗性基因污染[D].哈尔滨: 哈尔滨工业大学, 2014: 49-54
Dong Y F.Microbial community characteristics and antibiotic resistance gene in urban wet market air[D].Harbin: Harbin Institute of Technology, 2014: 49-54(in Chinese)
[14] 王婷, 李芬芬, 冯萃敏, 等.抗生素抗性基因在饮用水系统中的污染特征与去除研究进展[J].应用化工, 2020, 49(12): 3137-3142, 3149
Wang T, Li F F, Feng C M, et al.Pollution characteristics and removal of antibiotic resistant genes in drinking water system[J].Applied Chemical Industry, 2020, 49(12): 3137-3142, 3149(in Chinese)
[15] 王兰君.施用粪肥设施菜地土壤中抗生素抗性基因赋存特征和扩散机制[D].泰安: 山东农业大学, 2021: 35-37
Wang L J.Occurrence characteristics and diffusion mechanism of antibiotic resistance genes in greenhouse vegetable soil with manure application[D].Taian: Shandong Agricultural University, 2021: 35-37(in Chinese)
[16] 左金龙, 孙宇琪, 郭雅杰, 等.不同施肥处理对蔬菜土壤中抗生素抗性基因多样性与丰度的影响[J].环境污染与防治, 2021, 43(5): 553-556, 561
Zuo J L, Sun Y Q, Guo Y J, et al.Effects of different fertilization treatments on the diversity and abundance of antibiotic resistance genes in vegetable soil[J].Environmental Pollution &Control, 2021, 43(5): 553-556, 561(in Chinese)
[17] Thomas C M, Nielsen K M.Mechanisms of, and barriers to, horizontal gene transfer between bacteria[J].Nature Reviews Microbiology, 2005, 3(9): 711-721
[18] Xie W Y, Shen Q, Zhao F.Antibiotics and antibiotic resistance from animal manures to soil: A review[J].European Journal of Soil Science, 2018, 69: 181-195
[19] Li S, Yao Q, Liu J J, et al.Profiles of antibiotic resistome with animal manure application in black soils of northeast China[J].Journal of Hazardous Materials, 2020, 384: 121216
[20] Hill K E, Top E M.Gene transfer in soil systems using microcosms[J].FEMS Microbiology Ecology, 1998, 25(4): 319-329
[21] Ansari M I, Grohmann E, Malik A.Conjugative plasmids in multi-resistant bacterial isolates from Indian soil[J].Journal of Applied Microbiology, 2008, 104(6): 1774-1781
[22] 苏建强, 黄福义, 朱永官.环境抗生素抗性基因研究进展[J].生物多样性, 2013, 21(4): 481-487
Su J Q, Huang F Y, Zhu Y G.Antibiotic resistance genes in the environment[J].Biodiversity Science, 2013, 21(4): 481-487(in Chinese)
[23] Zhu Y G, Johnson T A, Su J Q, et al.Diverse and abundant antibiotic resistance genes in Chinese swine farms[J].Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440
[24] Han X M, Hu H W, Chen Q L, et al.Antibiotic resistance genes and associated bacterial communities in agricultural soils amended with different sources of animal manures[J].Soil Biology and Biochemistry, 2018, 126: 91-102
[25] Pu C J, Liu H, Ding G C, et al.Impact of direct application of biogas slurry and residue in fields: in situanalysis of antibiotic resistance genes from pig manure to fields[J].Journal of Hazardous Materials, 2018, 344: 441-449
[26] QianX, Gu J, Sun W, et al.Diversity, abundance, and persistence of antibiotic resistance genes in various types of animal manure following industrial composting[J].Journal of Hazardous Materials, 2018, 344: 716-722
[27] 张俊华, 陈睿华, 刘吉利, 等.宁夏养牛场粪污和周边土壤中抗生素及抗生素抗性基因分布特征[J].环境科学, 2021, 42(6): 2981-2991
Zhang J H, Chen R H, Liu J L, et al.Distribution characteristics of antibiotics and antibiotic resistance genes in manure and surrounding soil of cattle farms in Ningxia[J].Environmental Science, 2021, 42(6): 2981-2991(in Chinese)
[28] 李金阳.施用牛粪/堆肥对农田土壤和生菜中抗生素抗性基因传播的影响[D].济南: 济南大学, 2021: 16-19
Li J Y.Effects of application of cow manure/compost on the transmission of antibiotic resistance genes in farmland soil and lettuce[D].Jinan: Jinan University, 2021: 16-19(in Chinese)
[29] 刘志平, 周怀平, 解文艳, 等.长期氮磷配施对褐土细菌多样性及土壤酶活性的影响[J].干旱地区农业研究, 2022, 40(2): 163-171
Liu Z P, Zhou H P, Xie W Y, et al.Effects of long-term combined application of nitrogen and phosphorus on bacterial diversity and soil enzyme activities in cinnamon soil[J].Agricultural Research in the Arid Areas, 2022, 40(2): 163-171(in Chinese)
[30] 鲍士旦.土壤农化分析[M].3版.北京: 中国农业出版社, 2000: 90-129
[31] Stedtfeld R D, Guo X P, Stedtfeld T M, et al.Primer set 2.0 for highly parallel qPCR array targeting antibiotic resistance genes and mobile genetic elements[J].FEMS Microbiology Ecology, 2018, 94(9): 9
[32] Ramette A.Multivariate analyses in microbial ecology[J].FEMS Microbiology Ecology, 2007, 62(2): 142-160
[33] Zhang Y J, Hu H W, Gou M, et al.Temporal succession of soil antibiotic resistance genes following application of swine, cattle and poultry manures spiked with or without antibiotics[J].Environmental Pollution, 2017, 231: 1621-1632
[34] 谭璐.人类活动促进环境中抗生素抗性基因增殖扩散研究[D].天津: 天津大学, 2017: 5-6
Tan L.The promotion of anthropogenic activities on the propagation and dissemination of antibiotic resistance genes in environment[D].Tianjin: Tianjin University, 2017: 5-6(in Chinese)
[35] 陈默, 高敏, 宋渊, 等.堆肥过程逸散抗生素耐药菌气溶胶的浓度及粒径分布特征[J].农业环境科学学报, 2019, 38(7): 1633-1640
Chen M, Gao M, Song Y, et al.Characteristics of antibiotic-resistant bioaerosols during composting[J].Journal of Agro-Environment Science, 2019, 38(7): 1633-1640(in Chinese)
[36] Chihara S, Okuzumi K, Yamamoto Y, et al.First case of New Delhi metallo-β-lactamase 1-producing Escherichia coliinfection in Japan[J].Clinical Infectious Diseases, 2011, 52(1): 153-154
[37] Liu Y Y, Wang Y, Walsh T R, et al.Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study[J].The Lancet Infectious Diseases, 2016, 16(2): 161-168
[38] Wan K, Zhang M L, Ye C S, et al.Organic carbon: An overlooked factor that determines the antibiotic resistome in drinking water sand filter biofilm[J].Environment International, 2019, 125: 117-124
[39] Yang Y Y, Liu G H, Ye C, et al.Bacterial community and climate change implication affected the diversity and abundance of antibiotic resistance genes in wetlands on the Qinghai-Tibetan Plateau[J].Journal of Hazardous Materials, 2019, 361: 283-293
[40] Hu H W, Wang J T, Singh B K, et al.Diversity of herbaceous plants and bacterial communities regulates soil resistome across forest biomes[J].Environmental Microbiology, 2018, 20(9): 3186-3200