污水处理厂(wastewater treatment plants, WWTPs)被认为是抗生素抗性基因(antibiotic resistance genes, ARGs)的重要储存库,许多研究对WWTPs样品中的ARGs多样性和丰度进行了调查[1],然而以往研究主要关注的是汇集了居民生活、医疗卫生、畜禽养殖和部分工业生产等各方面污水的城镇污水处理厂;但是汇集各类工业废水的工业园区废水处理系统尚未被深入研究。工业园区特别是化学工业园区的废水通常含有大量的异源生物质,如抗生素、重金属、表面活性剂和消毒剂等,这可能为抗生素耐药性(antimicrobial resistance, AMR)的传播提供了“最佳条件”[2]。有研究表明,城市绿地[3]和工业园区土壤[4]中ARGs的发生和分布与工业活动密切相关,一些制药厂废水可能在很大程度上促进了水环境中ARGs的传播扩散[5-7]。因此,有必要研究工业园区废水的耐药基因组特征,并评估其对环境AMR发展的相对贡献。
质粒、转座子和整合子等可移动遗传元件(mobile genetic elements, MGEs)是ARGs在WWTPs中广泛分布和传播的重要驱动因素之一[8]。以往研究多基于PCR检测和相关性分析来探索ARGs与MGEs的相互关系,但是无法揭示二者在遗传上的直接关联,很难有效识别不同ARGs的水平转移机制。近年来,宏基因组学技术快速发展,为全面解析耐药基因组提供了有力支撑,通过将短序列进行组装的方法,可以获得ARGs的遗传位置信息,进而分析ARGs侧翼序列的基因组成,识别其与MGEs的共存模式。此外,众多杀菌剂和金属抗性基因(biocide and metal resistance genes, BMGs)与ARGs在遗传上存在着频繁的共现关系,反映了废水环境中多种化学污染物对AMR的共选择潜力[9]。
本研究基于宏基因组学方法,分析了城镇生活污水处理系统(W1-SD)、工业园区废水处理系统(W1-SI)以及2个城镇综合污水处理系统(W2-LH1和W2-LH2)中ARGs的组成特征,识别了不同类别ARGs的遗传位置(质粒和染色体),探究了MGEs、BMGs等基因与ARGs的相互关系,并进一步评估不同类型废水中ARGs的潜在传播风险。
1 材料与方法(Materials and methods)
1.1 污水处理系统与采样点设计
本研究选择浙江省2个典型的WWTPs(W1和W2)作为研究对象。W1位于杭州湾南岸,接纳和处理来自某城区、乡镇的生活污水,以及某精细化工园区的工业废水。该污水厂的总处理规模为22.5万m3·d-1,由2条规模为11.25万m3·d-1的污水处理系统构成,分别处理城镇生活污水(W1-SD)和化工园区废水(W1-SI)。W1-SD采用以厌氧-缺氧-好氧单元(anaerobic-anoxic-oxic tank, A2O)为核心的工艺路线,W1-SI在A2O工艺基础上增加芬顿(Fenton)和活性焦吸附工艺进行深度处理(图1)。化工园区内所有工业企业排放的废水均经由管网汇入W1-SI,其中染料、医药和农药等化工企业的废水占比最大,这些废水具有成分复杂、有毒有害污染物浓度高、可生化性差等特点,处理难度大。目前生活出水执行GB 18918一级A标准,工业出水执行GB 8978一级标准,其中化学需氧量(COD)排放标准为≤80 mg·L-1。
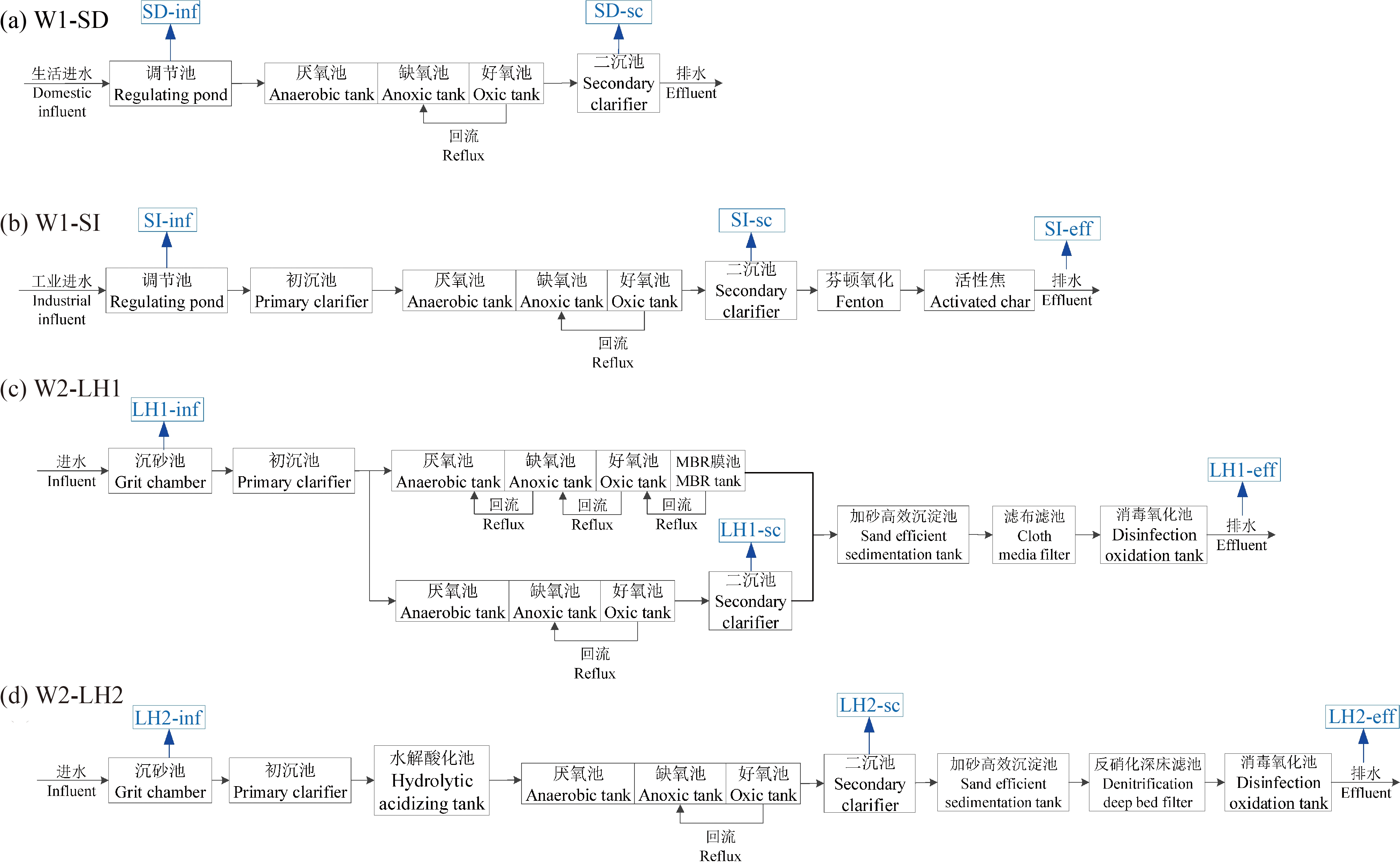
图1 各个污水处理系统工艺流程与采样点
Fig. 1 Treatment process flow and sampling sites of each wastewater treatment system
W2位于杭州湾北岸,接纳和处理来自某城市的城镇综合污水,生活污水和工业废水的比例大致相等,其中工业废水包括了石油化工、纺织印染和机械加工等废水类型。该污水厂的总处理规模为60万m3·d-1,包括一期(W2-LH1)和二期(W2-LH2)工程,处理规模均为30万m3·d-1。W2-LH1在生化处理阶段分为2条线(各处理15万m3·d-1),其中一条线增加膜生物反应器(membrane bio-reactor, MBR)工艺,后续又合并进行高效沉砂、消毒氧化等深度处理;W2-LH2采用A2O加深度处理的三级处理工艺。目前两期工程的出水水质均执行GB 18918一级A标准。
如图1所示,本研究在4个独立运行的污水处理系统(W1-SD, W1-SI, W2-LH1, W2-LH2)采集进水(influent, INF)、二次沉淀池出水(secondary clarifier, SC)和最终出水(effluent, EFF),W1-SD的二次沉淀池出水即为最终出水。以上污水处理系统均通过排海泵房将最终出水排入杭州湾。
1.2 样品采集、预处理与水质测定
污水处理系统的采样工作于2020年10月进行,工艺均在设计负荷下正常运行,并且无暴雨等突发事件。使用有机玻璃采水器采集不同污水处理系统进水(500 mL×3)、二次沉淀池出水(1 000 mL×3)和最终出水样品(1 500 mL×3),保存于无菌聚乙烯瓶中,在0~4 ℃条件下运回实验室,并在24 h之内采用0.22 μm混合纤维素酯滤膜(Millipore,德国)过滤截留水样中的细菌细胞,每张滤膜分别过滤适当体积的INF(50~100 mL)、SC(200~600 mL)和EFF(400~1 000 mL)样品,每个样点获得3~4张滤膜后存放到-20 ℃冰箱直至处理。同时,根据《水和废水监测分析方法(第四版)》测定污水的基本水质指标,包括pH、氨氮(NH3-N)、总氮(TN)、总磷(TP)和COD。
1.3 DNA提取与宏基因组测序
将0.22 μm滤膜剪碎为2~3 mm尺寸的方块,转移到DNA提取试剂盒的DNA提取管中。之后,采用PowerSoil DNA Isolation Kit(Mo Bio,美国)试剂盒进行DNA提取,具体操作参见试剂盒的操作说明书。提取完成后,采用1%琼脂糖凝胶电泳对DNA的片段长度进行检测。电泳条件为:电压100~110 V,通电时间为40 min,并使用λDNA/HindⅢ(TianGen,北京)作Marker。使用Nanodrop 2000型微量分光光度计(Thermo Fisher,美国)对DNA浓度和纯度进行检测,保证OD 260/280在1.8~2.0之间。提取纯化后的DNA保存在-20 ℃冰箱中,待后续实验。
检测合格的DNA样品加入fragmentation buffer,采用超声波细胞破碎仪进行随机打断,将打断后得到的短片段DNA用于文库构建,建好的文库需进行文库质检,对于质检合格的文库将采用Illumina HiSeq 2500高通量测序平台进行PE150测序,测序得到的原始图像数据文件,经碱基识别(base calling)分析转化为原始测序序列(raw reads),结果以FASTQ(简称为fq)文件格式存储,其中包含测序序列(reads)的序列信息以及其对应的测序质量信息。基因测序工作委托广东美格基因科技有限公司进行。原始序列上传至NCBI Sequence Read Archive(SRA)数据库(PRJNA718910)。
1.4 生物信息学分析
1.4.1 序列质控、组装和基因预测
本研究共获得186 Gb的原始数据,基于北京大学高性能计算平台,使用一系列生物信息学工具实现宏基因组测序数据的分析。
(1)序列质控和评估:首先使用FastQC(v0.11.8)[10]评估原始序列(raw reads)的整体质量,基于KneadData流程(https://github.com/biobakery/kneaddata)实现序列的质量控制,包括利用软件Trimmomactic(v0.39)[11]去除序列的双端接头序列和低质量序列(参数设置:SLIDINGWINDOW:4:20 MINLEN:50),通过软件Bowtie2(v2.3.5)[12]去除宿主污染。
(2)序列组装和评估:采用软件MEGAHIT(v1.1.3)[13]对每个样本的clean reads进行de novo拼接(参数设置:k-min 27 k-max 191 step 20),获得重叠群序列(contigs),筛选拼接长度在500 bp以上的contigs进行后续分析。序列组装的效果通过软件QUAST(v5.0.2)[14]来进行评估。
(3)基因预测和定量:采用软件Prodigal(v2.6.3)[15]对contigs进行ORFs(open reading frames)预测(参数设置:-p meta),利用软件CD-HIT(v4.8.1)[16]对获得的ORFs进行聚类(参数设置:-aS 0.9 -c 0.95),获得非冗余基因集;使用软件Salmon(v0.13.1)[17]计算出每个基因的reads count,并进一步标准化,获得TPM(transcripts per kilobase million)值,用以表征基因在各个样品中的丰度信息。
1.4.2 基于清洁序列的ARGs注释
使用ARGs-OAP(v2.0)[18]流程获得样品中ARGs的组成。该流程分为2步,第一步是使用UBLAST工具(v11.0.667)从质控后的宏基因组clean reads中预先筛选ARGs相似序列,第二步是使用BLASTX工具(v2.2.28+)按照默认的参数设置(e-value ≤ 10-7,序列相似度≥ 80%,比对长度≥25 aa)将上述相似序列与SARG(v2.2)数据库进行比对,并根据SARG的分级结构进行ARGs的注释和分类。基于CARD数据库(https://card.mcmaster.ca/home)匹配不同ARGs的抗性机制。根据下列公式[18],对宏基因组数据中ARGs的丰度进行标准化,单位为ARGs拷贝数/细胞数量(copies·cell-1),可在不同样品或不同基因类型之间进行自由比较。
ARG丰度![]()
细胞数量![]()
式中:Ni(mapped reads)是宏基因组clean reads中比对到某条特定ARG参考序列的序列条数;Lreads是Illumina高通量测序获得的序列长度(150 bp);Li(reference reads)是某条特定ARG参考序列的长度;N16S sequence是宏基因组clean reads中被鉴定为16S rRNA基因的序列条数;L16S sequence是所有16S rRNA基因序列的平均长度(1 432 bp);N16S copy number是微生物细胞中16S rRNA基因序列的平均拷贝数;n是样品中被注释到属于某个ARG类型或亚型的参考序列个数。
1.4.3 基于contigs的ARGs及其他功能基因注释
利用BLASTP工具(v2.6.0)将非冗余基因集分别与SARG数据库、MGE数据库(https://github.com/KatariinaParnanen/MobileGeneticElementDatabase)和BacMet数据库(v2.0, http://bacmet.biomedicine.gu.se/)进行比对,获得ARGs、MGEs和BMGs等基因的组成。序列比对参数为e-value ≤ 10-5,序列相似度(identity)≥ 80%,序列覆盖度(coverage)≥ 70%。
1.4.4 基于contigs的序列分型
利用软件Plasflow(v1.1)[19]按照默认参数(-c 0.7)对contigs(根据软件开发人员的建议,将序列长度<1 000 bp的contigs排除在外)进行分型,识别出染色体序列和质粒序列,并分析2类序列所携带的ARGs组成。利用软件WAAFLE(http://huttenhower.sph.harvard.edu/waafle)筛选不同宏基因组数据集中发生水平基因转移事件的contigs(>500 bp)。
1.5 统计分析和绘图
使用非度量多维标度(non-metric multidimensional scaling, NMDS)分析探索和可视化不同样本分组之间ARGs组成的相异程度,输入数据为已经计算好的两两样品间的距离测度矩阵(Bray-Curtis距离)。使用置换多元方差分析(permutational multivariate analysis of variance, PERMANOVA)确定2个矩阵之间差异的显著性。如果计算得到的P值<0.05,则表明2组数据或2个矩阵之间具有显著差异,P值采用BH(Benjamini &Hochberg)方法进行校正。使用Origin 2018b或R语言包ggplot2、pheatmap绘制堆积图、柱状图和热图。使用R语言包genoPlotR绘制不同基因在contigs上的排列结构图。通过R语言包circlize绘制Circos图展示序列类型与ARGs类型的对应关系。
2 结果与讨论(Results and discussion)
2.1 不同类型废水ARGs的结构组成和变化规律
基于ARGs-OAP流程对4个污水处理系统不同废水样品的宏基因组clean reads进行ARGs注释,共识别到477种ARGs亚型,分别编码17种抗性类型和6种抗性机制。如图2(a)和图2(c)所示,多重耐药类(multidrug, 13.4%~65.6%, 6.20×10-2~8.53×10-1 copies·cell-1)、磺胺类(sulfonamide, 2.5%~24.9%, 3.31×10-2~6.04×10-1 copies·cell-1)、氨基糖苷类(aminoglycoside, 1.6%~19.9%, 1.92×10-2~5.60×10-1 copies·cell-1)、杆菌肽类(bacitracin, 3.1%~19.0%, 3.71×10-2~2.47×10-1 copies·cell-1)、大环内酯-林可酰胺-链霉素类(MLS, 1.2%~13.1%, 1.54×10-2~3.70×10-1 copies·cell-1)、β-内酰胺类(beta-lactam, 2.0%~11.6%, 1.05×10-2~3.21×10-1 copies·cell-1)和四环素类(tetracycline, 1.3%~12.7%, 8.73×10-3~3.57×10-1 copies·cell-1)抗性基因是样品中主要的ARGs类型,这与其他研究中WWTPs的ARGs筛查结果较为一致[20]。同时,如图2(b)所示,477种ARGs亚型主要通过抗生素外排(efflux, 26.1%~59.0%)、抗生素灭活(inactivation, 5.0%~41.4%)、抗生素靶标替代(target replacement, 2.6%~25.2%)和抗生素靶标改变(target alteration, 4.5%~20.2%)等机制表达相应的耐药性,特别是绝大部分多重耐药类基因表现为抗生素外排机制,在各样品中均占据主导地位,这表明多药外排系统是环境微生物表达耐药性的重要途经[21]。

图2 不同废水样品中抗生素抗性基因(ARGs)组成
注:(a)抗性类型,(b)抗性机制的相对比例,(c)不同ARGs类型的相对丰度;热图(灰色方块表示未检出);MLS表示大环内酯类-林可酰胺类-链霉素类。
Fig. 2 Composition of detected antibiotic resistance genes (ARGs) in the different wastewater samples
Note: (a) The relative proportion of ARG types, (b) Antibiotic resistance mechanism, (c) Heatmap shows the relative abundance of different ARG types; gray in the heatmap means not detected; MLS means macrolide-lincosamide-streptogramin.
进一步计算了不同样品中ARGs的丰富度指数和总相对丰度,如图3所示。结果表明2个城镇综合污水处理系统的INF样品中ARGs的多样性(W2-LH1:347, W2-LH2:333)和相对丰度(W2-LH1:2.82 copies·cell-1, W2-LH2:2.14 copies·cell-1)更高,而工业园区废水处理系统(W1-SI)接收的ARGs多样性(260)和相对丰度(1.24 copies·cell-1)最低,这可能是因为工业进水中有毒有害的污染物浓度高,对微生物有持续的杀灭作用,导致微生物量和相关基因丰度的下降。污水处理过程对ARGs的多样性有一定的削减作用,从4个污水处理系统的INF到EFF,ARGs的丰富度指数均发生降低,但最终排水中依然含有较多种类的ARGs(145~270)。对于ARGs的相对丰度来说,经过二次沉淀工艺,SC样品中ARGs的总丰度均有较为明显的下降,表明通过二次沉淀池泥水分离将ARGs转移至污泥相是削减水相ARGs的重要途经[22];但是经过后续的深度处理,只有W2-LH1的EFF样品中ARGs总丰度持续下降,W1-SI和W2-LH2的EFF均相较于SC样品发生了ARGs的富集,这表明深度处理工艺可能并不能有效去除ARGs,反而可能刺激了某些ARGs的增殖扩散[23]。图2(c)结果表明,多种ARGs在SI_eff中的相对丰度要高于SI_sc,包括多重耐药类、磺胺类、氨基糖苷类和杆菌肽类等基因;而在LH2_eff中,主要是多重耐药类和杆菌肽类基因发生了显著的富集,其相对丰度甚至高于INF样品。
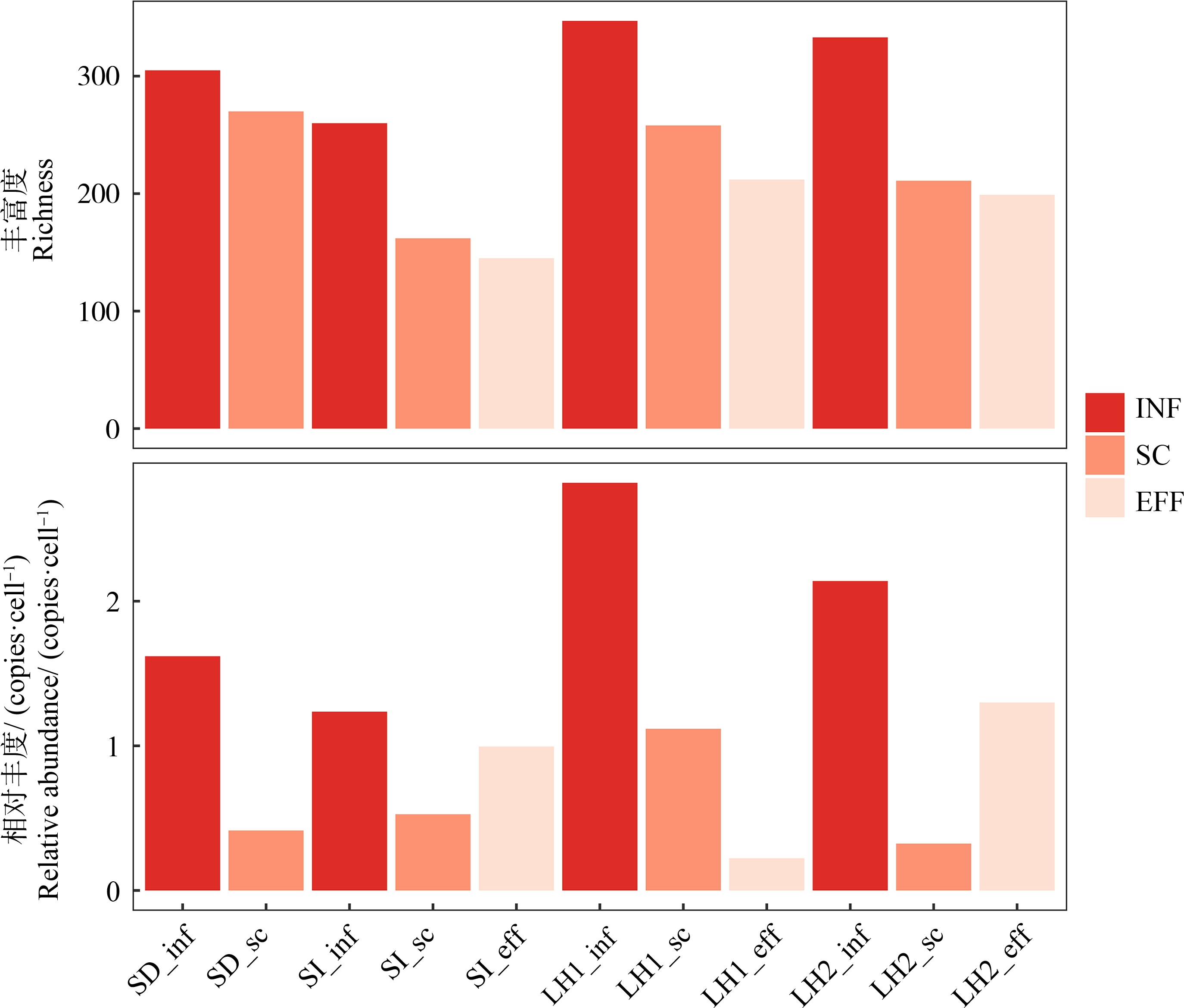
图3 不同废水样品中ARGs的多样性和总相对丰度
Fig. 3 The diversity and total relative abundance of detected ARGs in the different wastewater samples
通过基于Bray-Curtis距离的NMDS分析不同样品之间ARGs组成的差异,如图4所示。结果表明WWTPs不同处理单元废水中ARGs的组成有显著的差异,W1-SD、W2-LH1和W2-LH2的INF中ARGs的结构组成较为相似,但是经过污水处理过程,SC和EFF中ARGs结构组成的差异则较为明显,这表明不同的污水处理工艺或物理化学条件,可能对ARGs的增殖扩散施加了不同的选择效应[24]。然而,SI_inf的ARGs组成与另外3个INF样品有较大差异,且其与SI_sc和SI_eff的差异并不大,说明W1-SI中ARGs的结构组成是更稳定的。如表1所示,相比于其他3个污水处理系统的废水,W1-SI沿程废水均具有更高的COD、TN和TP,表示来自工业园区的各类废水富含大量的难降解污染物,前期的研究也发现W1-SI废水样品中抗生素和可吸附有机卤代物的浓度更高[25-26],这种持续的强选择压力可能降低了微生物携带ARGs的适应性代价,使得ARGs能够稳定地传播。
表1 废水样品水质
Table 1 The quality of wastewater samples
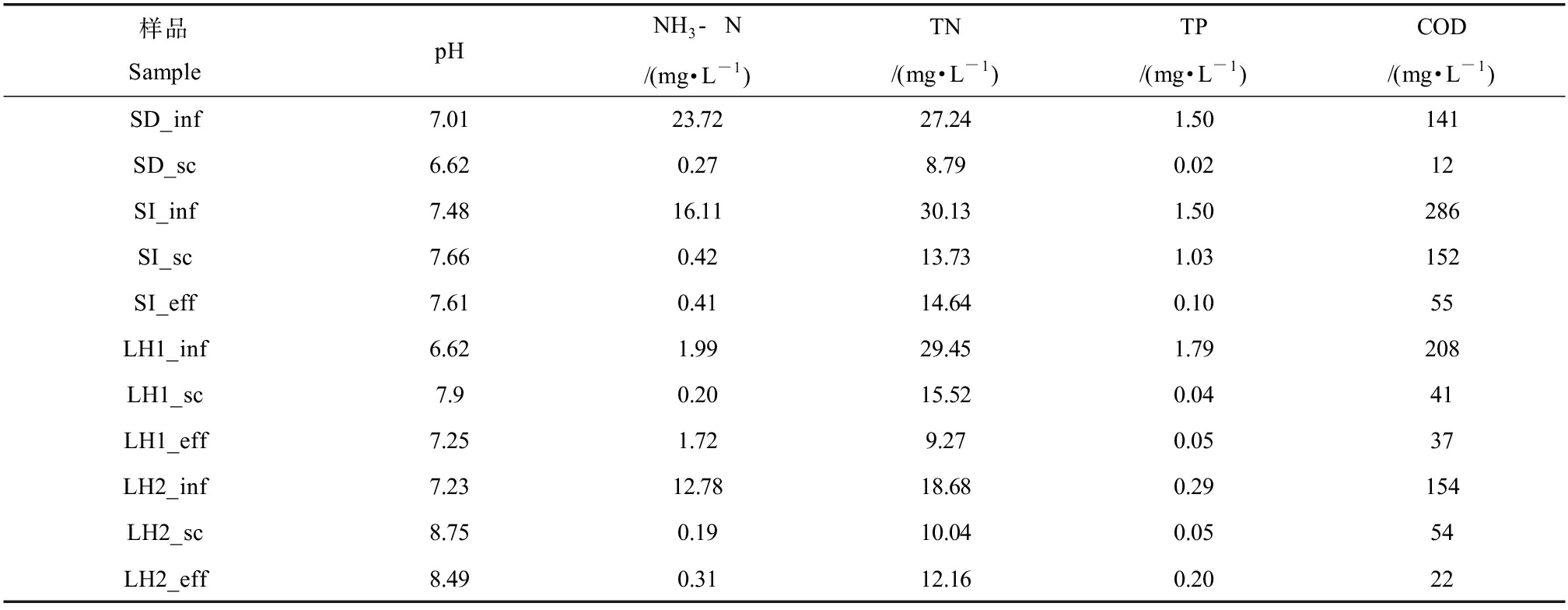
样品SamplepHNH3-N/(mg·L-1)TN/(mg·L-1)TP/(mg·L-1)COD/(mg·L-1)SD_inf7.0123.72 27.24 1.50 141SD_sc6.620.27 8.79 0.02 12SI_inf7.4816.11 30.13 1.50 286SI_sc7.660.42 13.73 1.03 152SI_eff7.610.41 14.64 0.10 55LH1_inf6.621.99 29.45 1.79 208LH1_sc7.90.20 15.52 0.04 41LH1_eff7.251.72 9.27 0.05 37LH2_inf7.2312.78 18.68 0.29 154LH2_sc8.750.19 10.04 0.05 54LH2_eff8.490.31 12.16 0.20 22
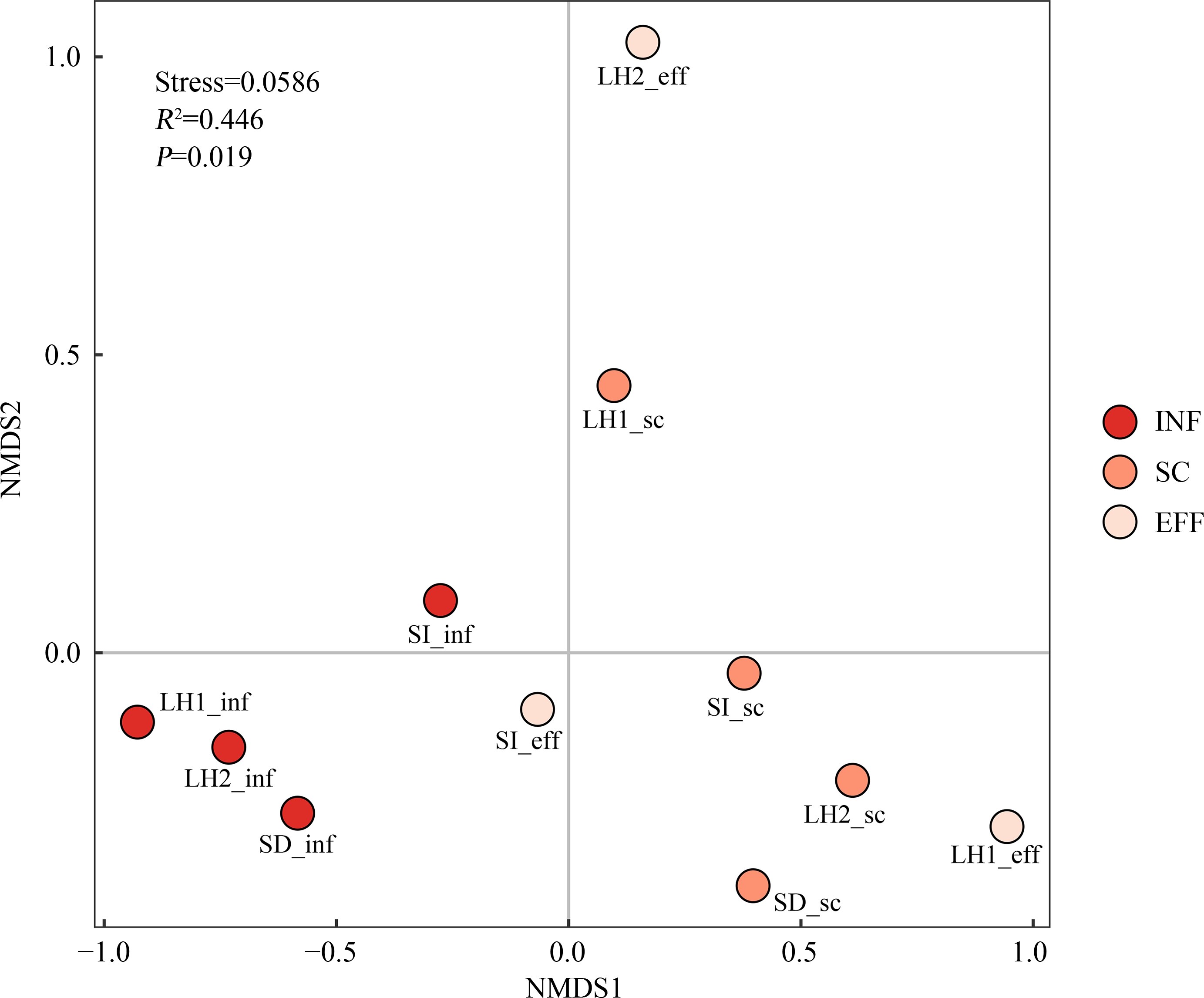
图4 不同废水样品ARGs组成的NMDS分析(基于Bray-Curtis距离)
注:PERMANOVA检验不同分组间差异的显著性。
Fig. 4 NMDS analysis of ARGs structure in the different wastewater samples based on Bray-Curtis distance
Note: PERMANOVA test the significance of differences.
2.2 ARGs与其他风险指示基因的共存模式
基于对重叠群序列(contigs)的基因注释结果,本研究进一步筛选出不同样品中同时携带ARGs、MGEs和BMGs的contigs,分析其基因排布特征。如图5所示,qacEdelta-sul1的基因组合在不同污水处理系统中最为普遍,这2个基因是Ⅰ型整合子的基本组件之一,位于整合子系统的3’保守末端(3’-CS)[27],在LH2_eff的Contig_120113中还发现intI1(Ⅰ型整合子的整合酶基因)与qacEdelta-sul1的共存关系,同时qacEdelta基因也与aadA、aac(6’)-Ⅰ、aac(6’)-Ⅱ、ereA、dfrA17、OXA-119、OXA-10和OXA-226等基因在contigs上邻近排列,这些基因是目前发现的整合子基因盒中携带的主要ARGs类型[28],这表明Ⅰ型整合子对污水处理系统中磺胺类、氨基糖苷类和β-内酰胺类ARGs的水平转移发挥了重要作用。值得注意的是,qacEdelta基因主要表达对季铵盐类化合物、吖啶和菲啶等消毒剂或杀菌剂的耐药性,因此该基因不仅被归类于MGEs,也属于ARGs中的多重耐药类别(qacEdelta1)和BMGs中的多重杀菌剂抗性类别(qacEdelta1)。tnpA、tnpA2等转座酶基因是污水处理系统样品中广泛分布的MGEs,也被发现与多种抗性基因在contigs上存在线性排列的关系,比如tnpA与tetA和多个汞抗性基因(merR, merT, merP, merA, merD, merE)在SD_inf的Contig_325513上同时排布,之前研究也发现汞抗性基因与多种ARGs在质粒上共存[9, 29],表明汞金属可能对ARGs的转移扩散发挥了重要的驱动作用。
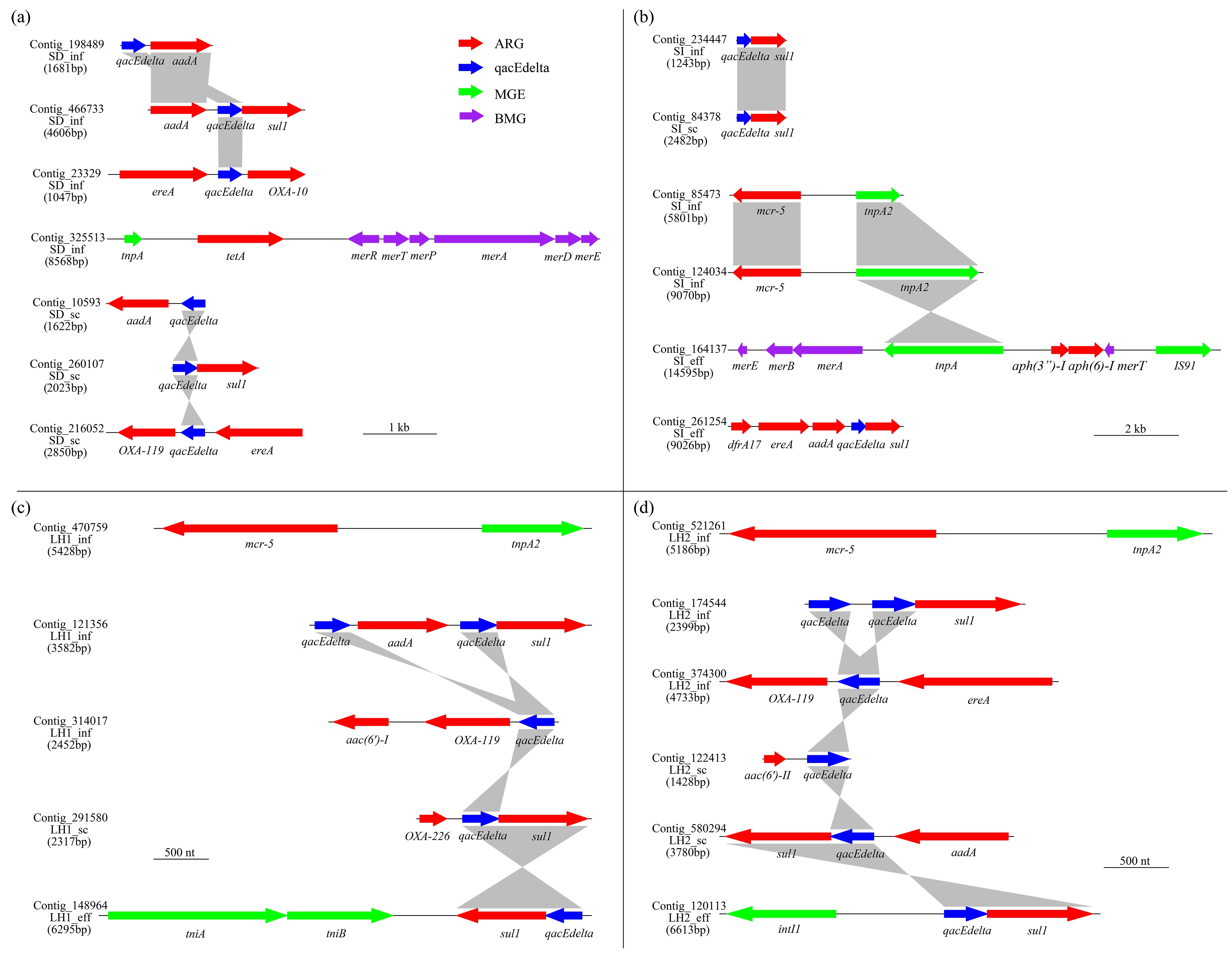
图5 不同类别风险指示基因在contigs上的排布特征
注:代表性contigs来自不同污水处理系统;(a) W1-SD;(b) W1-SI;(c) W2-LH1;(d) W2-LH2;MGE表示可移动遗传元件;BMG表示杀菌剂和金属抗性基因。
Fig. 5 The distribution characteristics of different types of risk indicator genes on the contigs
Note: Representative contigs are selected from different wastewater treatment systems; (a) W1-SD; (b) W1-SI; (c) W2-LH1; (d) W2-LH2; MGE means mobile genetic element; BMG means biocide and metal resistance gene.
此外SI_inf、LH1_inf和LH2_inf的contigs上均存在tnpA2-mcr-5的基因组合,mcr类抗性基因可以导致微生物产生对多黏菌素的耐药性,目前普遍认为多黏菌素类抗生素是临床治疗多重耐药革兰氏阴性菌感染的终极药物[30],因此tnpA2-mcr-5在污水处理系统中的共存模式可能会促进多黏菌素耐药性的发展,具有很高的健康风险。同时,本研究还在SI_eff的宏基因组序列中组装了2条比较长的contigs,分别是Contig_164137(14 595 bp)和Contig_261254(9 026 bp),其中Contig_164137携带2个氨基糖苷类ARGs(aph(3’)-I, aph(6’)-I),4个汞抗性基因(merA, merB, merE, merT)和2个MGEs(tnpA, IS91),而Contig_261254除了携带qacEdelta-sul1的基因组合之外,还有3个不同的ARGs(aadA, ereA, dfrA17),这暗示工业园区废水的处理可能会选择出携带多种抗性基因的细菌,可以对多种胁迫都有较强的适应性,若不能被现有工艺去除,将增大排水的潜在威胁。
以上结果表明,污水处理系统中Ⅰ型整合子(包括qacEdelta基因)、转座酶基因等MGEs在ARGs的增殖扩散中发挥了关键作用。而在宏基因组clean reads的注释结果中,sul1 (2.81×10-2~5.12×10-1 copies·cell-1)、aadA (5.39×10-3~3.58×10-1 copies·cell-1)和ereA (9.92×10-4~2.81×10-1 copies·cell-1)等基因亚型在废水样品中的相对丰度都比较高,占总ARGs相对丰度的比例分别为15.4%、8.7%和5.8%,因此MGEs可能是这些基因能够在废水处理过程中稳定遗传的主要原因之一;另外,污水处理系统中残留较多的重金属、杀菌剂等物质也会刺激微生物产生相应的抗性基因,进而通过共抗性、交叉抗性等机制,提高ARGs的共发生频率[31],增加多重耐药细菌出现的可能,成分复杂、污染物浓度高的工业园区废水更可能提供这样的条件,需要予以高度的关注。
2.3 不同ARGs的遗传位置分析
质粒是一类普遍存在于细菌细胞内的共价闭合环状的DNA分子,是独立的、可自我复制的遗传元件[27]。研究表明,质粒容易发生接合、转座和重组,是ARGs在微生物之间水平转移的重要载体[32],多种ARGs和BMGs等基因往往同时定位在相同质粒上,这使得多重耐药性相对更容易传播。因此分析定位于质粒上的ARGs组成和丰度,对评估ARGs的可移动性和传播风险具有重要意义[33]。
本研究基于Plasflow软件,将所有样品中携带有ARG的contigs(>1 000 bp)进行序列分型。如表2所示,共识别到211条染色体序列、289条质粒序列和213条未鉴别序列,分别占contigs总数量的29.6%、40.5%和29.9%。图6(a)展示了不同类别contigs所携带ARGs的总相对丰度大小,除了LH2_eff和LH1_sc,其他样品中编码在质粒序列上的ARGs相对丰度均高于其他2类序列,特别是在INF样品中,质粒型ARGs占据了绝对的优势(SI_inf: 156.76 TPM~LH1_inf: 542.15 TPM),说明废水样品中的大部分ARGs普遍具有较高的移动性。另外,在所有EFF样品中,SI_eff依然含有较高丰度的质粒型ARGs(127.53 TPM),仅次于4个INF样品。前述结果发现ARGs在SI_eff和LH2_eff中相较于SC样品均发生了富集现象,现进一步探明LH2_eff是富集了较高比例的染色体型ARGs(145.75 TPM),这表明工业园区废水处理系统排放的ARGs可能具有更高的传播风险。
表2 不同废水样品中被识别为染色体、质粒和未鉴别序列的contigs(>1 000 bp)数量
Table 2 The number of contigs (>1 000 bp) identified as chromosome, plasmid and unclassified sequences in different wastewater samples
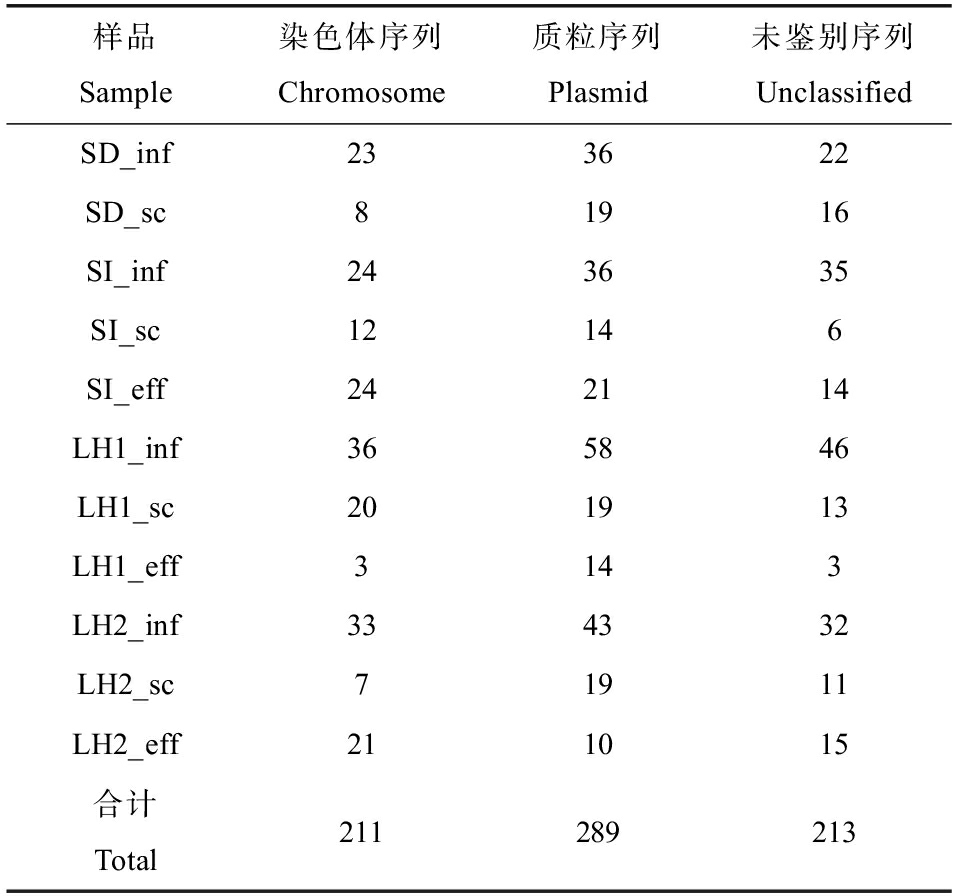
样品Sample染色体序列Chromosome质粒序列Plasmid未鉴别序列UnclassifiedSD_inf233622SD_sc81916SI_inf243635SI_sc12146SI_eff242114LH1_inf365846LH1_sc201913LH1_eff3143LH2_inf334332LH2_sc71911LH2_eff211015合计Total211289213
注:表中contigs均携带有至少一个ARG。
Note: Each contigs in the table carries at least one ARG.

图6 废水样品中ARGs在contigs(>1 000 bp)的遗传位置识别
注:(a)位于染色体、质粒和未鉴别序列的ARGs相对丰度;(b)不同类型ARGs与不同类别contigs的对应关系。
Fig. 6 Genetic location of ARG-carrying contigs (>1 000 bp) in wastewater samples
Note: (a) The relative abundance of ARGs located on plasmid, chromosomal or unclassified contigs; (b) The mapping relationship between different ARG types and different contig types.
对不同类别contigs携带的ARGs类型进行分析,如图6(b)所示,氨基糖苷类(88.8%)、四环素类(87.2%)、磺胺类(80.7%)、MLS类(68.9%)、β-内酰胺类(65.7%)和其他类(62.9%)抗性基因更多地编码在质粒序列上;而对相对丰度最高的多重耐药类抗性基因来说,44.7%属于质粒型ARGs,而43.4%属于染色体型ARGs;此外,有46.5%的杆菌肽类抗性基因编码在染色体序列上,还有50.0%则编码在未鉴别序列上。这与前述的研究结果一致,氨基糖苷类、磺胺类和β-内酰胺类等ARGs往往与多种MGEs共线性排列,因此具有较高的可移动性,其他研究也表明这几类ARGs更多地富集在质粒序列上[33]。而位于染色体序列上的杆菌肽类和多重耐药类抗性基因可能代表了微生物的固有耐药性,比如杆菌肽类主要的基因亚型bacA,被证明在环境细菌中具有较高的自然流行率[34-35];还有些多重耐药类基因属于外排/转运系统,如multidrug-transporter、multidrug-ABC-transporter等,这些功能系统不仅有助于抗生素耐药性,而且还发挥了关键的细胞生理作用,可以泵出各种代谢物[36]。
因此序列分型的方法为认识不同ARGs的移动属性提供了重要参考,一般来说,染色体型ARGs序列在遗传上比较保守,通常不易被水平转移[37],风险相对较低。但并不意味着这些基因是无风险的,因为在多重复杂的选择压力下,其中一些基因可以在病原体中动员和富集[38],一旦染色体DNA释放到环境中,其稳定性较强,也很有可能被邻近细菌重新捕获[39]。
更进一步地,本研究利用WAAFLE软件识别了不同样品contigs数据集中能够表征发生潜在水平基因转移(horizontal gene transfer, HGT)事件的contigs数量。如图7所示,SI_inf和SI_eff具有最多的发生HGT的contigs数量,分别为410个和289个,而在其他污水处理系统中,发生HGT的contigs数量均在INF样品中最高,且随废水处理过程逐级下降。这表明工业园区废水的微生物组中发生HGT的频率更高,间接推动了ARGs在微生物之间的广泛传播。
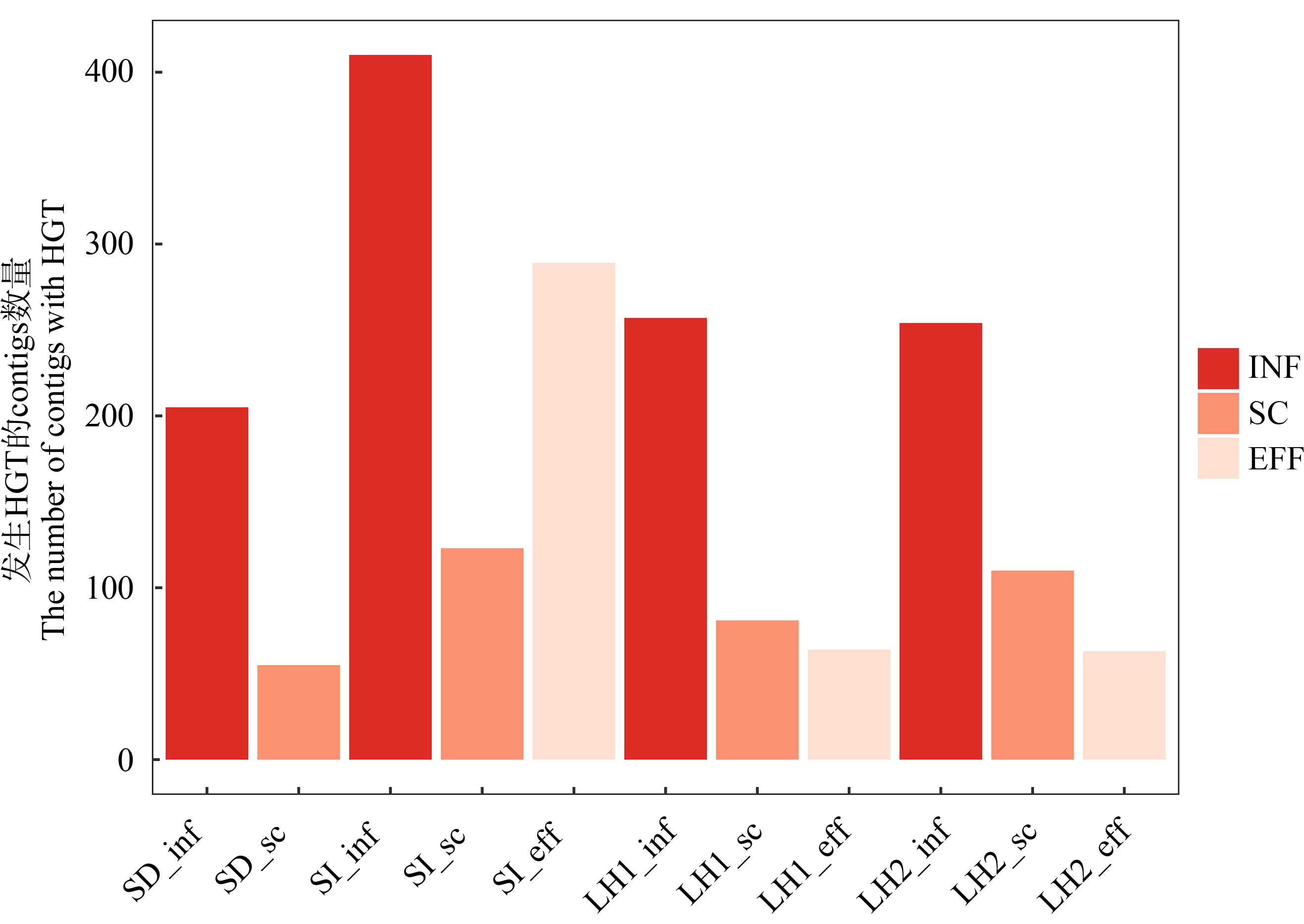
图7 基于WAAFLE识别发生水平基因转移(HGT)的contigs数量
Fig. 7 Identification of contigs with horizontal gene transfer (HGT) potential based on WAAFLE
综上所述,本研究通过宏基因组学技术解析了不同污水处理系统的耐药基因组特征和传播机制,结果表明多重耐药类、磺胺类、氨基糖苷类和杆菌肽类抗性基因是污水样品中的主要ARGs类型,qacEdelta-sul1的基因组合在不同污水处理系统中最为普遍,Ⅰ型整合子、转座酶基因等MGEs对sul1、aadA和ereA等基因亚型的增殖扩散发挥了关键作用。工业园区废水处理系统的ARGs组成与城镇污水处理系统的差异较大,经过工业园区废水处理系统的处理可能会选择出携带多种抗性基因的耐药菌,并富集了较高丰度的质粒型ARGs,表明工业园区废水处理系统排放的ARGs具有更高的传播风险。
[1] Qin K N, Wei L L, Li J J, et al. A review of ARGs in WWTPs: Sources, stressors and elimination [J]. Chinese Chemical Letters, 2020, 31(10): 2603-2613
[2] Bengtsson-Palme J, Milakovic M,  vecová H, et al. Industrial wastewater treatment plant enriches antibiotic resistance genes and alters the structure of microbial communities [J]. Water Research, 2019, 162: 437-445
vecová H, et al. Industrial wastewater treatment plant enriches antibiotic resistance genes and alters the structure of microbial communities [J]. Water Research, 2019, 162: 437-445
[3] Yan Z Z, Chen Q L, Zhang Y J, et al. Antibiotic resistance in urban green spaces mirrors the pattern of industrial distribution [J]. Environment International, 2019, 132: 105106
[4] Zheng B W, Liu W H, Xu H, et al. Occurrence and distribution of antimicrobial resistance genes in the soil of an industrial park in China: A metagenomics survey [J]. Environmental Pollution, 2021, 273: 116467
[![]() M, Vestergaard G, González-Plaza J J, et al. Effects of industrial effluents containing moderate levels of antibiotic mixtures on the abundance of antibiotic resistance genes and bacterial community composition in exposed creek sediments [J]. The Science of the Total Environment, 2020, 706: 136001
M, Vestergaard G, González-Plaza J J, et al. Effects of industrial effluents containing moderate levels of antibiotic mixtures on the abundance of antibiotic resistance genes and bacterial community composition in exposed creek sediments [J]. The Science of the Total Environment, 2020, 706: 136001
[6] Tang Y, Liang Z S, Li G Y, et al. Metagenomic profiles and health risks of pathogens and antibiotic resistance genes in various industrial wastewaters and the associated receiving surface water [J]. Chemosphere, 2021, 283: 131224
[7] Ding H J, Qiao M, Zhong J Y, et al. Characterization of antibiotic resistance genes and bacterial community in selected municipal and industrial sewage treatment plants beside Poyang Lake [J]. Water Research, 2020, 174: 115603
[8] Karkman A, Do T T, Walsh F, et al. Antibiotic-resistance genes in waste water [J]. Trends in Microbiology, 2018, 26(3): 220-228
[9] Pal C, Bengtsson-Palme J, Kristiansson E, et al. Co-occurrence of resistance genes to antibiotics, biocides and metals reveals novel insights into their co-selection potential [J]. BMC Genomics, 2015, 16: 964
[10] Chen S F, Zhou Y Q, Chen Y R, et al. Fastp: An ultra-fast all-in-one FASTQ preprocessor [J]. Bioinformatics, 2018, 34(17): i884-i890
[11] Bolger A M, Lohse M, Usadel B. Trimmomatic: A flexible trimmer for Illumina sequence data [J]. Bioinformatics, 2014, 30(15): 2114-2120
[12] Langmead B, Salzberg S L. Fast gapped-read alignment with bowtie 2 [J]. Nature Methods, 2012, 9(4): 357-359
[13] Li D H, Liu C M, Luo R B, et al. MEGAHIT: An ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph [J]. Bioinformatics, 2015, 31(10): 1674-1676
[14] Gurevich A, Saveliev V, Vyahhi N, et al. QUAST: Quality assessment tool for genome assemblies [J]. Bioinformatics, 2013, 29(8): 1072-1075
[15] Hyatt D, Chen G L, Locascio P F, et al. Prodigal: Prokaryotic gene recognition and translation initiation site identification [J]. BMC Bioinformatics, 2010, 11: 119
[16] Fu L M, Niu B F, Zhu Z W, et al. CD-HIT: Accelerated for clustering the next-generation sequencing data [J]. Bioinformatics, 2012, 28(23): 3150-3152
[17] Patro R, Duggal G, Love M I, et al. Salmon provides fast and bias-aware quantification of transcript expression [J]. Nature Methods, 2017, 14(4): 417-419
[18] Yin X L, Jiang X T, Chai B L, et al. ARGs-OAP v2.0 with an expanded SARG database and Hidden Markov Models for enhancement characterization and quantification of antibiotic resistance genes in environmental metagenomes [J]. Bioinformatics, 2018, 34(13): 2263-2270
[19] Krawczyk P S, Lipinski L, Dziembowski A. PlasFlow: Predicting plasmid sequences in metagenomic data using genome signatures [J]. Nucleic Acids Research, 2018, 46(6): e35
[20] Hendriksen R S, Munk P, Njage P, et al. Global monitoring of antimicrobial resistance based on metagenomics analyses of urban sewage [J]. Nature Communications, 2019, 10(1): 1124
[21] Du D J, Wang-Kan W K, Neuberger A, et al. Multidrug efflux pumps: Structure, function and regulation [J]. Nature Reviews Microbiology, 2018, 16(9): 523-539
[22] 洪颖忻, 吴浪, 张立秋, 等. 制药废水处理系统中抗生素抗性基因的研究进展[J]. 工业水处理, 2022, 42(4): 39-45
Hong Y X, Wu L, Zhang L Q, et al. Research progress of antibiotic resistance genes in pharmaceutical wastewater treatment systems [J]. Industrial Water Treatment, 2022, 42(4): 39-45 (in Chinese)
[23] Zheng J, Su C, Zhou J W, et al. Effects and mechanisms of ultraviolet, chlorination, and ozone disinfection on antibiotic resistance genes in secondary effluents of municipal wastewater treatment plants [J]. Chemical Engineering Journal, 2017, 317: 309-316
[24] Ju F, Beck K, Yin X L, et al. Wastewater treatment plant resistomes are shaped by bacterial composition, genetic exchange, and upregulated expression in the effluent microbiomes [J]. The ISME Journal, 2019, 13(2): 346-360
[25] Bao Y Y, Li F F, Chen L J, et al. Fate of antibiotics in engineered wastewater systems and receiving water environment: A case study on the coast of Hangzhou Bay, China [J]. The Science of the Total Environment, 2021, 769: 144642
[26] 颉亚玮. 杭州湾可吸附有机卤素污染溯源和源减排工艺研究[D]. 北京: 清华大学, 2017: 25-40
[27] 苏志国, 张衍, 代天娇, 等. 环境中抗生素抗性基因与Ⅰ型整合子的研究进展[J]. 微生物学通报, 2018, 45(10): 2217-2233
Su Z G, Zhang Y, Dai T J, et al. Antibiotic resistance genes and class Ⅰ integron in the environment: Research progress [J]. Microbiology China, 2018, 45(10): 2217-2233 (in Chinese)
[28] 李彦媚, 赵喜红, 徐泽智, 等. 新型细菌耐药元件——整合子系统[J]. 中国抗生素杂志, 2012, 37(1): 1-7
Li Y M, Zhao X H, Xu Z Z, et al. Novel antibiotic resistance mechanism-integron system [J]. Chinese Journal of Antibiotics, 2012, 37(1): 1-7 (in Chinese)
[29] Qian X, Gunturu S, Sun W, et al. Long-read sequencing revealed cooccurrence, host range, and potential mobility of antibiotic resistome in cow feces [J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(25): e2024464118
[30] Zhang H M, Hou M Y, Xu Y C, et al. Action and mechanism of the colistin resistance enzyme MCR-4 [J]. Communications Biology, 2019, 2: 36
[31] Wales A D, Davies R H. Co-selection of resistance to antibiotics, biocides and heavy metals, and its relevance to foodborne pathogens [J]. Antibiotics, 2015, 4(4): 567-604
[32] Che Y, Yang Y, Xu X Q, et al. Conjugative plasmids interact with insertion sequences to shape the horizontal transfer of antimicrobial resistance genes [J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(6): e2008731118
[33] Che Y, Xia Y, Liu L, et al. Mobile antibiotic resistome in wastewater treatment plants revealed by nanopore metagenomic sequencing [J]. Microbiome, 2019, 7(1): 44
[34] Lee J, Ju F, Maile-Moskowitz A, et al. Unraveling the riverine antibiotic resistome: The downstream fate of anthropogenic inputs [J]. Water Research, 2021, 197: 117050
[35] Su Z G, Wen D H. Characterization of antibiotic resistance across Earth’s microbial genomes [J]. The Science of the Total Environment, 2022, 816: 151613
[36] Henderson P J F, Maher C, Elbourne L D H, et al. Physiological functions of bacterial “multidrug” efflux pumps [J]. Chemical Reviews, 2021, 121(9): 5417-5478
[37] 张刚, 冯婕. 细菌固有耐药的研究进展[J]. 遗传, 2016, 38(10): 872-880
Zhang G, Feng J. The intrinsic resistance of bacteria [J]. Hereditas, 2016, 38(10): 872-880 (in Chinese)
[38] Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil [J]. Environment International, 2016, 92-93: 1-10
[39] Raza S, Shin H, Hur H G, et al. Higher abundance of core antimicrobial resistant genes in effluent from wastewater treatment plants [J]. Water Research, 2022, 208: 117882